-
农作物的精准识别有助于人们及时、准确地掌握农作物的种植结构及其时空变化信息,对区域内农作物的空间格局分布、产量的预测、农业资源的调查和灾害监测等问题具有重要意义[1]。传统多光谱遥感受传感器波段少、光谱分辨率低、作物光谱相似性等问题的影响,无法获得较高的作物类型识别精度;而新兴的高光谱技术通过获取连续地物的光谱信息,能在众多窄波段范围内对作物的细微差别进行探测,进而提高作物识别精度[2]。近年来国内外学者利用高光谱数据在作物识别与分类方面已开展了大量研究工作[3-6]。高光谱数据波段多、数据量大、信息冗余严重,因此在数据的应用与处理中如何选取有效的光谱数据形式和光谱特征变量是研究的重点。目前,该类研究对作物分类识别的精度不断提升,但这些方法主要基于传统统计方法结合主观判断选择波段,主观性较大,且数据处理操作较为繁琐,难以高效简便对作物进行精确分类识别。本研究以杭州地区常见的8种作物作为研究对象,基于实测的叶片光谱反射率数据,通过不同特征提取与分类方法对作物光谱分析,探寻识别不同作物的高效方法,从而为作物高光谱遥感解译和精准分类提供参考。
HTML
-
研究区浙江省杭州市(29°11′~30°33′N,118°21′~120°30′E),地处中国东南沿海、浙江省北部。研究区属亚热带季风气候,光温同步,雨热同季,日照和无霜期较长。研究区西部连山,东部近海,地势西高东低,地形地貌复杂多样。研究区具有丰富的水稻土,约占土壤总面积的14%,主要分布在平原地区,适宜于多种作物生长。研究区主要农作物有大豆Glycine max、番茄Lycopersicon esculentum、茄Solanum melongena、水稻Oryza sativa、茶Camellia sinensis、葡萄Vitis vinifera、玉米Zea mays、山核桃Carya cathayensis、番薯Ipomoea batatas、花生Arachis hypogaea、四季豆Phaseolus vulgaris等。
-
选取杭州市常见的大豆、玉米、茄、四季豆、花生、葡萄、番薯、水稻共8种农作物作为光谱测试对象。光谱测试部位为农作物叶片,叶片反射光谱采用美国ASD FieldSpec Pro FR地物光谱仪(光谱范围350~2 500 nm)进行测量。测定时间为2018年7月上旬,每天8:00-10:00,在天空晴朗无云,无风或微风,空气湿度小的情况下,户外采集农作物叶片后立即在室内用植被探头测量8种作物的叶片反射率。每种作物选择5片叶片,每片叶片选择3处测量点,以每一测量点连续测量10次的光谱平均值作为其光谱反射值,共获得150条光谱曲线,再对每10条光谱数据取其平均值作为一个样本数据,每种作物得到15个光谱样本数据。剔除有明显异常的波段数据,以剩余数据的平均值作为该样点的光谱反射率,再对光谱曲线进行平滑处理,消除光谱曲线上存在的噪声,最终获得8种作物反射光谱曲线数据。
-
采用随机森林方法与传统的高光谱识别与分类方法处理并分析作物高光谱数据,提取识别不同作物类型的高光谱特征,对作物进行精准识别与分类,并对结果分析与比较,这些传统方法包括一阶微分、二阶微分、倒数的对数等数学变换方法、去包络线法等。
-
① 简单数学变换法:运用光谱微分方法处理光谱曲线,能够部分消除大气效应、作物环境背景(阴影、土壤等)的影响,以反映作物本身的光谱特征[7];对数据进行倒数的对数可以减少因光照等变化引起的乘性因素对光谱数据的影响,使可见光区范围内光谱数据差异增大,从而更容易识别不同的作物[8]。采用一阶微分、二阶微分以及倒数的对数变换对原始光谱进行处理,观察分析光谱特征及其区分不同作物的能力。②去包络线法:包络线(envelope)是指每条光谱曲线的外凸包曲线,去包络线(continuum removal)是一种非线性光谱变换方法[9]。去包络线法对作物光谱曲线上反射率小、光谱曲线相近的可见光波段处理有效,能在很大程度上放大作物间光谱差异性,有利于作物识别分类[10]。本研究用MATLAB软件对原始光谱曲线进行去包络线处理,提取作物间光谱差异较大的波段,再使用欧氏距离法[11]对不同作物识别与分类。③随机森林法(random forest, RF)[12]:是一种基于分类与回归决策树(classification and regression tree, C&RT)的组合算法。随机森林算法对参与分类的变量没有限定,在处理高维数据分类时,更能体现随机森林的速度快、精度高、稳定性好的优势[13]。因此用随机森林法处理作物高光谱数据时,不用提前做光谱特征提取,在实施分类的同时,就可以对高光谱变量进行筛选优化[14],并分析判断特征波段的优劣。
1.1. 研究区概况
1.2. 数据采集与预处理
1.3. 技术路线
1.4. 研究方法
-
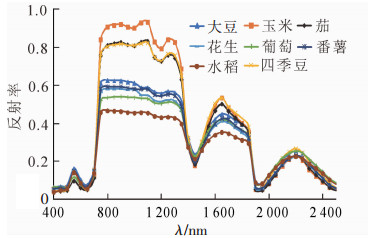
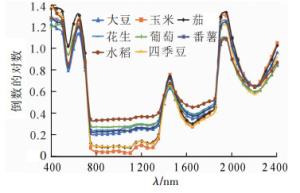
图 1为8种作物的平均光谱曲线,在可见光波段,绿峰波段反射率差异最大;在近红外波段,800~1 300 nm波段的光谱曲线差异最明显,反射率由高到低顺序为玉米、茄、四季豆、大豆、番薯、花生、葡萄、水稻,其中茄与玉米反射率较为接近,较难区分;在1 600~1 800 nm波段内不同作物的光谱曲线差异较为明显,除了葡萄与花生、茄与玉米的光谱差异较小外,剩余作物之间通过反射率差异可以区分。
-
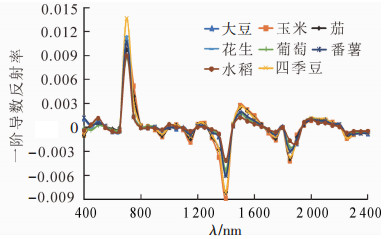
从图 2可观察到:8种作物的一阶微分光谱曲线的变化走向基本一致,在极大或极小值对应的波段,不同作物的一阶微分值差异较为明显, 其他波段内作物曲线相差不大。在685~770 nm波段内可以区分四季豆与其他作物,但不能明显区分出其余作物;在1 350~1 430 nm波段内不同作物一阶微分值有较大差异,能够区分出水稻、玉米与葡萄3种作物,其余作物无明显差别。
-
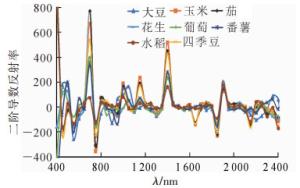
由图 3可知:8种作物二阶导数光谱曲线在650~790、1 300~1 500和1 820~1 940 nm等3个波段区间差异较大。表 1统计了8种作物在这3个波段内的极大值及其对应波段。即便在这些差异较大的波段内,部分作物的二阶导数光谱曲线十分接近,不利于区分不同作物;比如玉米、番薯、大豆、花生、葡萄在650~790 nm波段的光谱二阶导数值相近,大豆、花生、葡萄在1 300~1 500 nm波段的光谱二阶导数值相近,番薯、大豆、花生、葡萄在1 820~1 940 nm波段的光谱二阶导数值相近,这些二阶导数值相近的作物彼此难以区分。
作物类型 680~700 nm极大值对应波段 极大值×1 000 000 1 390~1 460 nm极大值对应波段 极大值×1 000 000 1 880~1 940 nm极大值对应波段 极大值×1 000 000 四季豆 693 1 101.0 1 401 431.4 1 888 416.4 茄 697 8 885.0 1 401 449.5 1 888 389.5 玉米 694 869.8 1 399 522.8 1 886 419.5 番薯 693 829.6 1 403 307.3 1 890 362.7 大豆 692 828.6 1 403 265.1 1 890 340.7 花生 691 788.7 1 403 284.2 1 890 318.9 葡萄 694 759.1 1 403 260.9 1 891 322.3 水稻 692 632.3 1 406 192.0 1 891 279.7 Table 1. Maximum value of second derivative of crop reflection spectrum and corresponding band
-
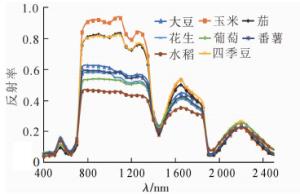
从图 4可知:与原始光谱曲线相比,波峰波谷发生了倒置,出现了“两峰一谷”,峰谷凸凹程度明显增加[15],原本相近的部分作物光谱曲线有所拉开,在800~1 300 nm波段内,水稻、玉米可以明显区分,但大豆、番薯和花生3种作物间的光谱差别过小,不易区分。
-
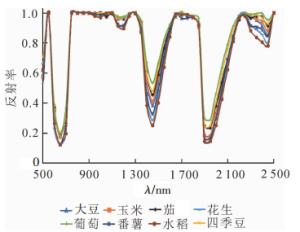
运用MATLAB软件,对作物反射光谱数据做去包络线处理。图 5可以看出:作物光谱在蓝谷,红谷,绿峰,1 170~1 190 nm,1 430~1 450 nm以及1 910~1 930 nm等波段上有较明显的差异。
利用这些差异较大的波段反射率,基于欧氏距离分类法,评价分析任意2种作物间的可分离性。以绿峰波段为例,任意2种作物间的欧氏距离结果见表 2。可以看出,绿峰波段,玉米、茄、花生等可以较为明显区分,其余作物较难区分。运用相同方法得出其他波段范围结果:在蓝谷波段,葡萄、茄、大豆、玉米、番薯、四季豆可以较明显区分,但水稻和花生较难以区分;红谷波段,茄、玉米、四季豆3者较难区分,其余作物较好区分;1 170~1 190 nm(水氧吸收波段)波段,葡萄、大豆、花生3种作物较难区分;1 430~1 450 nm波段,葡萄、大豆、四季豆3种作物较难区分;1 910~1 930 nm波段,葡萄、大豆两者较难区分。
作物类型 番薯 花生 大豆 葡萄 茄 水稻 四季豆 玉米 番薯 0.010 9 0.014 1 0.017 9 0.025 1 0.045 7 0.009 2 0.009 0 0.009 0 花生 0.014 1 0.015 8 0.016 3 0.039 9 0.060 5 0.017 7 0.018 0 0.018 0 大豆 0.017 9 0.016 3 0.008 1 0.043 9 0.064 6 0.020 3 0.020 9 0.020 9 葡萄 0.025 1 0.039 9 0.043 9 0.015 6 0.019 5 0.025 6 0.025 1 0.025 1 茄 0.045 7 0.060 5 0.064 6 0.019 5 0.007 2 0.042 1 0.041 5 0.041 5 水稻 0.009 2 0.017 7 0.020 3 0.025 6 0.042 1 0.016 7 0.016 8 0.016 8 四季豆 0.009 0 0.018 0 0.020 9 0.025 1 0.041 5 0.016 8 0.006 4 0.006 4 玉米 0.009 5 0.017 1 0.019 0 0.027 0 0.043 7 0.016 8 0.005 6 0.016 6 Table 2. Significant Euclidean distance table between any two crops
-
采用IBM SPSS modeler 18.0软件构建随机森林模型数据流,对农作物反射率数据处理。在软件中,使用C&RT算法构建随机森林的每棵树,决策树的棵数和候选分割属性集的大小设置均采用默认值,即构建500棵树,样本大小为百分百。作物在可见光波段(380~780 nm),近红外短波(780~1 100 nm),近红外长波(1 100~2 500 nm)的反射光谱特征与作物的生理生化特点有紧密联系。为了比较可见光、近红外波段的光谱对区分不同作物的效果,将作物光谱样本数据按3个不同的波段分成3组数据,与全波段(350~2 500 nm)数据构成4组不同的样本数据。样本数据一半用于随机森林法建模,提取区分不同作物的特征波段,一半用于随机森林法分类,验证所提取波段的重要性。为了减少数据处理量,将光谱反射率每10 nm取平均值用于随机森林法处理。依次将4组样本数据进行随机森林法处理,结果(表 3)表明:4组数据所建模型均都具有较高的分类准确性。随机森林法在建模的同时对参与分类的波段重要性进行了排序,表 4显示4组数据最重要的前10个波段,每个波段特征对分类精度的贡献程度不同,重要程度越高的波段对作物分类精度的影响越大。
输入变量波段/nm 输入的波段数 模型分类准确性/% 模型误分类率/% 380~780 40 82.4 17.6 780~1 100 32 74.8 25.2 1 100~2 500 140 81.5 18.5 350~2 500 215 87.4 12.6 Table 3. Random forest classification results for spectral data from different bands
380~780 nm 波段重要性 780~1 100 nm 波段重要性 1 100~2 500 nm 波段重要性 350~2 500 nm 波段重要性 770 2.37 800 2.20 1 200 1.59 550 1.04 710 2.09 780 1.68 1 110 1.51 2 490 0.93 400 1.82 1 010 1.57 1 150 1.04 370 0.91 380 1.77 1 050 1.21 1 510 0.90 770 0.87 740 1.71 840 1.15 1 230 0.84 560 0.84 670 1.71 790 1.06 1 190 0.81 380 0.83 550 1.59 850 1.01 2 430 0.75 540 0.83 730 1.49 830 0.98 1 140 0.69 530 0.83 390 1.32 810 0.94 1 310 0.66 570 0.74 540 1.15 1 000 0.92 1 930 0.62 350 0.74 Table 4. Sorting the importance of the bands involved in the classification
利用验证样本对所建随机森林法分类模型精度进行检验,结果(表 5)显示:4组样本的作物分类正确率均在84%及以上,精确度最高的样本组为350~2 500 nm全波段数据,分类精确率达99.17%。
波段/nm 正确 错误 精确度/% 380~780 118 2 98.33 780~1 100 101 19 84.17 1 100~2 500 115 5 95.83 350~2 500 119 1 99.17 Table 5. Analysis table of classification accuracy of different crops
为了检验所筛选的特征波段对区分不同作物的效果,依据350~2 500 nm波段样本数据随机森林分类输出的前10个重要波段,从验证样本数据中选出相应波段数据为变量,在模型参数设置不变的情况下进行随机森林分类,分类准确性达100%,在完全区分不同作物的同时,数据量减少达95.34%,在节省时间的同时保证了较高的准确度。前10个波段为550、2 490、370、770、560、380、540、530、570和350 nm波段。其中350和2 490 nm这2个波段在采集反射率时有噪声,虽然经过平滑方法消除了噪声,但不能完全排除噪声对识别区分作物的影响,因此在选择使用时要慎重。重要波段中的350、370和380 nm等3个波段属于蓝光吸收谷;530、540、550、560和570 nm等5个波段处于绿光反射峰;770 nm波段处于植被反射光谱陡坡;2 490 nm波段处于作物所含水分和二氧化碳的强吸收带。不同作物在这些波段的反射率差异均与作物的叶绿素含量、水分含量、叶片海绵组织及光合作用强烈相关[16]。由此可得出结论,随机森林法筛选出的特征波段不仅区分不同作物效果显著,而且能够反映不同作物生理与生化特性的差异,从生物本身特性的角度为高光谱区分不同作物提供了印证与依据。
2.1. 作物光谱平均反射率分析
2.2. 作物光谱一阶导数变换分析
2.3. 作物光谱二阶导数变换分析
2.4. 作物光谱倒数的对数变换分析
2.5. 作物光谱的包络线变换分析
2.6. 作物光谱的随机森林分析
-
本研究发现随机森林法对350~2 500 nm全波段反射率数据处理,不仅筛选出能够区分不同作物的特征波段,而且运用所选择的波段对作物进行分类识别的效果也是最优的。不仅如此,随机森林法对筛选出的波段进行了优劣排序,其中对分类贡献较大的波段集中在蓝光、绿峰、红光等波段,反映了作物生理生化特征差异。区分8种作物的特征波段主要有350~380 nm的蓝光波段、530~570 nm的绿峰波段、770 nm的植被陡坡波段、2 490 nm的水、二氧化碳的吸收波段。运用随机森林法能够克服作物光谱相似性较高、难分类等问题,快速高效确定区分不同作物的特征波段,且分类识别精度高。而用观察法分析不同作物的反射光谱及其一阶微分、二阶微分、倒数的对数,提取同时区分识别8种作物的波段难度较大;去包络线法突出了作物光谱在红谷、蓝谷、绿峰区域等特征波段的差异,但提取的特征波段只能区分部分作物,不能同时对8种作物分类。










 DownLoad:
DownLoad: