-
纤维素合成酶(cellulose synthase, CesA)是一种与膜结合的糖基转移酶,通过利用UDP-葡萄糖催化形成β-1, 4糖苷链,进一步合成纤维素[1]。纤维素合成酶被称之为“复合物的复合物”, 其家族有多个成员。在执行功能前,不同成员之间形成二聚体亚单元,6个亚单元之间形成1个独立单元,6个独立单元之间形成复杂的纤维素合成酶聚合体[2]。纤维素依赖于这种复合物的合成和加工,并储存在细胞壁中。因此,研究纤维素合成酶在植物中的功能,有助于认识植物细胞壁形成的机理以及富含纤维素的生物质能源的利用。拟南芥Arabidopsis thaliana中有10个CesA基因,水稻Oryza sativa中有10个CesA基因,玉米Zea mays中有20个CesA基因,大麦Triticum aestivum中有9个CesA基因[3-4]。对拟南芥的研究表明:AtCesA1,AtCesA3,AtCesA6,AtCesA10与初生壁合成相关,AtCesA4,AtCesA7,AtCesA8与次生壁合成相关,AtCesA2,AtCesA5,AtCesA9与AtCesA6在功能上有部分冗余[5]。另外,水稻、大麦、陆地棉Gossypium hirsutum中有关CesA4的基因研究表明:CesA4基因参与次生壁的形成。该基因的突变或过表达对纤维素的含量变化影响很大[6]。水稻中CesA9基因研究表明:OsCesA9与水稻次生壁的形成有关,CesA9的错义突变会导致植株矮化和不育[7]。这表明不同植物中的纤维素合成酶基因存在功能上的差异,且决定着植物中的纤维含量。毛竹Phyllostachys edulis是开发利用程度最高,经济规模最大的竹种,其纤维含量的高低更是影响着竹材的质地和竹笋的口感。目前,涉及毛竹纤维素合成酶的分子研究还比较少,相关报道中仅6个纤维素合成酶基因被克隆[8-9],而纤维素合成酶在毛竹幼竹生长过程中的功能仍不清楚。本研究利用生物信息学方法找数据库可信度高的纤维素合成酶家族成员16个,利用实时定量聚合酶链式反应(qRT-PCR)技术对其中8个纤维素合成酶基因进行时空表达模式分析。结合不同高度不同部位毛竹细胞壁超微结构观察,预测不同纤维素合成酶成员的功能,以期为毛竹幼竹生长发育机制的解析提供重要的分子依据。
-
实验所用材料均于2015年采自浙江省临安市板桥镇海拔50~200 m的向阳山坡上正常生长发育的野生毛竹林。采样条件:4月7日,气温5.0~10.0 ℃,相对湿度95%,采集0.1 m高(H1)的毛竹笋3株,0.3 m(H2)毛竹笋3株;4月15日,气温10.0~25.0 ℃,相对湿度30%,采集1.2 m(H3)高的毛竹笋3株;4月25日,气温15.0~28.0 ℃,相对湿度25%,采集6.0 m(H4)高的毛竹笋3株, 胸径40 cm;5月17日,气温20.0~30.0 ℃,相对湿度60%,采集14.0 m(H5)高的竹笋3株, 胸径40 cm。将毛竹笋地上部分按高度平均分成3个部分,取各部分的中间部位的材料作为实验样品,从上到下分别标记为顶部、中部和基部。地下部分标为根部。将采集的样品放液氮速冻,-80 ℃保存。另外一部分样品,放在体积分数为2.5%戊二醛固定液中进行固定,4 ℃保存。
-
取出保存在体积分数为2.5%的戊二醛固定液中的样品,经0.1 mol·L-1 pH 7.0的磷酸缓冲液冲洗,固定在体积分数为1.0%的锇酸溶液中。用不同体积分数梯度的乙醇对样品进行脱水处理,并用环氧化树脂包埋。样品在Leica EM UC7型超薄切片机中切片,获得70~90 nm的切片,经柠檬酸铅溶液、醋酸双氧铀和体积分数为50%饱和乙醇中各染色5~10 min,在Hitachi H-7650型透射电镜中观察。
-
在美国生物技术信息中心(NCBI)数据库中搜索毛竹纤维素合成酶基因的蛋白序列和DNA序列,并登陆BambooGDB(),下载毛竹基因组蛋白质序列。pfam网站上下载Cellulose-synt结构域的种子序列PF03552,利用HMMER2.3.2中hmmsearch的功能搜索毛竹基因组蛋白质序列库,凡是阈值E«1的均认为是其超家族成员[10]。将所得序列进行比对,去除纤维素合成酶类似蛋白家族基因序列、相同序列和相差几个内含子的基因序列。ProtScale(http://web.expasy.org/protscale/)用于蛋白的分子量和等电点分析,TMHMM()用于蛋白跨膜结构分析,Smart(http://smart.embl-heidelberg.de/)用于蛋白结构域分析。毛竹纤维素合成酶家族基因的蛋白序列经ClustalX2全序列比对后用MEGA6构建分子系统树。并在CellWallGenomics(https://cellwall.genomics.purdue.edu/intro/index.html)下载水稻和拟南芥的纤维素合成酶基因。将毛竹、拟南芥、水稻等3个物种的纤维素合成酶家族基因的蛋白序列经ClustalX2全序列比对后用MEGA6构建系统发育树[11]。
-
用TRIZOL法提取毛竹RNA[12],选择质量完备的RNA反转录成cDNA。cDNA经内参基因NTB(F:TCTTGTTTGACACCGAAGAGGAG,R: AATAGCTGTCCCTGGAGGAGTTT)和TIP41(F:AAAATCATTGTAGGCCATTGTCG,R: ACTAAATTAAGCCAGCGGGAGTG)检验后,-20 ℃保存[13]。选择合适的定量片段,设计定量引物(表 1)。以保存的cDNA为模板,扩增出的片段经胶回收,连接到T-Vector pMD19(Simple)(购自Takara公司),测序验证正确后,进行qRT-PCR实验,并用2-△△Ct的方法分析实验结果[14]。
表 1 毛竹纤维素合成酶基因定量信息
Table 1. Phyllostachys edulis cellulose synthase gene quantitative information
名称 全长/bp 定量片段/bp 起始/bp 结尾/bp 引物 测序结果 PeCesA1 3 525 156 43 198 F:GTCGTCACGATCCGCT 一致 R:ACGACAAACTGGGAAGGCA PeCesA2 3 354 299 439 737 F:AGGATGCTCACCTGGCACAT 一致 R:ACCCTCTCTTTCCACGCAAC PeCesA3 3 411 113 685 797 F:ATCCACCCGCTTCCTTATGC 一致 R:ATCCTCTCCTTCCACGCAAC PeCesA4 2 004 243 1 536 1 778 F:GTTCTGGGTCATCGGTGGT 一致 R:GACTCGTAGCCGCTGTTGA PeCesA5 3 249 173 2 785 2 957 F:TGGGTCATTGGAGGTGTCTC 一致 R:AGCGTGGTTGGAGGTATCAG PeCesA6 3 291 178 2 838 3 015 F:AGGTGTTTCTGCACATCTC 一致 R:AAAGTTCAACAATAGCAGTG PeCesA9 2 400 180 40 219 F:CGCAACGAGCTCGTGCTCATC 一致 R:GCGGCGCTCGTACTCGTAGCA PeCesA13 3 103 162 1 162 F:ATGTGGAGGGACAGGAACGACTCG 一致 R:CGGGATGTCATCAACCATCTGCCCGTTA -
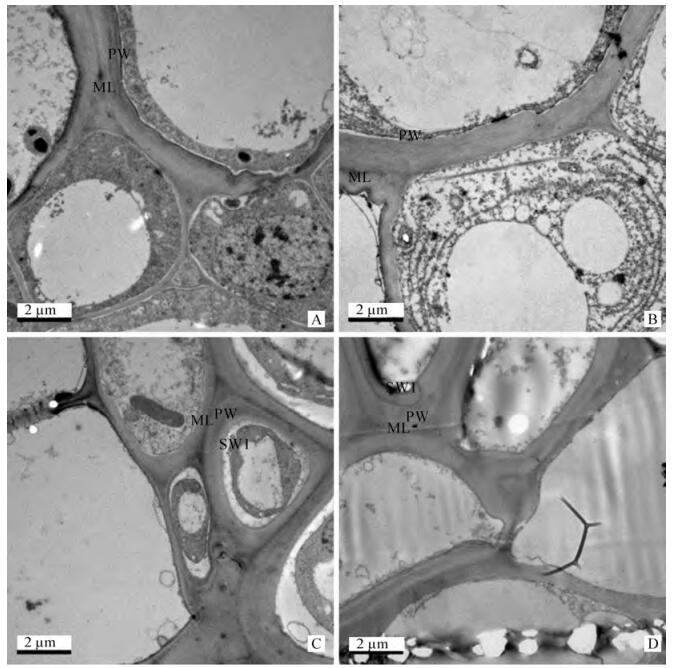
实验观察了5个高度毛竹的基部及H4时顶部、中部和根部的韧皮部细胞的超微结构。从细胞结构来看,在毛竹发育初期,细胞内含物较丰富,后期细胞内含物开始减少,逐渐变空(图 1,图 2)。这可能是早期细胞结构未发育成熟,仍具有分裂分化的能力。韧皮部的功能主要是运输有机营养物质,随着韧皮部结构的成熟,细胞变成与其功能相适应的稳定结构。从细胞壁结构来看,H1,H2基部皆有一层初生壁结构,H3基部次生壁开始出现,但不明显,H4和H5的基部的次生壁较明显,且H5比H4时的次生壁厚。这表明竹笋基部韧皮部的细胞壁结构层次随高度的增加逐渐增加。H4时,中部和基部有次生壁结构,顶部和根部未观察到次生壁形成(图 1,图 2)。这表明,次生壁先在成熟的基部产生。
-
利用HMMER 2.3.2中hmmsearch的搜索功能,得到48条靶标序列,NCBI中找到13条靶标序列。将所得61序列进行比对,去除纤维素合成酶类似蛋白家族基因序列、相同序列和相差几个内含子的基因序列后,共获得16条纤维素合成酶CesA基因。跨膜预测表明:纤维素合成酶一般有6~8个跨膜区,分子量在110 kD左右,等电点在7.0左右(表 2)。结构域分析表明:毛竹纤维素合成酶都含有cellulose_synt结构域,N端大多有锌(Zn)指结构(图 3)。用MEGA 6.0对毛竹纤维素合成酶家族基因蛋白质序列数据集进行验证,数据集的平均距离为0.568,适合构建NJ(neighbor joining)树,选择贝叶斯数最低的JTT(Jones-Taylor-Thornton)模型,步长值设为1 000。分子系统进化树表明:纤维素合成酶家族成员相似性高,遗传距离近。从拓扑分支来看,纤维素合成酶家族基因有3种聚类情况,PeCesA9和PeCesA12占有1个分支;PeCesA2,PeCesA8,PeCesA14,PeCesA16分别占有1个分支;其他成员聚在一簇(图 3)。这表明纤维素合成酶基因中,PeCesA2,PeCesA8,PeCesA14,PeCesA16可能独立行使功能,PeCesA9和PeCesA12和其他聚在一簇的成员可能存在功能上的冗余。
表 2 毛竹、拟南芥、水稻等纤维素合成酶家族成员分析
Table 2. Members of cellulose synthase family in Phyllostachys edulis, Oryza sativa and Arabidopsis thaliana
登录号 名称 氨基酸数 跨膜数 分子量大小/kD 等电点 PH01000357G0340 PeCesA1 1 174 7 131.0 6.72 PH01002002G0290 PeCesA2 1 117 6 124.5 7.46 PH010022C2G0080 PeCesA3 1 136 8 128.0 7.76 PH01000018G0C80 PeCesA4 667 7 74.0 5.82 PH0100C000G0020 PeCesA5 1 082 8 123.0 7.94 PH01000204G0C50 PeCesA6 1 096 8 123.0 7.61 PH01000693G0390 PeCesA7 488 1 55.0 9.20 PH01001427G0C90 PeCesA8 1 081 6 121.0 7.88 PH01000924G0590 PeCesA9 799 5 89.0 6.24 PH01000746G0570 PeCesA10 846 2 95.5 6.22 PH01001146G0100 PeCesA11 747 3 83.0 8.40 PH01000693G0340 PeCesA12 290 4 32.0 5.74 ACZ82299.1 PeCesA13 982 8 111.0 8.45 GU176305.1 PeCesA14 1 056 5 118.0 8.62 GU176303.1 PeCesA15 1 077 6 122.0 7.64 FJ799713 PeCesA16 1 065 6 119.0 8.51 AT4GC2410.1 AtCesA1 1 081 6 122.0 6.53 AT4G39350.1 AtCesA2 1 084 6 122.0 7.42 AT5G05170.1 AtCesA3 1 065 8 120.0 7.66 AT5G44030.1 AtCesA4 1 049 8 120.0 8.22 AT5G09870.1 AtCesA5 1 069 8 121.0 7.06 AT5G64740.1 AtCesA6 1 084 8 122.5 7.24 AT5G17420.1 AtCesAl 1 026 6 116.0 6.25 AT4G18780.1 AtCesA8 985 8 111.5 6.77 AT2G21770.1 AtCesA9 1 088 6 123.0 6.44 AT2G25540.1 AtCesA10 1 065 8 120.6 6.15 LOC_Os05g08370.1 OsCesA1 1 076 8 121.0 6.68 LOC_Os03g59340.1 OsCesA2 1 073 6 120.6 7.86 LOC_Os07g24190.1 OsCesA3 1 093 8 123.5 6.74 LOC_Os01g54620.1 OsCesA4 989 8 111.0 6.09 LOC_Os03g62090.1 OsCesA5 1 092 8 123.4 6.66 LOC_Os07g14850.1 OsCesA6 1 092 8 122.4 7.6 LOC_Os10g32980.1 OsCesAl 1 063 6 120.0 8.25 LOC_Os07g10770.1 OsCesA8 1 081 6 121.0 8.03 LOC_Os09g25490.1 OsCesA9 1 055 6 118.6 6.42 LOC_Os06g39970.1 OsCesA11 860 6 93.5 7.2 
图 3 毛竹纤维素合成酶家族基因分子系统进化树及结构域
Figure 3. Molecular phylogenetic tree and domains of cellulose synthase family members in Phyllostachys edulis
选择毛竹、水稻、拟南芥等3个物种的纤维素合成酶家族基因蛋白序列,用最大似然法(maximum likelihood method,ML)和LG+G模型构建系统发育树。系统发育树显示:这些纤维素合成酶被分为6个亚类(A亚类、B亚类、C亚类、D亚类、E亚类、F亚类)。其中,A亚类、B亚类、E亚类、F亚类包含3个物种的基因,C亚类仅有拟南芥纤维素合成酶基因,D亚类包含毛竹、水稻的纤维素合成酶基因,毛竹、水稻、拟南芥均属于被子植物门,毛竹、水稻属于单子叶植物纲,拟南芥属于双子叶植物纲。这表明CesA基因极有可能在单、双子叶植物分化前就已经出现分化,且在基因进化过程中,毛竹和水稻的纤维素合成酶的蛋白质序列同源性高(图 4)。
-
实验选择8个毛竹纤维素合成酶基因在5个高度20个部位进行时空表达分析。在H1时,PeCesA1,PeCesA2,PeCesA6在顶部表达最显著,PeCesA9在基部表达最显著,PeCesA4在根部表达最显著,在各个部位都表达的基因有PeCesA1,PeCesA2,PeCesA3,PeCesA6和PeCesA13。在H2时,所有基因在根部表达都最显著;除PeCesA4,其他基因在基部的表达都次显著,在所有部位都表达的基因有PeCesA3,PeCesA5,PeCesA6和PeCesA13。在H3时,所有的基因在基部的表达都最显著,根部的表达都次显著,在所有部位都表达的基因有PeCesA1,PeCesA2,PeCesA5,PeCesA6和PeCesA13。在H4时,中部表达最显著的基因有PeCesA1,PeCesA2,PeCesA5,基部表达最显著的基因有PeCesA4,PeCesA6,PeCesA9,PeCesA13,所有的基因根部的表达都次显著,各个部位都表达的基因有PeCesA1,PeCesA2,PeCesA3,PeCesA6和PeCesA13。在H5时,PeCesA4,PeCesA9在顶部的表达最显著,PeCesA1,PeCesA2,PeCesA3和PeCesA5在中部的表达最显著,PeCesA6在根部的表达最显著,PeCesA1,PeCesA2和PeCesA6在顶部的表达次显著,PeCesA3,PeCesA5和PeCesA4在中部表达次显著,PeCesA3,PeCesA5,PeCesA6和PeCesA9在各个部位都表达。从结果可以得出,PeCesA4在毛竹生长初期仅在根部和基部表达,随着高度的增加,中部和顶部的表达量升高,根部和基部的表达量降低。除H2时毛竹的基部PeCesA9表达量显著,PeCesA4无表达,其他情况下PeCesA9和PeCesA4的表达模式一致。PeCesA6的表达情况和PeCesA4相反,PeCesA6在毛竹生长初期在顶部表达最显著,随着高度的增加,慢慢在基部和根部的表达量增加,顶部的表达降低。另外,PeCesA1和PeCesA2的表达模式则基本一致,2个基因在各个时期各个部位都有表达,H1时在顶部表达显著,H4时在中部表达显著,H3和H5时在基部表达显著,H2时在根部表达显著。PeCesA3和PeCesA5的表达模式基本一致,2个基因在各个时期各个部位都有表达,随着高度的增加,表达显著部位从根部变成中部和基部。H4之前,PeCesA13在生长初期表达最显著部位是根部,后在中部基部表达最显著,到H5时,表达情况又恢复到初始情况。在毛竹生长发育过程中,根部和基部表达的纤维素合成酶基因多于顶部和中部。这表明毛竹纤维素合成酶家族成员之间的功能上存在不同的分工,而且竹杆的纤维素合成很可能从基部和根部启动的(图 5)。
-
毛竹笋刚出土时,每天仅长高几厘米,以后逐渐加快,每天长高几十厘米,高峰期一昼夜可长高1.0 m以上。这种独特的生长特性,使它在幼笋出土长成幼竹的短短几个月内即完成秆形生长。为探究毛竹纤维素合成酶基因在毛竹幼竹生长过程的表达模式和功能,本研究选取5个高度的毛竹作为研究对象。0.1 m和0.3 m时为毛竹生长初期,1.3 m为毛竹上升期,6.0 m左右为毛竹盛期,14.0 m时毛竹开始抽枝展叶,为末期[15-17]。细胞壁按形成先后,分为初生壁和次生壁[18]。毛竹韧皮部细胞超微结构观察表明,一般生长期的毛竹基部韧皮部细胞可观察到次生壁出现,爆发式生长期的毛竹中部和基部韧皮部细胞有次生壁结构,顶部和根部韧皮部细胞未观察到次生壁结构,成竹期毛竹基部韧皮部细胞的次生壁比爆发式生长期厚。这与刘波等[19]研究发现不同类型的细胞出现壁层的时间和层数不同,纤维、基本薄壁组织细胞和导管细胞壁层数在发育过程中不断增加有一定的吻合。研究表明:纤维素是构成毛竹细胞壁的主要成分之一,尤其是在幼嫩的毛竹中,纤维素含量高达75%[20]。在毛竹幼龄期,纤维素含量随着高度和时间的增加而升高,成竹后随着年龄的增加而降低的趋势[21-23]。而在毛竹幼竹生长发育中次生壁形成的部位,最显著表达的基因有PeCesA1,PeCesA2,PeCesA3,PeCesA4,PeCesA5,PeCesA6,PeCesA9和PeCesA13,在仅有初生壁结构的部位最显著表达的基因有PeCesA6,PeCesA9和PeCesA13。因此推测PeCesA1,PeCesA2,PeCesA3,PeCesA4和PeCesA5基因主要参与毛竹次生壁的合成,PeCesA6,PeCesA9和PeCesA13对初生壁和次生壁的形成都起到一定作用。另外,在毛竹生长发育过程中,根部和基部表达的纤维素合成酶基因多于顶部和中部,尤其是基部的纤维素合成酶基因表达数最多且表达量最显著。针对这一现象的解释,可能是竹秆的发育、成熟和老化从基部首先启动,中部和顶部要顺次晚一些[16]。
系统进化分析表明:毛竹与水稻的亲缘关系较近,AtCesA4,PeCesA7,OsCesA7和AtCesA8,PeCesA4,OsCesA4在功能上可能保守。有研究表明:AtCesA4和AtCesA8与次生壁合成相关[5],OsCesA4参与次生壁的合成[6]。本研究中预测PeCesA4功能也主要参与毛竹次生壁的形成。AtCesA9与AtCesA6在功能上有部分冗余,OsCesA9基因与水稻次生壁的形成有关[7]。本研究中预测PeCesA6和PeCesA9功能都对初生壁和次生壁的形成有重要作用,也是功能冗余的体现。
本研究对8个纤维素合成酶基因表达模式归纳发现:PeCesA1和PeCesA2的表达模式相同;PeCesA3和PeCesA5的表达模式相同;PeCesA4和PeCesA6表达模式相反;除生长初期毛竹的基部PeCesA9表达量显著,PeCesA4无表达,其他情况下PeCesA9和PeCesA4的表达模式一致。猜测,在发挥功能时,PeCESA1和PeCESA2蛋白可能会形成二聚体,PeCESA3和PeCESA5蛋白可能会形成二聚体[24-25]。PeCesA4和PeCesA9蛋白根据功能选择是否形成二聚体,PeCESA4和PeCESA6蛋白参与的过程可能不同。本研究实验证据不足,这些纤维素合成酶以怎样的聚合形式执行怎样的过程以及发挥何种功能仍不清楚。
Temporo-spatial expressions and prediction of cellulose synthase gene functions with growth of Phyllostachys edulis
-
摘要: 毛竹Phyllostachys edulis是中国主要的经济竹种,纤维素合成对于竹材的形成具有重要意义。纤维素主要由纤维素合成酶(CesA)合成,并储存在植物的初生壁和次生壁中。通过生物信息学方法、透射电镜观察以及荧光定量聚合酶链式反应(qRT-PCR)技术研究了毛竹纤维素合成酶的表达和功能。共获得16个毛竹纤维素合成酶家族基因。结构域分析表明:毛竹纤维素合成酶都含有cellulose_synt结构域,N端大多有锌(Zn)指结构。毛竹韧皮部细胞超微结构观察发现:次生壁随着高的增加而加厚,次生壁的初始形成在上升期(1.2 m)。定量分析表明:8个基因(PeCesA1,PeCesA2,PeCesA3,PeCesA4,PeCesA5,PeCesA6,PeCesA9和PeCesA13)在次生壁形成的部位表达最显著,3个基因(PeCesA6,PeCesA9和PeCesA13)在初生壁结构部位表达最显著。结合前人对相同时期毛竹的纤维素含量研究,推测在毛竹幼竹生长过程中PeCesA1,PeCesA2,PeCesA3,PeCesA4和PeCesA5基因主要参与毛竹次生壁的合成,PeCesA6,PeCesA9和PeCesA13对初生壁和次生壁的形成都起到一定作用。Abstract: Phyllostachys edulis, a major commercial bamboo species in China, utilizes cellulose synthesis, which mainly synthesizes cellulose deposited in the primary and secondary cell walls and is key in bamboo growth and development. This study was conducted to determine the structure and function of cellulose synthase as it promotes bamboo growth and development. Expression and function of cellulose synthase were researched with a bioinformatics analysis, transmission electron microscope (TEM), and quantitative real-time polymerase chain reaction (qRT-PCR). Results indicated that 16 members of the cellulose synthase family were predicted, and the proteins contained a cellulose-synt motif and a zinc finger motif on the N terminus using domain prediction. Cell phloem ultrastructures of Ph. edulis showed that the secondary cell wall was thicker with an increasing height of bamboo, and first appeared in the basal part (first 1.2 m). Eight cellulose synthase genes (PeCesA1, PeCesA2, PeCesA3, PeCesA4, PeCesA5, PeCesA6, PeCesA9, and PeCesA13) had a high expression on parts of the secondary cell wall; whereas, three cellulose synthase genes (PeCesA6, PeCesA9, and PeCesA13) had a high expression on parts of the primary cell wall. Based on previous studies of bamboo cellulose content for the same growth stages, for young bamboo, PeCesA1, PeCesA2, PeCesA3, PeCesA4, and PeCesA5 were the main genes responsible for the secondary cell wall; and PeCesA6, PeCesA9, and PeCesA13 were likely involved in building both primary and secondary cell walls.
-
Key words:
- botany /
- Phyllostachys edulis /
- cellulose synthase /
- phloem /
- ultrastructure /
- primary cell wall /
- secondary cell wall
-
表 1 毛竹纤维素合成酶基因定量信息
Table 1. Phyllostachys edulis cellulose synthase gene quantitative information
名称 全长/bp 定量片段/bp 起始/bp 结尾/bp 引物 测序结果 PeCesA1 3 525 156 43 198 F:GTCGTCACGATCCGCT 一致 R:ACGACAAACTGGGAAGGCA PeCesA2 3 354 299 439 737 F:AGGATGCTCACCTGGCACAT 一致 R:ACCCTCTCTTTCCACGCAAC PeCesA3 3 411 113 685 797 F:ATCCACCCGCTTCCTTATGC 一致 R:ATCCTCTCCTTCCACGCAAC PeCesA4 2 004 243 1 536 1 778 F:GTTCTGGGTCATCGGTGGT 一致 R:GACTCGTAGCCGCTGTTGA PeCesA5 3 249 173 2 785 2 957 F:TGGGTCATTGGAGGTGTCTC 一致 R:AGCGTGGTTGGAGGTATCAG PeCesA6 3 291 178 2 838 3 015 F:AGGTGTTTCTGCACATCTC 一致 R:AAAGTTCAACAATAGCAGTG PeCesA9 2 400 180 40 219 F:CGCAACGAGCTCGTGCTCATC 一致 R:GCGGCGCTCGTACTCGTAGCA PeCesA13 3 103 162 1 162 F:ATGTGGAGGGACAGGAACGACTCG 一致 R:CGGGATGTCATCAACCATCTGCCCGTTA 表 2 毛竹、拟南芥、水稻等纤维素合成酶家族成员分析
Table 2. Members of cellulose synthase family in Phyllostachys edulis, Oryza sativa and Arabidopsis thaliana
登录号 名称 氨基酸数 跨膜数 分子量大小/kD 等电点 PH01000357G0340 PeCesA1 1 174 7 131.0 6.72 PH01002002G0290 PeCesA2 1 117 6 124.5 7.46 PH010022C2G0080 PeCesA3 1 136 8 128.0 7.76 PH01000018G0C80 PeCesA4 667 7 74.0 5.82 PH0100C000G0020 PeCesA5 1 082 8 123.0 7.94 PH01000204G0C50 PeCesA6 1 096 8 123.0 7.61 PH01000693G0390 PeCesA7 488 1 55.0 9.20 PH01001427G0C90 PeCesA8 1 081 6 121.0 7.88 PH01000924G0590 PeCesA9 799 5 89.0 6.24 PH01000746G0570 PeCesA10 846 2 95.5 6.22 PH01001146G0100 PeCesA11 747 3 83.0 8.40 PH01000693G0340 PeCesA12 290 4 32.0 5.74 ACZ82299.1 PeCesA13 982 8 111.0 8.45 GU176305.1 PeCesA14 1 056 5 118.0 8.62 GU176303.1 PeCesA15 1 077 6 122.0 7.64 FJ799713 PeCesA16 1 065 6 119.0 8.51 AT4GC2410.1 AtCesA1 1 081 6 122.0 6.53 AT4G39350.1 AtCesA2 1 084 6 122.0 7.42 AT5G05170.1 AtCesA3 1 065 8 120.0 7.66 AT5G44030.1 AtCesA4 1 049 8 120.0 8.22 AT5G09870.1 AtCesA5 1 069 8 121.0 7.06 AT5G64740.1 AtCesA6 1 084 8 122.5 7.24 AT5G17420.1 AtCesAl 1 026 6 116.0 6.25 AT4G18780.1 AtCesA8 985 8 111.5 6.77 AT2G21770.1 AtCesA9 1 088 6 123.0 6.44 AT2G25540.1 AtCesA10 1 065 8 120.6 6.15 LOC_Os05g08370.1 OsCesA1 1 076 8 121.0 6.68 LOC_Os03g59340.1 OsCesA2 1 073 6 120.6 7.86 LOC_Os07g24190.1 OsCesA3 1 093 8 123.5 6.74 LOC_Os01g54620.1 OsCesA4 989 8 111.0 6.09 LOC_Os03g62090.1 OsCesA5 1 092 8 123.4 6.66 LOC_Os07g14850.1 OsCesA6 1 092 8 122.4 7.6 LOC_Os10g32980.1 OsCesAl 1 063 6 120.0 8.25 LOC_Os07g10770.1 OsCesA8 1 081 6 121.0 8.03 LOC_Os09g25490.1 OsCesA9 1 055 6 118.6 6.42 LOC_Os06g39970.1 OsCesA11 860 6 93.5 7.2 -
[1] FESTUCCI-BUSELLI R A, OTONI W C, JOSHI C P. Structure, organization, and functions of cellulose synthase complexes in higher plants[J]. Braz J Plant Physiol, 2007, 19(1): 1-13. [2] MUTWIL M, DEBOLT S, PERSSON S. Cellulose synthesis: a complex complex[J]. Curr Opin Plant Biol, 2008, 11(3): 252-257. [3] ENDLER A, PERSSON S. Cellulose synthases and synthesis in Arabidopsis[J]. Mol Plant, 2011, 4(2): 199-211. [4] 公维丽, 王禄山, 张怀强.植物细胞壁多糖合成酶系及真菌降解酶系[J].生物技术通报, 2015, 31(4): 149-165. GONG Weili, WANG Lushan, ZHANG Huaiqiang. Diverse synthetases and fungi degradation enzymes for the polysaccharides of plant cell walls[J]. Biotechnol Bull, 2015, 31(4): 149-165. [5] CARROLL A, MANSOORI N, LI Shundai, et al. Complexes with mixed primary and secondary cellulose synthases are functional in Arabidopsis plants[J]. Plant Physiol, 2012, 160(2): 726-737. [6] YAN Changjie, YAN Song, ZENG Xiuhong, et al. Fine mapping and isolation of Bc7 (t), allelic to OsCesA4[J]. J Genet Genom, 2007, 34(11): 1019-1027. [7] WANG Daofeng, YUAN Shoujiang, YIN Liang, et al. A missense mutation in the transmembrane domain of CESA9 affects cell wall biosynthesis and plant growth in rice[J]. Plant Sci, 2012, 196(11): 117-124. [8] 张智俊, 杨洋, 何沙娥, 等.毛竹纤维素合成酶基因PeCesA的克隆及组织表达谱分析[J].园艺学报, 2010, 37(9): 1485-1492. ZHANG Zhijun, YANG Yang, HE Sha'e, et al. Cloning and expression characterization of the cellulose synthase gene (PeCesA) from moso bamboo (Phyllostachys edulis) shoot[J]. Acta Hortic Sin, 2010, 37(9): 1485-1492. [9] 袁丽钗. 毛竹纤维素生物合成相关基因研究[D]. 北京: 中国林业科学研究院, 2009. YUAN Lichai. Study on the Gene Involved in the Cellulose Biosynthesis of Moso Bamboo(Phyllostachys edulis)[D]. Beijing: Chinese Academy of Forestry, 2009. [10] FINN R D, CLEMENTS J, EDDY S R. HMMER web server: interactive sequence similarity searching[J]. Nucl Acid Res, 2011, 39(8): 29-37. [11] TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. [12] SIMMS D, CIZDZIEL P E, CHOMCZYNSKI P. TRIzol: a new reagent for optimal single-step isolation of RNA[J]. Focus, 1993, 15(4): 532-535. [13] FAN Chunjie, MA Jinmin, GUO Qirong, et al. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis)[J]. PLoS One, 2013, 8(2): e56573. doi: 10.1371/journal.pone.0056573. [14] SCHEFE J H, LEHMANN K E, BUSCHMANN I R, et al. Quantitative real-time RT-PCR data analysis: current concepts and the novel "gene expression's C T difference" formula[J]. J Mol Med, 2006, 84(11): 901-910. [15] PENG Zhenhua, ZHANG Chunling, ZHANG Ying, et al. Transcriptome sequencing and analysis of the fast growing shoots of moso bamboo (Phyllostachys edulis)[J]. PLoS One, 2013, 8(11): e78944. doi: 10.1371/journal.pone.0078944. [16] 崔凯, 何彩云, 张建国, 等.毛竹茎秆组织速生的时空发育特征[J].林业科学研究, 2012, 25(4): 425-431. CUI Kai, HE Caiyun, ZHANG Jianguo, et al. Characteristics of temporal and spatial tissue development during rapidly growing stage of moso bamboo culms[J]. For Res, 2012, 25(4): 425-431. [17] 方伟, 桂仁意, 林新春, 等.中国经济竹类[M].北京:科学出版社, 2015: 62. [18] 吴庆余.基础生命科学[M].北京:高等教育出版社, 2002: 62-68. [19] 刘波. 毛竹发育过程中细胞壁形成的研究[D]. 北京: 中国林业科学研究院, 2008. LIU Bo. Formation of Cell Wall in Developmental Culms of Phyllostachys pubescens[D]. Beijing: Chinese Academy of Forestry, 2008. [20] 张世源.竹纤维及其产品加工技术[M].北京:中国纺织出版社, 2008. [21] 胡铮瑢. 6月和12月竹龄毛竹竹竿半纤维素的分离及热稳定性研究[D]. 南昌: 南昌大学, 2011. HU Zhengrong. Study on Separation and Thermal Stability of Hemicellulose from Bamboo Stems Aged Six Months and Twelve Months[D]. Nanchang: Nanchang University, 2011. [22] 周斌雄. 基于近红外及拉曼光谱技术的毛竹化学成分和细胞结构研究[D]. 杭州: 浙江大学, 2015. ZHOU Binxiong. Revealing the Cell Structure and Chemical Formation of Bamboo with Near-infrared and Confocal Raman Microscopy[D]. Hangzhou: Zhejiang University, 2015. [23] 李权, 林金国, 刘主凰, 等.福建主要竹材纤维化学性质的差异分析[J].竹子研究汇刊, 2014, 33(2): 5-10. LI Quan, LIN Jinguo, LIU Zhuhuang, et al. The difference in fiber properties of major bamboo species in Fujian Province[J]. J Bam Res, 2014, 33(2): 5-10. [24] KUMAR M, TURNER S. Plant cellulose synthesis: CESA proteins crossing kingdoms[J]. Phytochemistry, 2015, 112(1): 91-99. [25] SETHAPHONG L, HAIGLER C H, KUBICKI J D, et al. Tertiary model of a plant cellulose synthase[J]. Proc Nat Acad Sci, 2013, 110(18): 7512-7517. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2017.04.001







 下载:
下载: