-
玉米Zea mays是主要的食品、饲料和工业原料作物,在粮食安全中发挥着至关重要的作用。容重作为玉米商品质量的重要指标,已成为国际贸易中质量分级的重要因素[1]。容重由单位容积内的粒数和质量决定,在生长发育过程中,受到多种因素的影响,包括玉米整株的外形、吐丝期、抽雄期、灌浆期等[1]。前人研究发现:籽粒灌浆率是影响作物籽粒容重的关键因素[2]。授粉 2~3周后,籽粒的灌浆速度较快,内部物质在此期间迅速积累[3]。由于玉米胚乳质量占籽粒质量的85%,玉米籽粒的饱满度主要受胚乳的影响[3]。胚乳细胞的增殖和发育决定了籽粒的质量和品质,玉米胚乳的发育包括籽粒库容建成和籽粒库充实[4]。籽粒库容的建成主要是胚乳细胞的增殖和生长扩张,以及胚乳细胞中淀粉颗粒的发育;籽粒库的充实主要通过淀粉的持续合成和积累,以及胚乳中淀粉颗粒的逐渐膨胀[5]。胚乳淀粉质量分数为70%~75%[5],由此可知,玉米中淀粉质量分数与容重紧密相关,容重可以反映籽粒的品质和成熟度[6]。以往研究对玉米品质的研究主要集中在营养品质和加工品质方面,对容重的研究较少。
籽粒容重与其他品质性状间有着密不可分的联系。在玉米中,吴春胜等[7]发现籽粒容重与蛋白质、脂肪含量负相关;张丽等[8]发现容重与蛋白质、淀粉含量正相关;张静等[9]发现容重与蛋白质含量负相关,与脂肪、淀粉含量正相关;而DORSEY-REDDING等[10]则发现容重与淀粉含量负相关。综上所述,玉米容重与其他品质性状之间的相关性存在争议。
前期通过数量性状基因座(QTL)或全基因组关联 (GWAS) 分析发现容重在小麦Triticum aestivum和玉米中具有高度遗传性,并且筛选到一些关键 QTLs、单核苷酸多态性位点(SNPs)或基因[11−16]。在玉米中,ZHANG等[11]对 B73 × Mo17构建的IBMSyn10 DH群体对容重性状进行关联分析,筛选到17个候选基因,其中包括 ATHB-4 (Zm00001d044081)。DING等[17]利用 Chang 7-2 × Zheng 58 构建的 225 个自交系群体通过混合模型(MLM)进行 QTL 分析,鉴定到 5 个与容重相关的 QTLs。LIU等[18]利用 10 个重组自交系群体进行 QTL 分析,在玉米中鉴定出 70 个 QTLs。由于容重是一个复杂的数量性状,目前关于控制容重的关键基因鉴定进展缓慢。
本研究使用 517份玉米种质对容重与粗淀粉、粗蛋白、粗脂肪、直链淀粉进行相关性分析,并选取极端种质进行转录组测序(RNA-Seq) 分析,挖掘控制容重的关键基因,为容重调控机制研究和高容重新品种选育提供候选基因和优异种质。
-
以 517 份玉米种质资源为材料,其中包括 368 份浙江地方玉米种质以及 149 份国内外骨干自交系。对玉米种质进行自交授粉 4 代。2022年4月种植于浙江省东阳市(29.28′N,120.33′E)、11月种植于海南省乐东县(18.52′N,108.93′E)和7月种植于浙江省杭州市临安区(30.23′N,119.72′E)。玉米种质在每个地点种植设3个随机区组重复。每个材料每行种植 20 株,选取9 个授粉良好的果穗进行测定。
B73作为玉米重要的骨干自交系,被广泛用于品种选育及各种基础研究的参考品种,因此以B73作为参照,以便监测和标准化不同批次样品的品质性状数据。
-
玉米果穗脱粒后去除杂质,自然干燥后含水量降至14%以下。用 GHCS-1000AP KTW 测定仪(浙江托普云农科技股份有限公司)按照 SAC 方法(GB 1353—2009《玉米》)检测容重。从 9 个果穗的混合籽粒中随机采集约50 g种子,用磨样机粉碎成粉末,进行其他品质性状测定。
-
取 10 g样本,用电热鼓风干燥器(DHG9030A,浙江托普云农科技股份有限公司)通过高温烘干法测定含水量。
-
含氮量按照改进的凯氏定氮法[19],使用全自动凯氏定氮仪(K9860,海能仪器股份有限公司)进行测定。取约 0.100 0 g的样本,籽粒中粗蛋白是检测含氮量的 6.25倍。
-
玉米籽粒粗脂肪的测定采用改良索氏抽提法[20],参考GB 2906—1982《粮油作物种子粗脂肪测定方法》,用索氏脂肪抽提仪(SOX606,海能仪器股份有限公司)提取2.000 0 g籽粒中的粗脂肪,然后用烘箱(XMTD-8222,上海精宏实验设备有限公司)去除脂肪抽提剂石油醚。
-
籽粒粗淀粉的测定参考ISO 10520: 1997《本地淀粉 淀粉含量的测定 旋光法》和GB 5006—1985《谷物籽粒粗淀粉测定法》。将 2.5000 g样本通过氯化钙-乙酸溶液水解,然后使用硫酸锌和亚铁氰化钾溶液进行提取,使用全自动旋光仪(P850,海能仪器股份有限公司)进行测定。
-
直链淀粉的测定参考GB 7648—1987《水稻、玉米和谷子籽粒直链淀粉测定法》的改进方法[21]。用 Synergy H1 多模式微孔板酶标仪(BioTek 仪器公司)测定直链淀粉-碘复合物在 720 nm 波长处的吸光度。
-
支链淀粉质量分数的计算方法是从粗淀粉中减去直链淀粉质量分数。每个样品测定2个重复,如果2次重复偏差超过5%,重复测定1次。直支比为直链淀粉质量分数与支链淀粉的比值。
-
授粉20~25 d是玉米种子发育和灌浆的重要阶段[22−23],因此RNA提取自授粉20 d的籽粒。2023年8月,将517份玉米种质种植于浙江省杭州市临安区,种植区域与2022年相同,进行3个随机区组重复,每个材料每行种植 20 株。 根据2022年品质分析结果,从具有极端表型的种质中分离籽粒,筛选4个容重较高、粗淀粉质量分数较高、粗蛋白质量分数较低、粗脂肪质量分数较低的种质(梅玉米、F1白轴、 CML324、975-12),以及 4 个容重较低、粗淀粉质量分数较低、粗蛋白质量分数较高、粗脂肪质量分数较高的种质(浙单14、M270、孝丰白马玉米、金辉黑糯玉米),上述8个极端种质的生育期均为101 d。将9个果穗的籽粒混合作为1个重复。提取的RNA送至北京诺禾致源科技股份有限公司进行RNA-Seq测序和分析。
-
授粉 20 d后,采集籽粒并保存在−80 ℃冰箱。使用 TransZol Up Plus RNA试剂盒(北京全式金生物科技股份有限公司)提取总RNA。使用 Hifair III 1st Strand cDNA Synthesis SuperMix 逆转录试剂盒(上海翌圣生物技术有限公司)将 RNA 反转录为 cDNA,使用稀释的 cDNA 进行实时荧光定量PCR (RT-qPCR)鉴定。以玉米管家基因ZmGAPDH作为内参,对样本进行RT-qPCR鉴定。引物组合:Zm00001d043468,使用引物1F 和1R;Zm00001d028709,使用引物2F和2R;Zm00001d021653,使用引物3F和3R;Zm00001d004438,使用引物 4F 和 4R;Zm00001d043511,使用引物 5F 和 5R; Zm00001d041775 使用引物 6F 和 6R;Zm00001d032224 使用引物 7F 和 7R;Zm00001d035156 使用引物 8F 和 8R;ZmGAPDH 使用引物 9F 和 9R,检测各基因表达。每个样品包含3个生物学重复,3个玉米的籽粒作为1个生物学重复。
-
利用Excel和SPSS 19.0进行数据分析,计算最大值、最小值、平均数、标准差、变异系数,绘制群体频率分布图,对玉米籽粒品质性状数据进行联合方差分析和相关性分析等。
-
从表1可见:玉米籽粒容重最大值和最小值均在东阳,分别为 904.00和 254.55 g·L−1。东阳容重的变异系数最大,临安的变异系数最小。经过最佳线性无偏预测法(BLUP)分析后,容重为431.41~798.55 g·L−1,变异系数降至 8.47%。粗蛋白质量分数最大值在乐东,最小值在东阳。东阳粗蛋白质量分数的变异系数最大,临安的变异系数最小。经 BLUP 分析,粗蛋白质量分数为6.17%~17.89%,变异系数降至 10.09%。粗脂肪质量分数最大值和最小值均在东阳,分别为 14.57%和 2.01%。东阳的粗脂肪质量分数变异系数最大,而临安最小。经 BLUP 分析,粗脂肪质量分数为 2.64%~12.99%,变异系数降至 29.01%。粗淀粉质量分数最大值在乐东,最小值在东阳。东阳的变异系数最大,临安的变异系数最小。经 BLUP 分析,粗淀粉质量分数为 12.45%~71.25%,变异系数降至 11.04%。直链淀粉质量分数最大值和最小值均在东阳,分别为 36.44% 和 2.66%。东阳的直链淀粉质量分数变异系数最大,而临安最小。经 BLUP 分析,直链淀粉质量分数在 5.70%~32.68%,变异系数降至 26.44%。支链淀粉质量分数最大值和最小值均在乐东,分别为65.94%和18.66%。经 BLUP 分析,支链淀粉质量分数为 16.65%~61.79%,变异系数降至 16.65%。直支比最大值在东阳,最小值在乐东。东阳的变异系数最大,而乐东的变异系数最小。经 BLUP 分析,直支比为 0.16~2.30,变异系数降至 33.08%。
表 1 玉米籽粒容重和其他品质性状统计及分析
Table 1. Descriptive statistics for maize test weight and other quality traits
项目 地点 容重/(g·L−1) 质量分数/% 直支比 粗蛋白 粗脂肪 粗淀粉 直链淀粉 支链淀粉 均值±标准差 东阳 606.84±101.03 12.48±2.08 4.98±2.00 62.73±9.64 21.87±8.94 40.83±10.64 0.60±0.32 乐东 692.46±69.45 13.98±2.08 5.01±1.70 64.23±6.00 21.89±6.77 42.35±8.30 0.55±0.21 临安 712.90±52.82 12.77±1.35 5.03±1.17 64.88±4.25 21.06±6.85 43.75±8.06 0.52±0.21 BLUP 664.42±56.29 13.17±1.33 5.04±1.46 63.21±6.98 22.10±5.84 41.46±6.90 0.58±0.19 数值范围 东阳 254.55~904.00 7.66~20.30 2.01~14.57 19.95~72.46 2.66~36.44 4.98~65.13 0.07~3.00 乐东 366.67~840.00 9.05~20.70 2.46~13.61 34.36~77.08 3.20~34.12 18.66~65.94 0.06~1.10 临安 431.82~871.43 9.72~17.91 3.13~12.00 36.76~72.17 4.36~31.50 20.70~64.65 0.07~1.17 BLUP 431.41~798.55 6.17~17.89 2.64~12.99 12.45~71.25 5.70~32.68 16.65~61.79 0.16~2.30 变异系数/% 东阳 16.65 16.66 40.13 15.37 40.88 26.07 53.39 乐东 10.03 14.85 33.83 9.34 30.95 19.59 37.44 临安 7.41 10.58 23.29 6.55 32.50 18.41 40.06 BLUP 8.47 10.09 29.01 11.04 26.44 16.65 33.08 -
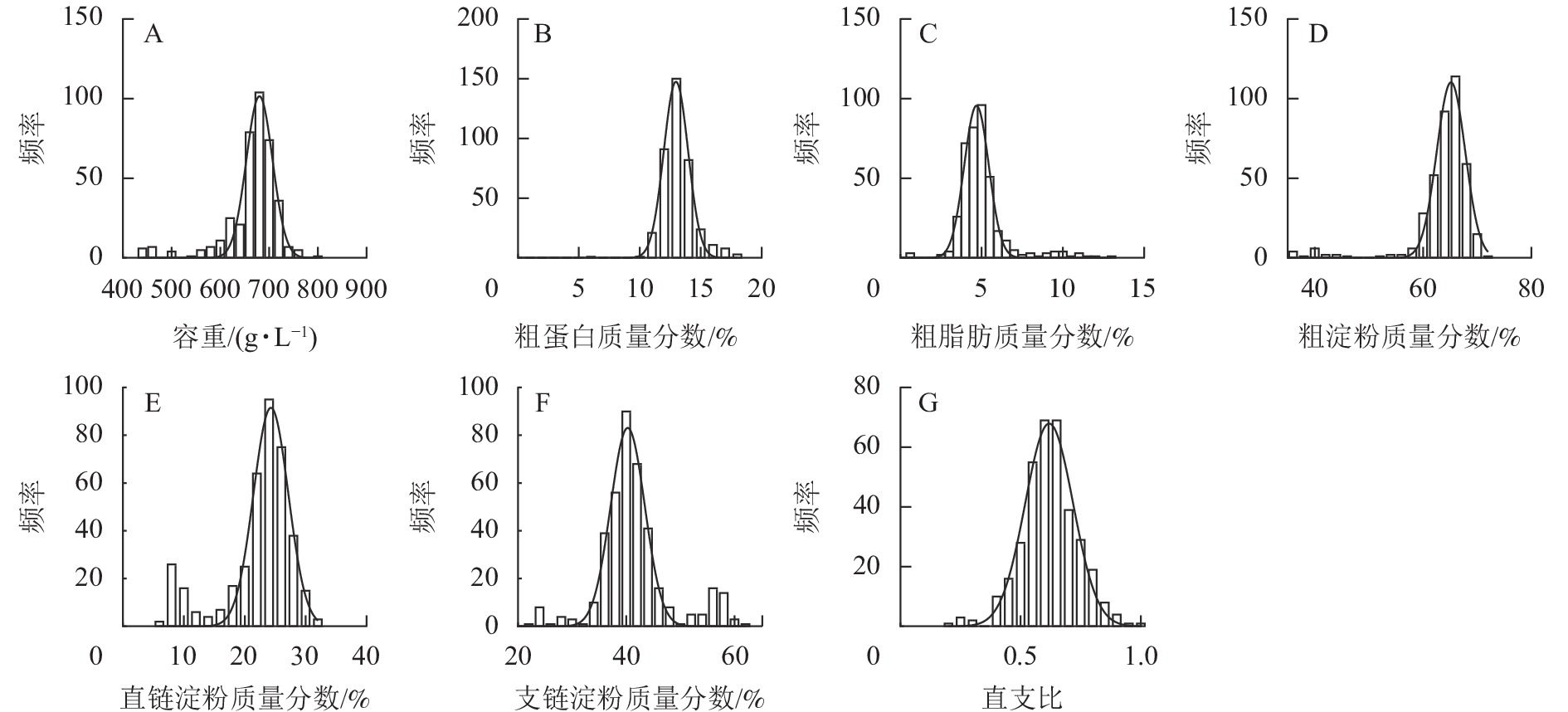
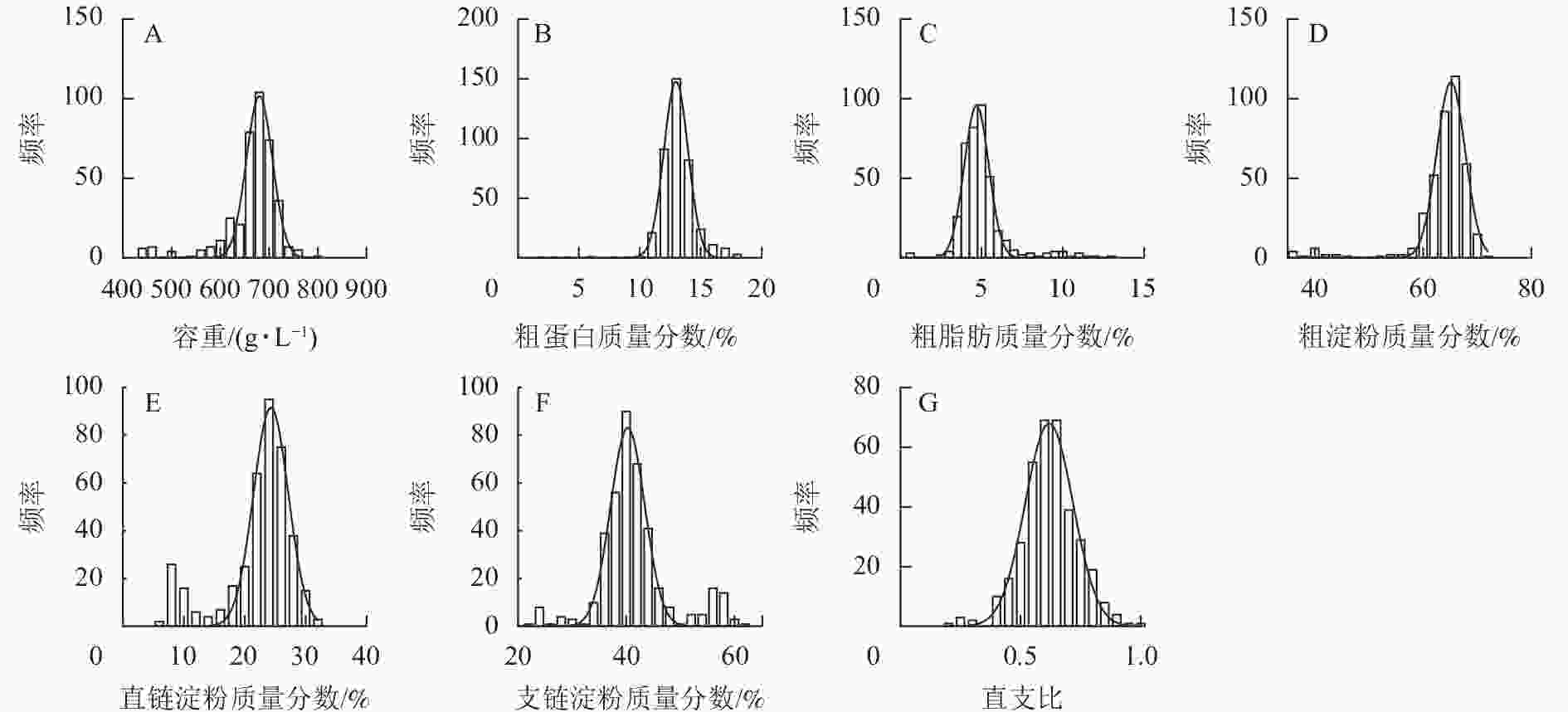
如图1所示:容重主要分布在 650.00~700.00 g·L−1,群体分布呈标准正态分布。粗蛋白、粗脂肪、粗淀粉、直链淀粉、支链淀粉质量分数和直支比分别主要分布在 12.00%~13.00%、4.00%~5.00%、64.00%~68.00%、24.00%~27.00%,40.00%~46.00%,0.60~0.80内。粗蛋白、粗淀粉质量分数的群体分布呈正态分布,粗脂肪、直链淀粉质量分数和直支比的群体分布呈偏态分布。
-
品质性状作为因变量,品种和环境作为自变量进行分析(表2)发现:容重、粗脂肪和粗淀粉质量分数受品种、环境以及品种与环境交互作用的极显著影响(P<0.01)。直链淀粉和支链淀粉的质量分数受品种影响极显著(P<0.01),但不受环境以及品种与环境间交互作用的影响。粗蛋白质量分数、直支比受品种和环境的显著影响(P<0.01或P<0.05),但不受品种和环境之间交互作用的影响。
表 2 不同试验点玉米种质籽粒容重和其他品质性状的联合分析
Table 2. Joint analysis of kernel test weight and other quality traits of maize inbred lines in different experimental sites
性状 变异来源 平方和 F P 性状 变异来源 平方和 F P 容重 品种 415221.072 100.721 <0.001** 直链淀粉 品种 4869.787 135.622 <0.001** 环境 159948.328 38.799 <0.001** 环境 91.143 2.538 0.112 品种×环境 13620.659 3.304 0.003** 品种×环境 65.838 1.834 0.140 粗蛋白 品种 171.774 50.379 <0.001** 支链淀粉 品种 5845.288 86.835 <0.001** 环境 94.612 27.749 <0.001** 环境 59.727 0.887 0.347 品种×环境 4.487 1.316 0.268 品种×环境 80.387 1.194 0.311 粗脂肪 品种 317.333 171.032 <0.001** 直支比 品种 3.630 71.754 <0.001** 环境 14.765 7.958 0.005** 环境 0.284 5.613 0.018* 品种×环境 7.527 4.057 0.007** 品种×环境 0.038 0.758 0.518 粗淀粉 品种 6118.363 164.49 <0.001** 环境 330.135 8.876 0.003** 品种×环境 240.086 6.455 <0.001** 说明:*表示P<0.05;**表示P<0.01。 -
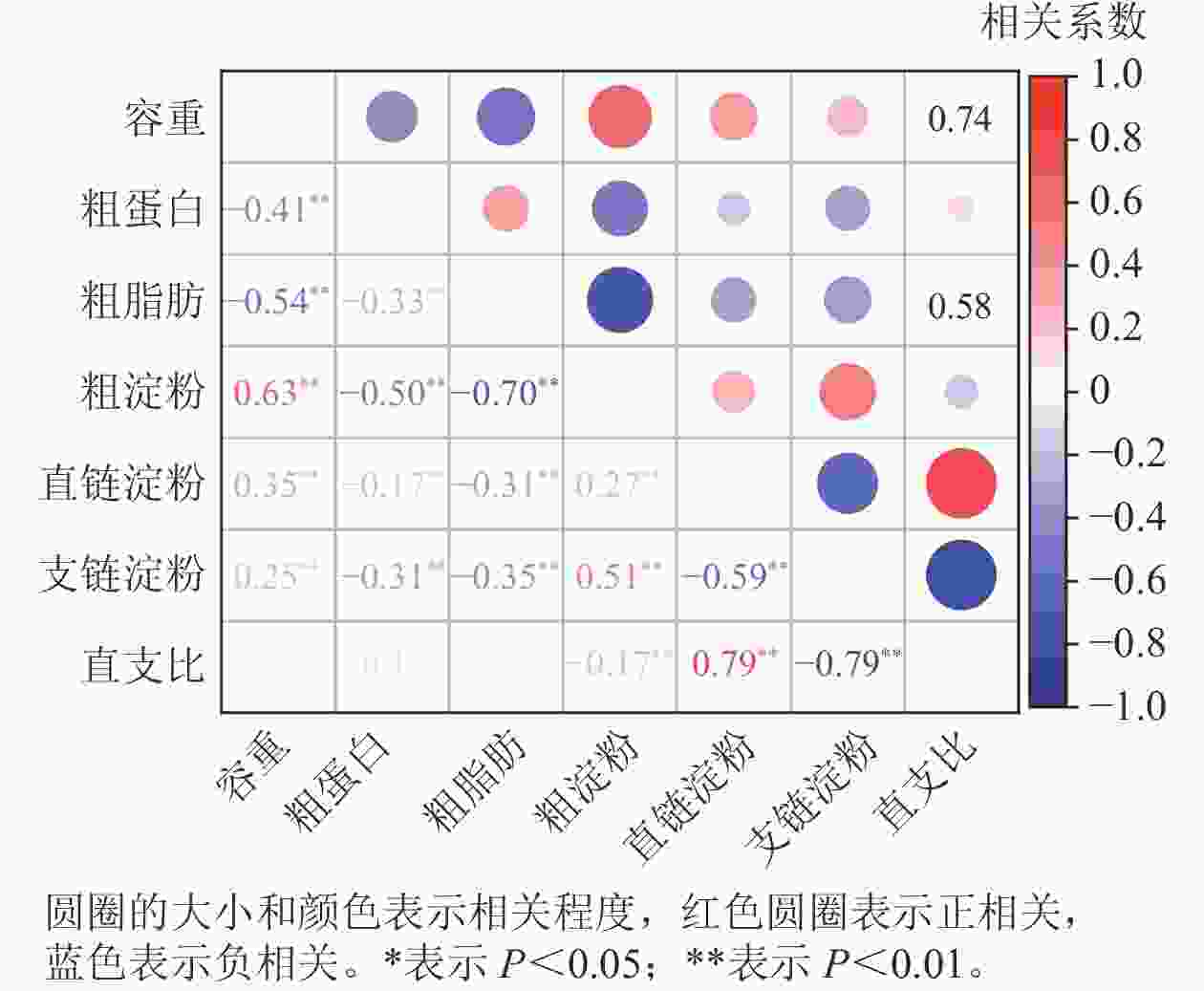
相关性分析(图2)显示:容重与粗蛋白、粗脂肪质量分数呈极显著负相关(P<0.01),而与粗淀粉、直链淀粉和支链淀粉质量分数呈极显著正相关(P<0.01)。容重与直支比之间无相关性。粗蛋白、粗脂肪质量分数与粗淀粉、直链淀粉和支链淀粉呈极显著负相关(P<0.01),而粗蛋白与粗脂肪质量分数呈极显著正相关(P<0.01)。粗淀粉与直链淀粉、支链淀粉质量分数呈极显著正相关(P<0.01),而与直支比呈极显著负相关(P<0.01)。直链淀粉与支链淀粉质量分数呈极显著负相关(P<0.01),而与直支比呈极显著正相关(P<0.01)。支链淀粉质量分数与直支比呈极显著负相关(P<0.01)。
-
通过主成分分析,容重较高、粗淀粉质量分数较高、粗蛋白质量分数较低的种质被划分为 A 组,而容重较低、粗淀粉质量分数较低、粗蛋白质量分数较高的种质被划分为 B 组(图3A),说明 A 组和 B 组之间的基因表达量有显著性差异。在主成分分析中,PC1 可解释 43.9% 的表型,PC2 可解释 15.7% 的表型(图3B)。与 A 组相比,B1、B2、B3、B4中分别有1 847、4 729、2 273、1 918个基因上调,而 B1、B2、 B3 、 B4 中分别有 391 、 350 、 433 、 352 个基因下调(图3C~D)。经过韦恩分析发现:其中140个基因在B组中共同上调,19个基因受到下调。对上述159个基因进行基因本体(GO)富集分析,发现分别有 50 和 47 个基因富集于细胞过程、生物过程的代谢,54 个基因富集在细胞构造及组成中,46 个基因分别具有结合和催化活性的分子功能(图3E)。通过京都基因与基因组百科全书(KEGG)富集分析,有 7 个基因富集于糖酵解/葡萄糖生成(ko00010),5 个基因富集于光合生物碳固定(ko00710),8 个基因富集于碳代谢(ko01200),7 个基因富集于氨基酸生物合成中的碳固定(ko01230) (图3F)。
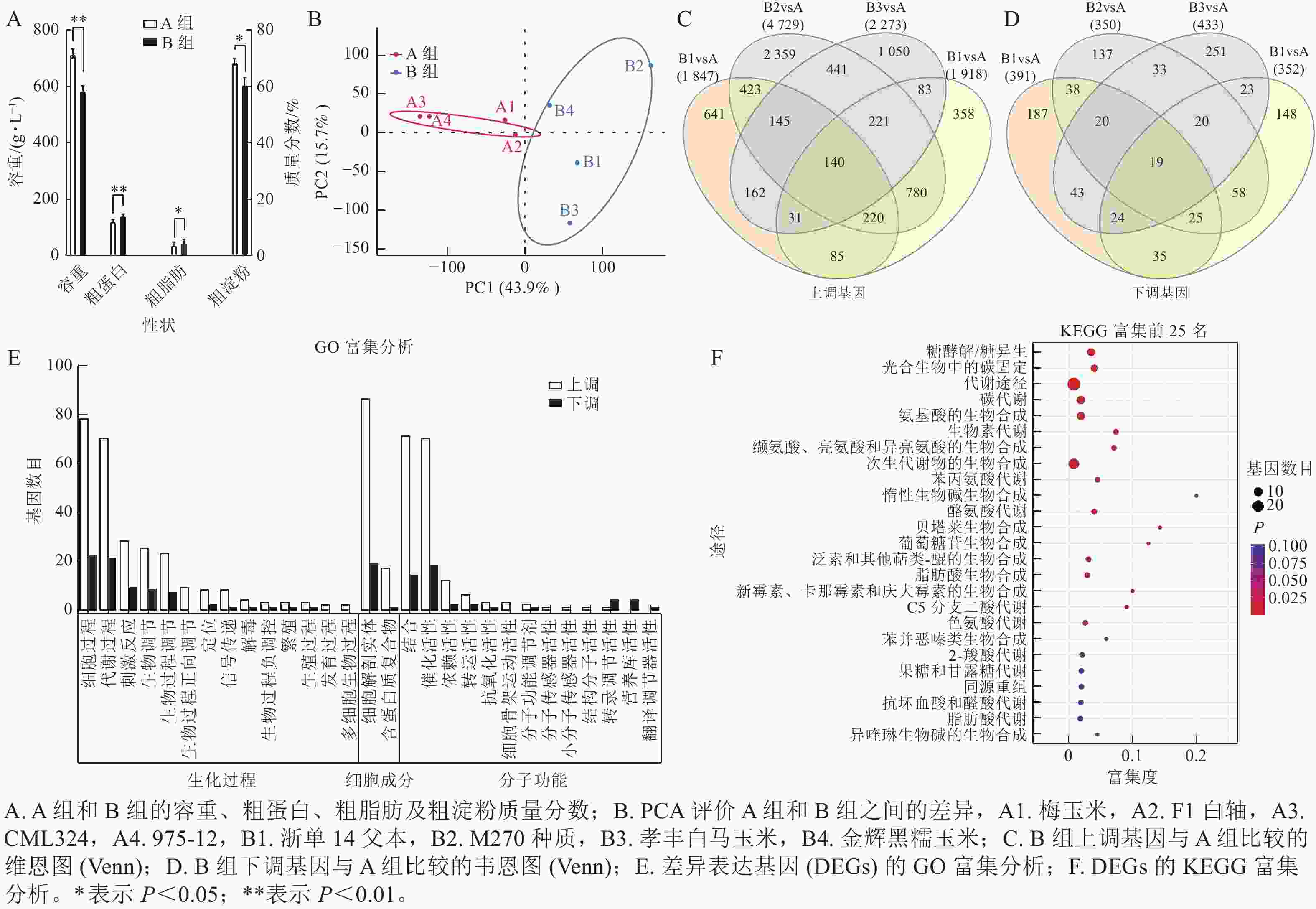
图 3 转录组分析筛选控制容重和品质性状的候选基因
Figure 3. Transcriptome screening of control genes for traits such as bulk density and quality
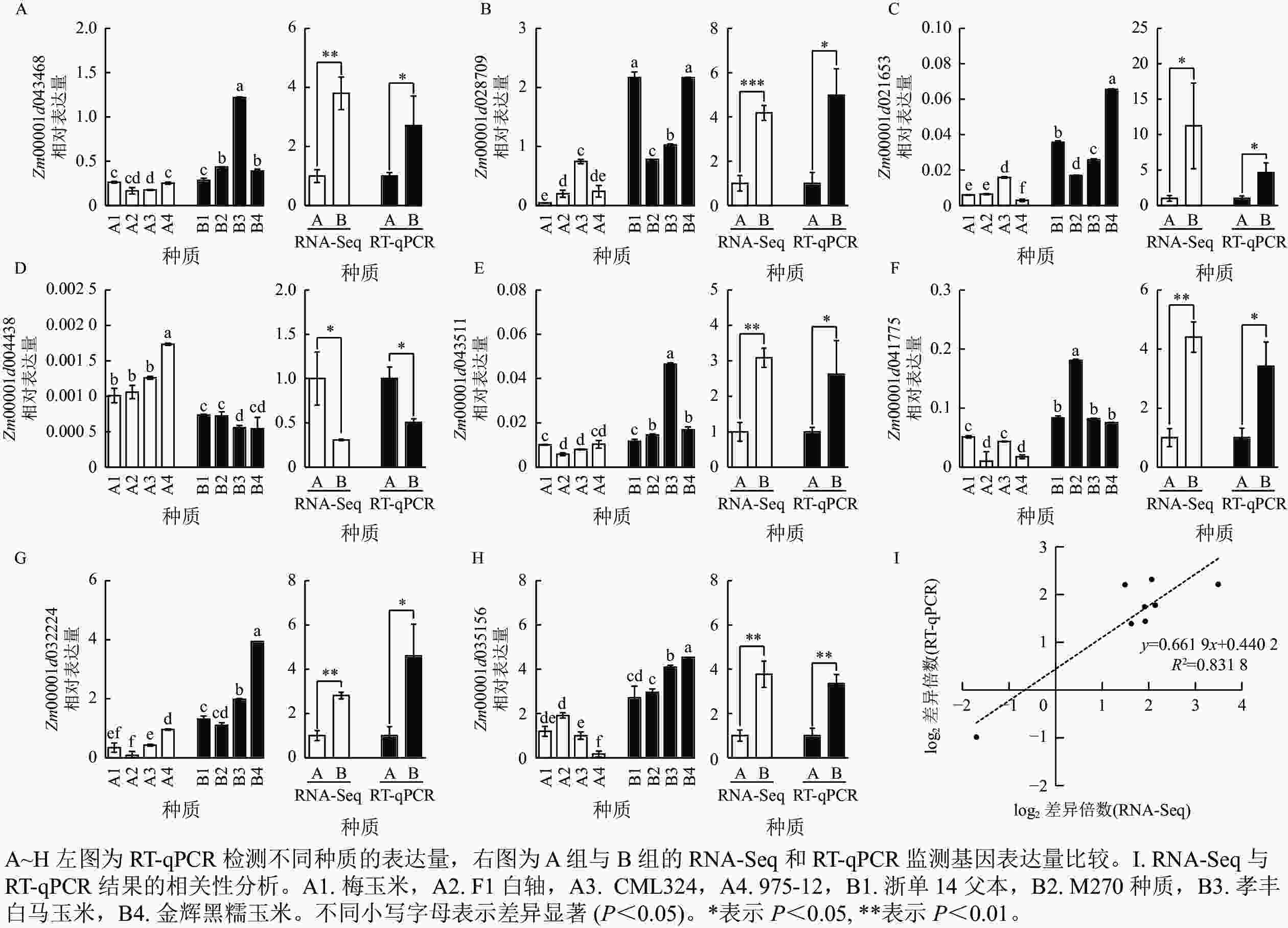
为了进一步挖掘控制容重和品质性状的关键基因,从与碳代谢、光合组织中的碳固定以及氨基酸的生物合成相关途径中筛选出 8 个候选基因,通过 RT-qPCR 或 RNA-Seq 进行了验证分析。通过 RT-qPCR 分析,编码6-磷酸海藻糖合酶6的 Zm00001d043468 在 B2 中的表达量是 A2 的 3.7 倍。RNA-Seq 数据中,Zm00001d043468 的表达水平在 B 组中较 A 组升高 2.8 倍,而通过RT-qPCR检测发现该基因在 B 组中的表达量是 A 组的 2.7 倍 (图4A)。通过 RT-qPCR 分析,编码抗坏血酸过氧化物酶同源基因-3的Zm00001d028709在B1和B4组的表达量是 A1 组的51倍。通过 RNA-Seq 分析发现,Zm00001d028709 的表达水平在 B 组较 A 组升高 3.1 倍,而RT-qPCR分析显示该基因在 B 组中表达量是 A 组的 4.9 倍 (图4B)。通过RT-qPCR 分析,编码葡萄糖-6-磷酸/磷酸转运体-2 的 Zm00001d021653 在 B4 组的表达量是 A4 组的 21.4 倍。通过 RNA-Seq分析发现 Zm00001d021653 的表达水平在B 组较 A 组升高 10.2 倍,而RT-qPCR实验验证发现该基因在 B 组表达量是 A 组的 4.6倍 (图4C)。通过 RT-qPCR 分析,编码普鲁兰酶型淀粉脱支酶 1 的 Zm00001d004438 在 A4 组的表达量是 B3 组的 1.4 倍。通过 RNA-Seq 分析发现 Zm00001d004438在A 组的表达量较 B 组升高 2.3 倍,而使用RT-qPCR验证发现该基因在 A 组的表达量是 B 组的1.9倍 (图4D)。通过 RT-qPCR分析,编码己糖激酶-6 的 Zm00001d043511 在 B3 组的表达量是 A2 组的 6.9 倍。通过 RNA-Seq 分析发现Zm00001d043511在 B 组的表达量较A 组升高 2.1 倍,而RT-qPCR验证该基因在 B 组表达量是 A 组的 2.6 倍 (图4E)。通过RT-qPCR分析,编码2-脱氢-3-脱氧磷酰辛酸醛缩酶的 Zm00001d041775 在 B2 中的表达量是A2的17倍。通过 RNA-Seq 分析发现Zm00001d041775在 B 组中的表达量较 A 组升高 3.4 倍,而 RT-qPCR检测该基因在B 组的表达量是 A 组的 3.4 倍 (图4F)。通过RT-qPCR分析,编码丙酮酸脱氢酶复合体叶绿体二氢脂酰赖氨酸-残基乙酰转移酶组分-4的 Zm00001d032224 在 B4 组的表达量是A2组的 46.9倍。通过 RNA-Seq 分析发现 Zm00001d032224 在B 组的表达量较 A 组升高 1.8倍,而 RT-qPCR检测该基因在B 组的表达量是 A 组的 4.6倍 (图4G)。通过 RT-qPCR 分析,编码细胞膜甘油醛-3-磷酸脱氢酶GAPC2的Zm00001d035156在B4组的表达量是A4组的25.6倍。通过 RNA-Seq分析发现:Zm00001d035156在B组的表达量较A组升高 2.8 倍,而RT-qPCR显示该基因在B组的表达量是 A 组的3.4倍 (图4H)。从以上结果可以看出:RT-qPCR和 RNA-Seq 检测基因表达的趋势是一致的。相关性分析发现:RT-qPCR 和 RNA-Seq 数据具有很高的相关性,R2 为 0.831 8 (图4I)。
-
容重是一个复杂的数量性状,受多因素影响,但与其他品质性状的关系仍有争议。在玉米中,张丽等[8]研究表明:容重与蛋白质质量分数呈正相关,也有研究发现容重与蛋白质质量分数呈负相关[7, 9]。本研究发现玉米籽粒容重与粗蛋白质量分数呈负相关。向日葵Helianthus annuus中粗脂肪质量分数与容重呈正相关,但在燕麦Avena sativa中无显著关系,在大豆Glycine max中呈负相关[24−26]。在玉米中,吴春胜等[7]发现籽粒容重与脂肪质量分数呈负相关,张静等[9]发现容重与脂肪质量分数呈正相关。本研究发现玉米容重与粗脂肪质量分数呈负相关。玉米籽粒主要由胚和胚乳组成,胚乳质量占了85%,且以淀粉为主,而胚的主要成分是蛋白质、脂肪和水。这可能有助于解释容重与粗脂肪和粗蛋白质量分数之间的负相关关系。张丽等[8]利用29个山东省审定玉米品种研究发现玉米籽粒容重和淀粉呈正相关关系,但是DORSEY-REDDING 等[10]分别使用183和195个玉米杂交种发现玉米籽粒容重和淀粉呈负相关。本研究利用517个玉米种质发现淀粉质量分数和容重呈正相关。此外,容重与直链淀粉或支链淀粉的质量分数之间存在弱的正相关性,而容重与直支比之间没有相关性。这表明容重可能是由淀粉质量分数决定,而不是由淀粉类型决定。
容重是一个可遗传的数量性状,但它也受环境因素的影响。本研究表明:容重、粗脂肪和粗淀粉质量分数受品种、环境或品种和环境交互作用的影响,而直链淀粉和支链淀粉只受品种影响,不受环境影响。这些结果说明在不同环境下的容重可能受到粗蛋白、粗脂肪或粗淀粉质量分数的影响,而不受到直链淀粉和支链淀粉的影响。
在玉米中,ZHANG等[11]利用 B73 × Mo17(IBM)Syn10 DH群体进行 QTL 关联分析,筛选到3个与容重相关的候选基因 ZmSWEET4c、ZmYUC1和 ZmTCRR,这些基因均与胚乳发育及碳代谢有关[11, 27−28]。本研究通过RT-qPCR 方法筛选并验证了 8 个与碳代谢和氨基酸代谢相关的基因。其中,与碳代谢相关的海藻糖-6-磷酸对胚发育、产量形成和非生物胁迫耐受性非常重要[29−30]。在水稻Oryza sativa中,OsTPS8在耐盐性和糖分积累方面起着重要作用[31]。本研究发现编码6-磷酸海藻糖合酶6的 Zm00001d043468与容重和品质性状有关。在拟南芥Arabidopsis thaliana中,基质抗坏血酸过氧化物酶(s-APX)已被证明可通过调节抗坏血酸的生物合成来控制碳水化合物的供应[32]。编码抗坏血酸过氧化物酶同源物 3 并与碳水化合物代谢(K00434)相关的 Zm00001d028709在容重较低的种质中上调,该基因位于容重相关QTL位点 PZE-103144282或qKTW3-2附近[11]。在拟南芥中,葡萄糖-6-磷酸/磷酸转运体 2(GPT2)可增加叶绿体内膜的碳还原,影响光合作用和植物生长[33]。在玉米中,编码葡萄糖-6-磷酸/磷酸转运体2的 Zm00001d021653可能是容重增加的原因。Zm00001d004438编码普鲁兰酶型淀粉脱分支酶1 (Zpu1),可能在胚乳发育过程中起重要作用[34],在容重较高的种质中高表达,该基因位于已报道容重关联位点 SYN18432、qKTW7和phi082-umc1799附近[11, 17]。ZmHXK3a 催化葡萄糖转化为葡萄糖-6-磷酸在淀粉代谢中发挥关键作用[35]。本研究表明编码己糖激酶-6的Zm00001d043511在容重和粗淀粉质量分数较高的种质中高表达。在桑Marus alba叶中,编码 2-脱氢-3-脱氧磷酸醛缩酶的 KdsA 在高盐和干旱胁迫下下调[36]。本研究发现编码 2 -脱氢- 3 -脱氧磷酰辛酸醛缩酶的 Zm00001d041775与容重和品质性状的形成有关,该基因位于容重关联位点PZE-10314428和qKTW3-2附近[11]。在拟南芥中,细胞质甘油醛-3-磷酸脱氢酶GAPC2在细胞代谢和种子脂肪积累中起着重要作用[37]。本研究发现编码细胞质甘油醛-3-磷酸脱氢酶 GAPC2 的 Zm00001d035156 在容重较高的种质中高表达。
-
群体结构分析表明:玉米籽粒容重,粗蛋白、粗脂肪、粗淀粉、直链淀粉、支链淀粉质量分数和直支比的主要分布符合正态分布或近似正态分布。联合方差分析表明:容重、粗脂肪和粗淀粉质量分数受品种、环境以及品种与环境交互作用的显著影响;直链淀粉和支链淀粉的质量分数受品种影响较大,但不受环境以及品种与环境间交互作用的影响;粗蛋白质量分数、直支比受品种和环境的显著影响,但不受品种和环境之间交互作用的影响。品质性状之间相关性分析表明:容重与粗蛋白、粗脂肪质量分数呈显著负相关,而与粗淀粉、直链淀粉和支链淀粉质量分数呈显著正相关。容重与直支比之间无相关性。
进一步对极端玉米种质籽粒进行RNA-Seq,共鉴定到8个与碳代谢和氨基酸代谢途径相关的基因,并利用RT-qPCR验证其在极端种质中的表达差异,发现这些基因在不同种质中的表达变化规律与品质性状差异变化趋势一致,其中Zm00001d021653、Zm00001d004438、Zm00001d043511、Zm00001d035156可能在碳还原、胚乳发育、淀粉代谢、细胞代谢和种子脂肪积累中起着重要作用。
本研究筛选获得了可能与品质性状相关的候选基因,可以为高品质玉米新品种选育提供特异性种质资源及候选基因,但是对于这些基因调控玉米籽粒品质性状的分子机制仍然不够清晰,还需要进一步通过分子生物学实验来鉴定这些基因的功能,以揭示玉米籽粒品质性状的分子调控机制。
Correlation analysis of test weight and other quality traits and candidate gene mining in 517 maize germplasms
-
摘要:
目的 玉米Zea mays籽粒容重是国家玉米区试标准中品质性状的重要指标之一,是一个复杂的数量性状,目前其影响因素和调控机制尚未完全解析。淀粉、蛋白质、脂肪等品质性状是影响容重的重要因素,但其与容重的关系仍存在争议。本研究旨在阐明容重与其他品质性状之间的相关性,揭示容重的调控机制。 方法 测定以浙江省地方玉米种质资源为主要构成的 517 份玉米种质的容重,粗淀粉、粗蛋白、粗脂肪、直链淀粉质量分数等性状,并进行相关性分析。在此基础上,选取 4 个高容重、高淀粉质量分数、低粗蛋白质量分数、低脂肪质量分数的玉米种质籽粒和 4 个低容重、低淀粉质量分数、高粗蛋白质量分数、高脂肪质量分数的玉米种质籽粒进行转录组测序(RNA-Seq)分析。 结果 容重与总淀粉质量分数呈显著正相关(P<0.01),而容重与粗蛋白和粗脂肪质量分数呈显著负相关(P<0.01)。对极端种质进行RNA-Seq 分析,共筛选获得 159 个差异表达基因,这些基因主要参与碳代谢、光合组织中的碳固定、氨基酸生物合成等过程。进一步实时荧光定量PCR对其中与碳代谢或氨基酸生物合成相关的8个候选基因表达量进行验证,发现这些基因在不同种质中的表达变化规律与容重差异变化趋势一致。 结论 阐明了玉米籽粒容重与其他品质性状之间的相关性,为高品质玉米新品种选育提供特异性种质及8个在碳还原、胚乳发育、淀粉代谢、细胞代谢和种子脂肪积累中起着重要作用的候选基因。图4表2参40 Abstract:Objective As an important factor of quality traits in national maize standard regional tests, test weight is a complex quantitative trait, and its influencing factor and regulatory mechanism is still unclear. Quality traits such as starch, protein, and fat content are important factors affecting test weight, but the relationship between them is still unclear. Method This research determined the test weight, starch, crude protein, gross fat, and amylose content of 517 maize germplasms mainly composed of local maize germplasm resources in Zhejiang Province. On the basis of correlation analysis, the kernels of four germplasms with higher test weight, higher starch content, lower protein content, lower fat content, and four germplasms with opposite phenotypes were selected for RNA-Seq analysis. Result By correlation analysis, this research found there was a significant positive correlation between test weight and starch content, while a negative correlation was found between test weight and protein or fat content. RNA-Seq analysis was performed for the extreme germplasm, a total of 159 differentially expressed genes were screened between 2 groups, and 8 candidate genes related to carbon metabolism or amino acid biosynthesis were used for further analysis. The expression difference of these genes was consistent with the change of test weight in different inbred lines by RT-qPCR. Conclusion The correlation between test weight and other quality traits was preliminarily clarified, providing superior germplasms and 8 candidate genes that play important roles in carbon reduction, endosperm development, starch metabolism, cellular metabolism, and seed fat accumulation for the selection of new high-quality maize cultivars. [Ch, 4 fig. 2 tab. 40 ref.] -
Key words:
- maize /
- kernel test weight /
- quality traits /
- regulation mechanism
-
表 1 玉米籽粒容重和其他品质性状统计及分析
Table 1. Descriptive statistics for maize test weight and other quality traits
项目 地点 容重/(g·L−1) 质量分数/% 直支比 粗蛋白 粗脂肪 粗淀粉 直链淀粉 支链淀粉 均值±标准差 东阳 606.84±101.03 12.48±2.08 4.98±2.00 62.73±9.64 21.87±8.94 40.83±10.64 0.60±0.32 乐东 692.46±69.45 13.98±2.08 5.01±1.70 64.23±6.00 21.89±6.77 42.35±8.30 0.55±0.21 临安 712.90±52.82 12.77±1.35 5.03±1.17 64.88±4.25 21.06±6.85 43.75±8.06 0.52±0.21 BLUP 664.42±56.29 13.17±1.33 5.04±1.46 63.21±6.98 22.10±5.84 41.46±6.90 0.58±0.19 数值范围 东阳 254.55~904.00 7.66~20.30 2.01~14.57 19.95~72.46 2.66~36.44 4.98~65.13 0.07~3.00 乐东 366.67~840.00 9.05~20.70 2.46~13.61 34.36~77.08 3.20~34.12 18.66~65.94 0.06~1.10 临安 431.82~871.43 9.72~17.91 3.13~12.00 36.76~72.17 4.36~31.50 20.70~64.65 0.07~1.17 BLUP 431.41~798.55 6.17~17.89 2.64~12.99 12.45~71.25 5.70~32.68 16.65~61.79 0.16~2.30 变异系数/% 东阳 16.65 16.66 40.13 15.37 40.88 26.07 53.39 乐东 10.03 14.85 33.83 9.34 30.95 19.59 37.44 临安 7.41 10.58 23.29 6.55 32.50 18.41 40.06 BLUP 8.47 10.09 29.01 11.04 26.44 16.65 33.08 表 2 不同试验点玉米种质籽粒容重和其他品质性状的联合分析
Table 2. Joint analysis of kernel test weight and other quality traits of maize inbred lines in different experimental sites
性状 变异来源 平方和 F P 性状 变异来源 平方和 F P 容重 品种 415221.072 100.721 <0.001** 直链淀粉 品种 4869.787 135.622 <0.001** 环境 159948.328 38.799 <0.001** 环境 91.143 2.538 0.112 品种×环境 13620.659 3.304 0.003** 品种×环境 65.838 1.834 0.140 粗蛋白 品种 171.774 50.379 <0.001** 支链淀粉 品种 5845.288 86.835 <0.001** 环境 94.612 27.749 <0.001** 环境 59.727 0.887 0.347 品种×环境 4.487 1.316 0.268 品种×环境 80.387 1.194 0.311 粗脂肪 品种 317.333 171.032 <0.001** 直支比 品种 3.630 71.754 <0.001** 环境 14.765 7.958 0.005** 环境 0.284 5.613 0.018* 品种×环境 7.527 4.057 0.007** 品种×环境 0.038 0.758 0.518 粗淀粉 品种 6118.363 164.49 <0.001** 环境 330.135 8.876 0.003** 品种×环境 240.086 6.455 <0.001** 说明:*表示P<0.05;**表示P<0.01。 -
[1] WANG Zhen, GUO Yanan, WANG Keru, et al. Effects of planting density on test weight and related indexes of maize [J]. Crop Science, 2023, 63(6): 3470 − 3481. [2] PINHEIROBR N, COSTA R, ALMEIDA A, et al. Durum wheat breeding in Mediterranean environments-influence of climatic variables on quality traits [J]. Journal of the Science of Food &Agriculture, 2013, 25(12): 962 − 973. [3] MARINCIU C M, ERBAN G, MANDEA V, et al. Nardi Fundulea, romania cultivar and crop management effects on test weight in winter wheat (Triticum aestivum) [J]. Romanian Agricultural Research, 2021, 38: 1 − 7. [4] QI Xin, LI Shixue, ZHU Yaxi, et al. ZmDof3, a maize endosperm-specific Dof protein gene, regulates starch accumulation and aleurone development in maize endosperm [J]. Plant Molecular Biology, 2017, 93(1): 7 − 20. [5] DAI Dawei, MA Zeyang, SONG Rentao. Maize endosperm development [J]. Journal of Integrative Plant Biology, 2021, 63(4): 613 − 627. [6] MCNEECE B T, GILLENWATER J H, LI Z, et al. Assessment of soybean test weight among genotypes, environments, agronomic and seed composition traits [J]. Agronomy Journal, 2021, 113(3): 2303 − 2313. [7] 吴春胜, 贾士芳, 王成己, 等. 高蛋白玉米、高油玉米与普通玉米品质的对比研究[J]. 玉米科学, 2004, 12(1): 57 − 60. WU Chunsheng, JIA Shifang, WANG Chengji, et al. A comparison of quality among high-protein corn, high-oil corn and common corn [J]. Journal of Maize Science, 2004, 12(1): 57 − 60. [8] 张丽, 董树亭, 刘存辉, 等. 玉米籽粒容重与产量和品质的相关分析[J]. 中国农业科学, 2007, 40(2): 405 − 411. ZHANG Li, DONG Shuting, LIU Cunhui, et al. Correlation analysis on maize test weight, yield and quality [J]. Scientia Agricultura Sinica, 2007, 40(2): 405 − 411. [9] 张静, 王彩红, 赵永锋, 等. 玉米种质资源子粒容重和品质性状差异性分析[J]. 植物遗传资源学报, 2016, 17(5): 832 − 839. ZHANG Jing, WANG Caihong, ZHAO Yongfeng, et al. Difference analysis of kernel test weight and nutritional quality traits in maize( Zea mays L. ) germplasm resources [J]. Journal of Plant Genetic Resources, 2016, 17(5): 832 − 839. [10] DORSEY-REDDING C, HURBURGH C R, JOHNSON L A, et al. Relationships among maize quality factors [J]. Cereal Chemistry, 1991, 68(6): 602 − 605. [11] ZHANG Xiaoxiang, GUAN Zhongrong, WANG Lei, et al. Combined GWAS and QTL analysis for dissecting the genetic architecture of kernel test weight in maize [J]. Molecular Genetics and Genomics, 2020, 295(2): 409 − 420. [12] KRISTENSEN P S, AHMED J, ANDERSEN J R, et al. Genome-wide association studies and comparison of models and cross-validation strategies for genomic prediction of quality traits in advanced winter wheat breeding lines [J/OL]. Frontiers in Plant Science, 2018, 9: 69[2024-01-10]. doi: 10.3389/fpls.2018.00069. [13] CABRAL A L, JORDAN M C, LARSON G, et al. Relationship between QTL for grain shape, grain weight, test weight, milling yield, and plant height in the spring wheat cross RL4452/‘ACDomain’ [J/OL]. PLoS One, 2018, 13(1): e0190681[2024-01-10]. doi: 10.1371/journal.pone.0190681. [14] SUN Xianyin, WU Ke, ZHAO Yan, et al. QTL analysis of kernel shape and weight using recombinant inbred lines in wheat [J]. Euphytica, 2009, 165(3): 615 − 624. [15] REIF J C, GOWDA M, MAURER H P, et al. Association mapping for quality traits in soft winter wheat [J]. Theoretical and Applied Genetics, 2011, 122(5): 961 − 970. [16] SCHULTHESS A W, REIF J C, JIE L, et al. The roles of pleiotropy and close linkage as revealed by association mapping of yield and correlated traits of wheat (Triticum aestivum L. ) [J]. Journal of Experimental Botany, 2017, 68(15): 4089 − 4101. [17] DING Junqiang, MA Jinliang, ZHANG Chunrong, et al. QTL mapping for test weight by using F2: 3 population in maize [J]. Journal of Genetics, 2010, 90(1): 75 − 80. [18] LIU Jie, HUANG Juan, GUO Huan, et al. The conserved and unique genetic architecture of kernel size and weight in maize and rice [J]. Plant Physiology, 2017, 175(2): 774 − 785. [19] NOEL R J. Collaborative study of a semiautomated method for the determination of crude protein in animal feeds [J]. Journal - Association of Official Analytical Chemists, 1976, 59(1): 134 − 140. [20] SUBRAMANIAN R, SUBBRAMANIYAN P, AMEEN J N, et al. Double bypasses soxhlet apparatus for extraction of piperine from Piper nigrum [J]. Arabian Journal of Chemistry, 2016, 9(suppl 1): S537 − S540. [21] ZHU Thianming, JACKSON D S, WEHLING R L, et al. Comparison of amylose determination methods and the development of a dual wavelength iodine binding technique 1 [J]. Cereal Chemistry, 2008, 85(1): 51 − 58. [22] DUVICK D N. Protein granules of maize endosperm cells [J]. Cereal Chemistry, 1961, 38: 374 − 385. [23] CHEN Jian, ZENG Biao, ZHANG Mei, et al. Dynamic transcriptome landscape of maize embryo and endosperm development [J]. Plant Physiology, 2014, 166(1): 252 − 264. [24] MIJIĆ A, LIOVIĆ I, ZDUNIĆ Z. Quantitative analysis of oil yield and its components in sunflower (Helianthus annuus L. ) [J]. Romanian Agricultural Research, 2009, 26(26): 41 − 46. [25] HOLLAND J B, FREY K J, HAMMOND E G. Correlated responses of fatty acid composition, grain quality and agronomic traits to nine cycles of recurrent selection for increased oil content in oat [J]. Euphytica, 2001, 122(1): 69 − 79. [26] SHEA Z, SINGER W M, ROSSO L, et al. Determining genetic markers and seed compositions related to high test weight in Glycine max [J]. Plants, 2023, 12(16): 1 − 12. [27] BERNARDI J, LANUBILE A, LI Qinbao, et al. Impaired auxin biosynthesis in the defective endosperml8 mutant is due to mutational loss of expression in the ZmYuc1 gene encoding endosperm-specific YUCCA1 protein in maize [J]. Plant Physiology, 2012, 160(3): 1318 − 1328. [28] MUÑIZ L M, ROYO J, GÓMEZ E, et al. The maize transfer cell-specific type-a response regulator ZmTCRR-1 appears to be involved in intercellular signalling [J]. The Plant Journal, 2006, 48(1): 17 − 27. [29] NUCCIO M L, WU J, MOWERS R, et al. Expression of trehalose-6-phosphate phosphatase in maize ears improves yield in well-watered and drought conditions [J]. Nature Biotechnology, 2015, 33(8): 862 − 869. [30] LI Zhiyong, WEI Xiangjin, TONG Xiaohong, et al. The OsNAC23-Tre6P-SnRK1a feed-forward loop regulates sugar homeostasis and grain yield in rice [J]. Molecular Plant, 2022, 15(4): 706 − 722. [31] VISHAL B, KRISHNAMURTHY P, RAMAMOORTHY R, et al. OsTPS8 controls yield-related traits and confers salt stress tolerance in rice by enhancing suberin deposition [J]. New Phytologist, 2019, 221(3): 1369 − 1386. [32] HEIBER I, CAI W, BAIER M. Linking chloroplast antioxidant defense to carbohydrate availability: the transcript abundance of stromal ascorbate peroxidase is sugar-controlled via ascorbate biosynthesis [J]. Molecular Plant, 2014, 7(1): 58 − 70. [33] WEISE S E, LIU T, CHILDS K L, et al. Transcriptional regulation of the glucose-6-phosphate/phosphate translocator 2 is related to carbon exchange across the chloroplast envelope [J/OL]. Frontiers in Plant Science, 2019, 10: 827[2024-01-10]. doi: 10.3389/fpls.2019.00827. [34] CHEN Erwang, YU Huiqin, HE Juan, et al. The transcription factors ZmNAC128 and ZmNAC130 coordinate with Opaque2 to promote endosperm filling in maize [J]. The Plant Cell, 2023, 35(11): 4066 − 4090. [35] XIAO Yingni, THATCHER S, WANG Min, et al. Transcriptome analysis of near-isogenic lines provides molecular insights into starch biosynthesis in maize kernel [J]. Journal of Integrative Plant Biology, 2016, 58(8): 713 − 723. [36] YANG X B, WU S L, ZHU D P, et al. Expression of the 2-dehydro-3-deoxyphosphooc-tonate aldolase (KdsA) gene in mulberry leaves (Morus alba L. ) is down-regulated under high salt and drought stress [J]. Genetics and Molecular Research:GMR, 2015, 14(4): 11955 − 11964. [37] GUO Liang, MA Fangfang, WEI Fang, et al. Cytosolic phosphorylating glyceraldehyde-3-phosphate dehydrogenases affect arabidopsis cellular metabolism and promote seed oil accumulation [J]. The Plant Cell, 2014, 26(7): 3023 − 3035. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20240145






 下载:
下载: