-
植物病害是导致现代农林业减产的主要原因,及时准确的植物病害识别技术是实施有效防治的关键。在实际生产中,植物病害识别主要依靠人工肉眼观察及经验判断,需要人们在实地进行持续监测[1-2]。这种人工评估方法耗时费力且具有一定的主观性,阻碍了现代农林业的快速发展,因此,快速准确的植物病害自动识别成为了精准农业、高通量植物表型和智能温室等领域的研究热点[3-4]。基于图像处理的植物病害识别方法得到了广泛的研究和应用。早期的识别过程需要从图片中分割病斑,人工提取病斑特征,再利用机器学习算法对特征进行分类。HIARY等[5]提取病斑的纹理特征,采用k-means聚类算法和人工神经网络(artificial neural network,ANN)对5种植物病害进行识别,准确率达94%。TIAN等[6]提出用基于支持向量机(support vector machine,SVM)的多分类器识别小麦Triticum aestivum叶部病害。秦丰等[7]对4种苜蓿Medicago叶部病害进行识别研究,分析比较了多种分割方法、特征选择和分类方法。虽然以上方法在特定场景取得了较好效果,但仍无法实现病害的现场实时诊断。这些方法极大程度上基于阈值的病斑分割算法,对亮度、物体形态和遮挡程度都非常敏感[8−9],都只适合背景单一且对比度高的扫描式图像。此外,特征提取和选择复杂耗时,仅局限于有限几种病害,难以处理复杂背景的大数据。近年来,深度学习在计算机视觉领域取得重大突破。深度卷积网络神经网络(convolutional neural network,CNN)可在大数据中自动端到端提取特征,避免了人工图像分割和特征工程[10]。MOHANTY等[11]针对PlantVillage数据集[11] 54 306张植物病害图像,使用AlexNet[12]和GoogLeNet[13]识别38种植物病害。孙俊等[14]在同样的数据集上,将AlexNet进行改进,提出一种批归一化与全局池化相结合的识别模型。龙满生等[15]采用参数精调的迁移学习方式训练AlexNet,用于油茶Camellia oleifera病害图像识别。张建华等[16]基于改进的VGG16模型,通过迁移学习实现自然条件下棉花Anemone vitifolia病害图像分类。DECHANT等[17]提出了集成多个CNN的方法,实现玉米Zea mays大斑病图像的高精度识别。PICON等[18]利用深度残差网络ResNet对3种早期小麦病害进行识别,改善了复杂背景下的病害识别率。通常深度学习模型部署在云平台,需要将拍摄图像上传至云平台进行识别。但这种方法严重依赖高速的4G/5G无线网络和强大的云平台,不仅无法覆盖广大偏远农田林地,长时间大范围的上传与识别还导致能耗、流量及云服务成本大幅上涨,限制了物联网的建设规模。然而,目前的监控设备借助低成本低功耗加速芯片,即可支持边缘计算,仅在发现病害时通过低功耗广覆盖的NB-IoT网络[19]上报,可显著降低网络及云服务成本,促进大规模的农林业物联网普及。但现有的CNN模型计算量和参数量过大,不适用于边缘部署。轻量级模型MobileNet[20]在速度和精度两者间达到了一个较好的均衡,但其目标平台是手机等高端嵌入式平台,参数量及运算量仍超过PaddlePi等廉价边缘设备的承受能力。近年来,学术界也提出了多种模型压缩方法。模型通道剪枝[21]剪裁掉模型一部分冗余或低权重的卷积核,减少模型的参数量。量化[22]将模型由32 bit浮点数转化为定点整数,减少模型参数占用的空间。然而上述压缩方法仅应用于ResNet等重量级模型,尚未对MobileNet等轻量级模型压缩进行优化,而且这些压缩方法彼此相互独立,未能联合使用实现模型的深度压缩。为解决上述问题,本研究提出了面向边缘计算的植物病害识别模型构建方法,主要贡献为:①首次针对轻量级模型MobileNet实现深度压缩。②通过联合通道剪枝、量化等多种模型压缩方法,得到了深度压缩的轻量级边缘端模型,可在廉价边缘节点运行。③将模拟学习方法[23]与量化相结合,实现模型压缩的同时,提升识别效果,最后得到的边缘端模型可达到与原模型相近的识别准确率。
-
本研究使用PlantVillage植物病害数据集。PlantVillage既包含单一背景下的植物叶片扫描式图像,也收录自然背景下的植物叶片图像,包括叶片重叠、阴影和土壤干扰等情形。截至目前已收集了87 280张图像,包括25种植物和29种病害组成的58类植物-病害组合(图1)。
数据集按图1所示的编号将各种叶片归类并制作标签。随机抽取数据集中60%图像作为训练集,剩余的40%作为测试集。单一背景图像与自然背景图像使用相同的分割比例。由于PlantVillage数据集包含从不同角度对同一叶片拍摄的多张图像,因此相同叶片的图像仅存在于训练集或测试集中。
-
选择轻量级卷积神经网络MobileNet[20]作为本研究的基准模型。MobileNet模型将传统的卷积分解为一个深度卷积(depthwise convolution,DC)和一个卷积核为1×1的逐点卷积(pointwise convolution,PC),计算速度比传统卷积快8~9倍,主要面向智能手机等高端嵌入式系统。为深度压缩MobileNet,本研究提出了如图2所示的面向边缘计算的植物病害识别模型二阶段构建方法。
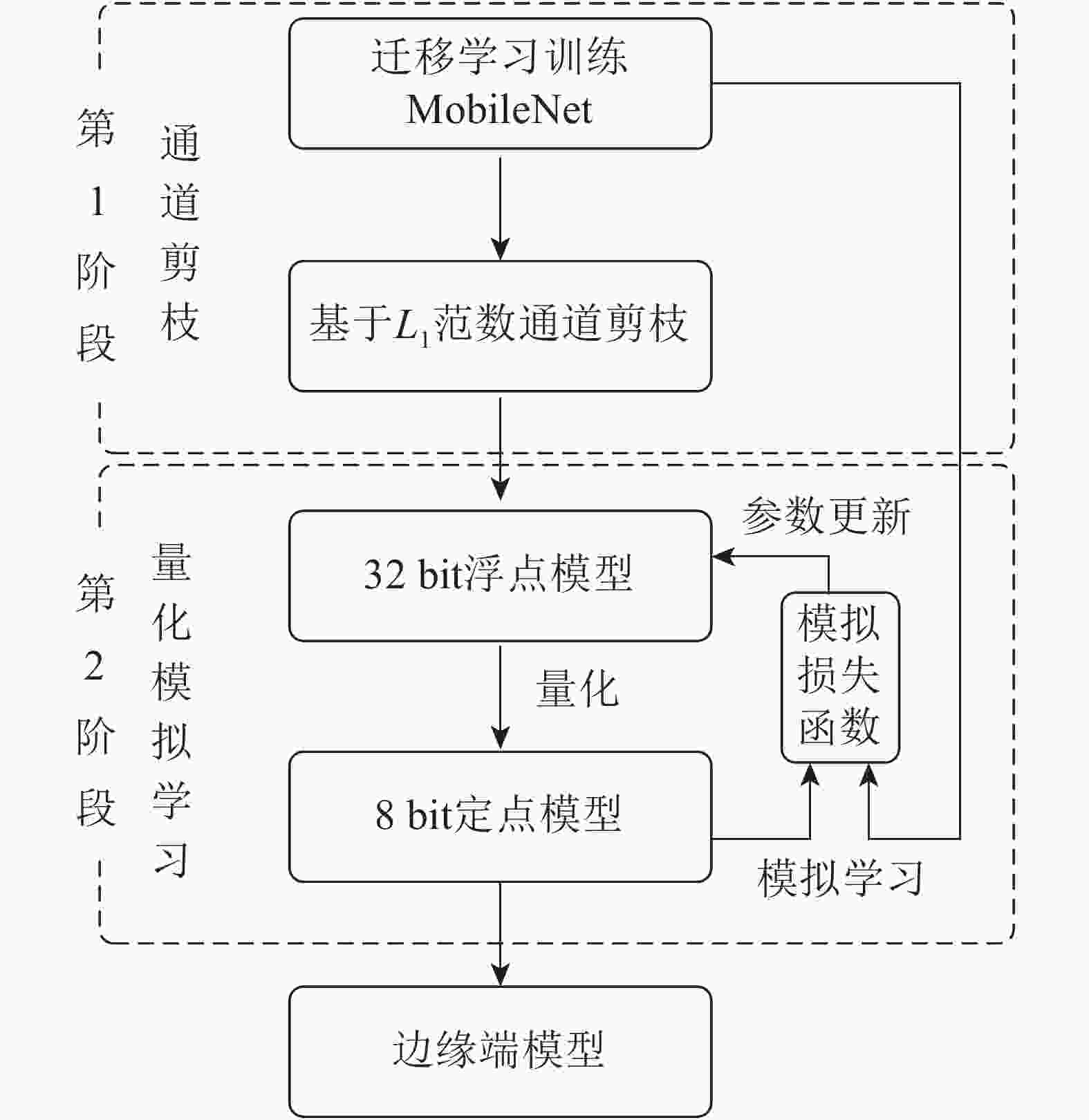
图 2 面向边缘计算的植物病害识别模型构建方法:通道剪枝和量化模拟学习
Figure 2. Plant disease recognition model for edge computing building pipeline: channel pruning and quantized mimic learning
第1阶段使用通道剪枝压缩迁移学习训练的MobileNet模型。与从头训练方法相比,迁移学习可以有效提升模型的识别准确率。迁移学习是用ImageNet数据集上预训练好的参数初始化模型,然后在PlantVillage数据集上通过标准多分类损失函数优化模型参数。最后使用基于L1范数的通道剪枝[21]精简低权值的卷积核,同时将该卷积核所有的输入输出连接从网络中删除,降低了模型计算量和存储空间。
第2阶段对剪枝后的模型通过量化方法进一步压缩,得到轻量级的边缘端模型。量化是将模型的权值和激活值由32 bit降低至8 bit,分为训练时量化和训练后量化。虽然训练时量化方法更适用于轻量级模型,但直接使用该方法压缩剪枝后的MobileNet模型仍会导致识别精度显著下降,因此第2阶段将模拟学习与训练时量化相结合,利用迁移学习的MobileNet监督剪枝后模型的量化训练过程,实现量化模拟学习,在模型压缩的同时提升识别准确率。
-
模型通道剪枝采用均匀剪枝方法对MobileNet模型的分离卷积层进行通道剪枝,即每层都减掉同样比例的卷积核。依据需要减少模型的浮点运算数量(floating point operations, FLOPs)来确定每层的剪枝比例。计算各层中每个卷积核权值的绝对值和(即L1范数),L1范数越大,代表该卷积核对模型的贡献越大,反之越小。每层按L1范数由高到低的顺序排序卷积核,优先剪枝L1范数低的卷积核。为实现模型的深度压缩,需进行较高比例的通道剪枝,分别对模型减掉70%、80%和90%的FLOPs。
-
通常训练时,量化的损失函数是标准的多分类损失函数。量化模拟学习是用模拟学习损失函数作为训练时量化的损失函数。模拟学习方法使剪枝量化后模型的输出特征尽量接近迁移学习训练的MobileNet输出特征。利用2个输出特征之间的L2范数作为模拟损失函数,即:
$$ {L_{L_2}}\left( {{W_{\rm{s}}},{W_{\rm{t}}}} \right) =|| F\left( {x;{W_{\rm{t}}}} \right) - F\left( {x;{W_{\rm{s}}}} \right)||_2^2{\text{。}} $$ (1) 式(1)中:Wt和Ws分别是迁移学习训练的MobileNet和剪枝量化后模型的权值矩阵,F (x; Wt)和F(x; Ws)分别表示这2个模型的输出特征值。
剪枝量化后模型的输出特征再经Softmax归一化得到预测类别概率,与分类标签比较后得到交叉熵,作为标准多分类损失函数Lclass(Ws)。模拟学习的完整损失函数就是分类损失函数与模拟损失函数的权重和:
$$ L\left( W \right) = {L_{\rm{class}}}\left( {{W_{\rm{s}}}} \right) + \alpha {L_{{L}_{{\rm}2}}}\left( {{W_{\rm{s}}},{W_{\rm{t}}}} \right){\text{。}} $$ (2) 式(2)中:α为平衡损失权重的超参数。相较于普通多分类问题的损失函数,模拟学习方法可提供额外的监督信息。
将训练时量化方法与模拟学习相结合,实现量化模拟学习具体的训练步骤为:①在训练的前向传播中,将模型的权值wf和激活值af进行量化得到定点值wq和aq,对于浮点数x具体的量化过程为:
$$ {x_{{\rm{int}}}} = {\rm{round}}\left( {\frac{x}{\varDelta }} \right);\; {x_{\rm{Q}}} = {\rm{clamp}}\left[ { - \left( {\frac{N}{2} - 1} \right),\frac{N}{2} - 1,{x_{{\rm{int}}}}} \right]{\text{。}} $$ (3) xQ即为得到的量化值。其中:clamp函数对于输入的变量a,b,c输出为:
$$\begin{aligned} {\rm{clamp}}(a,b,c) & = a\;\;\;\;x {\text{≤}} a \\ & = x\;\;\;a {\text{<}} x {\text{≤}} b \\ & = b\;\;\;x {\text{>}} b\text{。} \end{aligned}$$ (4) 也就是将浮点数除以缩放因子Δ,再最近邻取整,最后把范围限制到1个区间内。N与量化后整数类型占用的比特数有关。本研究采用有符号8 bit整数类型,N=256。对于权值,每层权值的最大绝对值作为缩放因子。对于激活值,计算各训练批次激活的最大绝对值的滑动平均值作为缩放因子。②计算剪枝量化后模型对迁移学习训练的MobileNet进行模拟学习的损失函数,即计算公式(2),得到损失值L(wq)。③后向传播过程,利用步骤②得到的损失函数值对量化之后的权值求梯度,公式为
$ \dfrac{{{\rm{\partial}} L\left( {{w_q}} \right)}}{{{\rm{\partial}} {w_q}}}$ 。④用步骤③计算梯度去更新量化前的浮点值,也就是将模型的权值反量化回有误差的浮点类型。公式为$ {w_{\rm{f}}} = {w_{\rm{f}}} - v\dfrac{{{\rm{\partial}} L\left( {{w_q}} \right)}}{{{\rm{\partial}} {w_q}}}$ ,其中:ν为学习率。因此,模型的后向传播过程仍然是浮点数计算。⑤重复步骤①至步骤④,直至完成训练。最后再对模型按照步骤①量化,得到最终的边缘端模型。 -
模型实现和训练采用的软件环境为Ubuntu1 6.04操作系统和PaddlePaddle深度学习框架,硬件环境为GPU工作站,使用NVIDIA Titan X显卡(12 GB显存)和AMD Ryzen 7 1700X处理器(32 GB内存)。采用模型的平均识别准确率(accuracy)作为衡量模型精度的标准。同时为了更好地评价模型的鲁棒性,将每类病害样本分别进行测试,计算每个类别的查准率(precision)、查全率(recall)以及查全率与查准率的加权平均分数,并在所有类别上求平均。
-
训练CNN模型需要对输入图片进行预处理。首先,利用数据增广技术对原图像进行变换,将训练图像变换为256×256大小,然后再随机剪枝成224×224,再进行随机水平翻转和随机垂直翻转。该过程极大扩充了训练数据集的多样性,可提升CNN模型的准确率,降低网络过拟合的风险。之后,计算训练集的红(R)、绿(G)、蓝(B)3个颜色通道的均值和方差,所有图像都减去该均值,除以方差,得到归一化后的数据作为CNN的输入,可加速训练过程收敛。对于测试集中的每一张图片,需要变换至224×224大小,减去训练集各通道均值,除以其方差进行归一化后就可以输入CNN模型进行识别。
利用迁移学习训练MobileNet,使用ImageNet数据集预训练的参数初始化模型,采用批量训练的方法将训练集分为多个批次(batch),使用随机梯度下降算法来实现模型优化,批次大小为32,遍历1次训练集中的所有图片作为1个周期(epoch),共迭代50个周期,初始学习率为0.005,动量值为0.9,之后每迭代20个周期就将学习率减小为原来的0.1倍。训练好的模型参数量为3.3 M,识别准确率为96.23%,查准率、查全率和加权平均分数分别为96.62%、95.46%和95.75%。
-
研究不同压缩率下本研究方法的有效性,使用不同的剪枝率,分别对模型减掉不同比例的FLOPs。结果表明:当剪枝率低于60%时,即使使用无模拟训练方法重新训练模型,得到的识别准确率与原MobileNet模型差别很小,说明原模型在该数据集上具有较高的冗余性,只有当剪枝率高于70%时,才能体现不同压缩方法表现的差距。因此,设置剪枝率为70%、80%和90%,对应的模型参数量大小为0.91、0.58和0.23 M,模型的参数量压缩了3.6、5.7、14.3倍,量化又将精度由32 bit降低至8 bit,压缩率为4倍,得到的边缘端模型的整体压缩率分别为14.4、22.8和57.2倍。为快速恢复剪枝后模型精度,首先利用模拟学习损失函数进行30个周期的32 bit浮点模型训练,使用随机梯度下降算法优化模型,批次大小为32,初始学习率为0.005。之后,每迭代15个周期就将学习率减小为原来的0.1倍。公式(2)的α值设置为1。之后再进行20个周期的量化模拟学习,学习率为0.005,公式(2)的α值为0.1,其余超参数值不变。训练结果如表1所示。
表 1 边缘端模型植物病害识别结果
Table 1. Plant disease recognition results of models on the edge
剪枝率/% 参数量/M 剪枝压缩率/倍 量化压缩率/倍 整体压缩率/倍 准确率/% 查准率/% 查全率/% 加权平均分数/% 70 0.91 3.6 4 14.4 95.99 96.18 94.41 94.92 80 0.58 5.7 4 22.8 95.55 95.51 93.52 93.99 90 0.23 14.3 4 57.2 94.58 94.87 92.41 93.15 表1表明:整体压缩率分别为14.4、22.8和57.2倍的边缘端模型,识别准确率分别为95.99%、95.55%和94.58%,与迁移学习训练的MobileNet模型相比仅下降了0.24%、0.68%和1.65%。同时查准率、查全率和加权平均分数值也表明边缘端模型具有较高的鲁棒性。
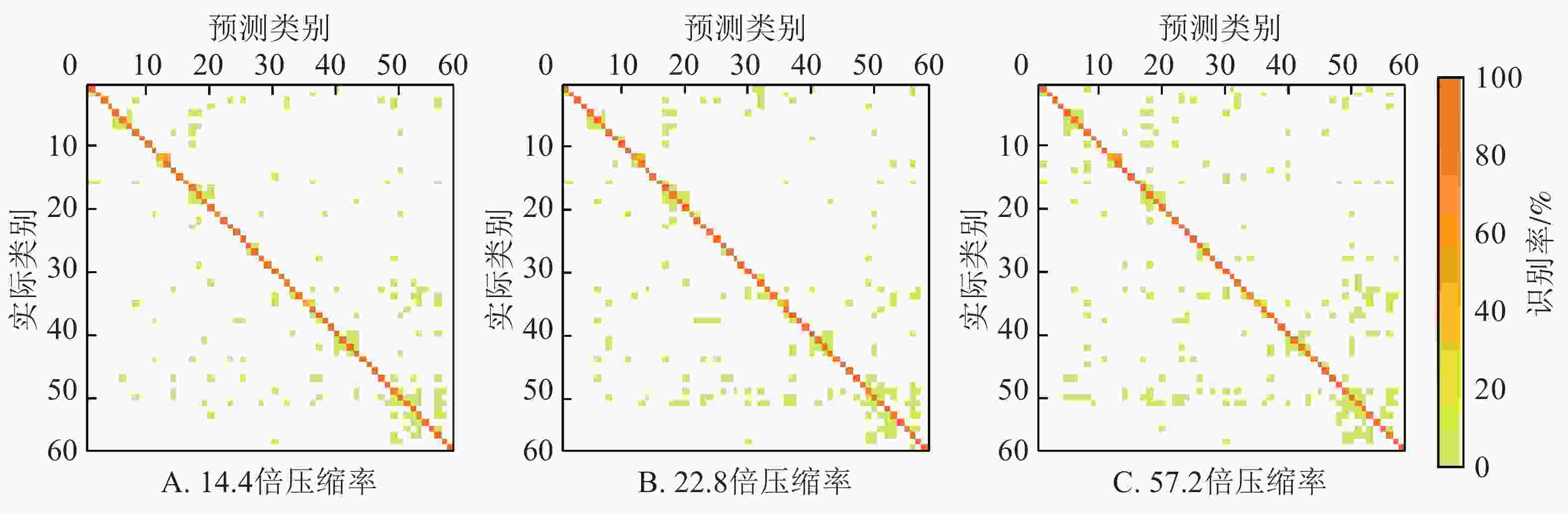
不同压缩率的边缘端模型在测试集的混淆矩阵如图3所示。图3列出了58个类中的每类被正确分类的比例(对角线上的值)和被误识别为其他类的比例(非对角线上的值)。每类的编号与图1一致。可以看出:边缘端模型对不同植物的不同病害均具有较强的识别能力,但不同病害识别结果之间存在着较大的差异。58类病害中,这3个边缘端模型的识别准确率均超过90%的有43类,均超过80%的有51类,均超过70%的有55类。其中有11号哈密瓜健康叶、24号葫芦霜霉病、25号葡萄健康叶、39号树莓健康叶、46号草莓健康叶这5类的识别准确率在3个模型均达到了100%。识别效果最差,在3个模型上识别准确率几乎均低于70%的病害是12号木薯褐斑病(3个模型识别率分别为48.15%、58.33%、48.15%),35号马铃薯健康叶(3个模型识别率分别为48.33%、33.33%、48.33%),21号黄瓜健康叶(3个模型识别率分别为67.92%、66.04%、77.36%)。这些病害大多都被误识别为外形相似的其他病害,例如12号木薯褐斑病被误识别为13号木薯绿螨病,35号马铃薯健康叶被误识别为病斑较小的36号马铃薯晚疫病,21号黄瓜健康叶被误识别为11号哈密瓜健康叶。
-
为进一步测试边缘端模型性能,分别在剪枝率70%、80%和90%的条件下,利用无模拟学习方法,即标准的多分类损失函数分别训练通道剪枝后模型和通道剪枝并量化模型,训练的超参数与本研究的训练超参数一致,与本研究模型进行对比实验。从表2可见:在不同的剪枝率的情况下,本研究模型与其他模型压缩方法相比均具有更高的模型压缩率和识别准确率,而且压缩率越高,识别准确率相比其他方法提升越明显,能更好识别植物病害类别并部署于边缘设备。
表 2 不同压缩方法边缘端模型植物病害识别结果
Table 2. Plant disease recognition results of models on the edge compressed by different methods
剪枝率/% 参数量/M 边缘端模型 精度/bit 压缩率/倍 准确率/% 70 0.91 剪枝+无模拟学习 32 3.6 95.48 剪枝+量化+无模拟学习 8 14.4 95.45 本研究模型 8 14.4 95.99 80 0.58 剪枝+无模拟学习 32 5.7 94.95 剪枝+量化+无模拟学习 8 22.8 94.92 本研究模型 8 22.8 95.55 90 0.23 剪枝+无模拟学习 32 14.3 93.40 剪枝+量化+无模拟学习 8 57.2 93.53 本研究模型 8 57.2 94.58 -
本研究针对边缘环境下计算资源的限制,在迁移学习训练的MobileNet模型基础上,联合使用2种压缩算法降低模型参数量和运算量,并结合模拟学习恢复识别精度,得到深度压缩的边缘端模型。在PlantVillage的实验结果表明:利用本研究方法对MobileNet进行不同程度的深度压缩,均能够大大减少网络计算量并保留原始识别能力。其中减少70%~90% FLOPs的模型,参数量压缩了3.6~14.3倍,再经过量化模拟学习后整体压缩率为14.4~57.2倍,准确率达到了95.99%~94.58%,较迁移学习训练的MobileNet模型仅降低0.24%~1.65%,同时还具有较高的鲁棒性,对不同植物的不同病害均具有较强的识别能力。实验结果证明了该压缩方法的可行性和有效性。
随着PlantVillage数据集的不断扩展,深度学习模型能更多更准地识别植物病害。本研究提出的模型构建方法可平衡识别的速度和精度,满足植物病害识别边缘部署的需求。
Lightweight plant disease recognition model for edge computing
-
摘要:
目的 传统深度学习模型因参数和计算量过大不适用于边缘部署,在网络边缘的植物病害自动识别是实现长时间大范围低成本作物监测的迫切需求。 方法 联合使用多种模型压缩方法,得到可部署于算力有限的嵌入式系统的轻量级深度卷积神经网络,在边缘节点实现植物病害智能识别。模型压缩分2个阶段:第1阶段利用基于L1范数的通道剪枝方法,压缩MobileNet模型;第2阶段将模拟学习与量化相结合,在模型量化的同时恢复识别精度,得到高精度轻量级的端模型。 结果 在PlantVillage数据集58类植物病害的实验结果表明:通道剪枝将MobileNet压缩了3.6~14.3倍,量化又将模型的参数精度由32 bit降低至8 bit。整体压缩率达到了14.4~57.2倍,识别准确率仅降低0.24%~1.65%。与通道剪枝后无模拟学习训练、通道剪枝结合量化后无模拟学习训练这2种压缩方法相比,具有更高的模型压缩率和识别准确率。 结论 联合使用多种模型压缩方法可以少量的精度损失深度压缩人工智能模型,可为农林业提供面向边缘计算的植物病害识别模型。图3表2参23 Abstract:Objective The traditional deep learning model is not suitable for edge deployment because of too many parameters and too much calculation. Automatic identification of plant diseases on the edge of the network is urgently needed to realize long-term and large-scale low-cost crop monitoring. Method By using multiple model compression methods, a light weight deep convolution neural network was obtained, which could be deployed in the embedded system with limited computing power to realize intelligent identification of plant diseases at edge nodes. The model compression was divided into two stages. The first stage used the channel pruning method based on L1 norm to compress the MobileNet model. In the second stage, simulation learning and quantization were combined to restore the recognition accuracy while the model was quantized, and a high-precision lightweight end model was obtained. Result Experimental results of 58 kinds of plant diseases in PlantVillage dataset showed that channel pruning compressed MobileNet by 3.6−14.3 times, and quantization reduced the parameter accuracy of the model from 32 bit to 8 bit. The overall compress rate reached 14.4−57.2 times, and the recognition accuracy was only reduced by 0.24% to 1.65%. Compared with the pruning method trained by common learning, and pruning with quantization trained by common learning, this method achieved higher compression rate and recognition accuracy. Conclusion The combination of multiple model compression methods can compress the artificial intelligence models in depth with only tiny loss of accuracy, and provide plant disease recognition models for agriculture and forestry based on edge computing. [Ch, 3 fig. 2 tab. 23 ref.] -
Key words:
- deep learning /
- plant disease identification /
- edge computing /
- model compression /
- MobileNet
-
表 1 边缘端模型植物病害识别结果
Table 1. Plant disease recognition results of models on the edge
剪枝率/% 参数量/M 剪枝压缩率/倍 量化压缩率/倍 整体压缩率/倍 准确率/% 查准率/% 查全率/% 加权平均分数/% 70 0.91 3.6 4 14.4 95.99 96.18 94.41 94.92 80 0.58 5.7 4 22.8 95.55 95.51 93.52 93.99 90 0.23 14.3 4 57.2 94.58 94.87 92.41 93.15 表 2 不同压缩方法边缘端模型植物病害识别结果
Table 2. Plant disease recognition results of models on the edge compressed by different methods
剪枝率/% 参数量/M 边缘端模型 精度/bit 压缩率/倍 准确率/% 70 0.91 剪枝+无模拟学习 32 3.6 95.48 剪枝+量化+无模拟学习 8 14.4 95.45 本研究模型 8 14.4 95.99 80 0.58 剪枝+无模拟学习 32 5.7 94.95 剪枝+量化+无模拟学习 8 22.8 94.92 本研究模型 8 22.8 95.55 90 0.23 剪枝+无模拟学习 32 14.3 93.40 剪枝+量化+无模拟学习 8 57.2 93.53 本研究模型 8 57.2 94.58 -
[1] SHAMSHIRBAND S, ANUAR N B, KIAH M L M, et al. Survey an appraisal and design of a multi-agent system based cooperative wireless intrusion detection computational intelligence technique [J]. Eng Appl Artif Intell, 2013, 26(9): 2105 − 2127. [2] HILLNHUTTER C, MAHLEIN A. Early detection and localisation of sugar beet diseases: new approaches [J]. Gesunde Pflanzen, 2008, 60(4): 143 − 149. [3] CAMARGO A, SMITH J S. An image-processing based algorithm to automatically identify plant disease visual symptoms [J]. Biosyst Eng, 2009, 102(1): 9 − 21. [4] MUTKA A M, BART R S. Image-based phenotyping of plant disease symptoms [J]. Front Plant Sci, 2015, 5: 1 − 8. [5] HIARY H A, AHMAD S B, REYALAT M, et al. Fast and accurate setection and classification of plant diseases [J]. Int J Comput Appl, 2011, 17(1): 31 − 38. [6] TIAN Yuan, ZHAO Chunjiang, LU Shenglian, et al. Multiple classifier combination for recognition of wheat leaf diseases [J]. Intell Automation Soft Comput, 2011, 17(5): 519 − 529. [7] 秦丰, 刘东霞, 孙炳达, 等. 基于图像处理技术的4种苜蓿叶部病害的识别[J]. 中国农业大学学报, 2016, 21(10): 65 − 75. QIN Feng, LIU Dongxia, SUN Bingda, et al. Recognition of four different alfalfa leaf diseases based on image processing technology [J]. J China Agric Univ, 2016, 21(10): 65 − 75. [8] BARBEDO J G A. An automatic method to detect and measure leaf disease symptoms using digital Image processing [J]. Plant Dis, 2014, 98(12): 1709 − 1716. [9] BARBEDO J G A. A new automatic method for disease symptom segmentation in digital photographs of plant leaves [J]. Eur J Plant Pathol, 2016, 147(2): 349 − 364. [10] HE Kaiming, ZHANG Xiangyu, REN Shaoqing, et al. Deep residual learning for image recognition [C]// BAJCSY R, LI Feifei, TUYTELAARS T. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas: IEEE Press, 2015: 770−778. [11] MOHANTY S P, HUGHES D P, SALATHÉ M. Using deep learning for image-based plant disease detection [J]. Front Plant Sci, 2016, 7: 1419. [12] KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks [J]. Commun ACM, 2017, 60(6): 84 − 90. [13] SZEGEDY C, LIU Wei, JIA Yangqing, et al. Going deeper with convolutions [C]// BISCHOF H, FORSYTH D, SCHMID C, et al. 2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Boston: IEEE Press, 2015: 1 − 9. [14] 孙俊, 谭文军, 毛罕平, 等. 基于改进卷积神经网络的多种植物叶片病害识别[J]. 农业工程学报, 2017, 33(19): 209 − 215. SUN Jun, TAN Wenjun, MAO Hanping, et al. Recognition of multiple plant leaf diseases based on improved convolutional neural network [J]. Trans Chin Soc Agric Eng, 2017, 33(19): 209 − 215. [15] 龙满生, 欧阳春娟, 刘欢, 等. 基于卷积神经网络与迁移学习的油茶病害图像识别[J]. 农业工程学报, 2018, 34(18): 194 − 201. LONG Mansheng, OUYANG Chunjuan, LIU Huan, et al. Image recognition of Camellia oleifera diseases based on convolutional neural network & transfer learning [J]. Trans Chin Soc Agric Eng, 2018, 34(18): 194 − 201. [16] 张建华, 孔繁涛, 吴建寨, 等. 基于改进VGG卷积神经网络的棉花病害识别模型[J]. 中国农业大学学报, 2018, 23(11): 161 − 171. ZHANG Jianhua, KONG Fantao, WU Jianzhai, et al. Cotton disease identification model based on improved VGG convolution neural network [J]. J China Agric Univ, 2018, 23(11): 161 − 171. [17] DECHANT C, WIESNER-HANKS T, CHEN Siyuan, et al. Automated identification of northern leaf blight-infected maize plants from field imagery using deep learning [J]. Phytopathology, 2017, 107(11): 1426 − 1432. [18] PICON A, ALVAREZ-GILA A, SEITZ M, et al. Deep convolutional neural networks for mobile capture device-based crop disease classification in the wild [J]. Comput Electron Agric, 2019, 161: 280 − 290. [19] WANG Y P E, LIN Xingqin, ADHIKARY A, et al. A primer on 3GPP narrowband internet of things [J]. IEEE Commun Mag, 2017, 55(3): 117 − 123. [20] 陈方. MobileNet压缩模型的研究与优化[D]. 武汉: 华中师范大学, 2018. CHEN Fang. Research and Optimization of MobileNet Compression Model [D]. Wuhan: Central China Normal University, 2018. [21] HAN S, POOL J, TRAN J, et al. Learning both weights and connections for efficient neural networks [C]// CORTES C, LAWRENCE N D, LEE D D, et al. The 28th International Conference on Neural Information Processing Systems. Montreal: MIT Press, 2015: 1135 − 1143. [22] JACOB B, KLIGYS S, CHEN B, et al. Quantization and training of neural networks for efficient integer-arithmetic-only inference [C]// BROWN M, MORSE B, PELEG S, et al. 2018 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Salt Lake City: IEEE Press, 2018: 2704 − 2713. [23] BA J L, CARUANA R. Do deep nets really need to be deep [C]// GHAHRAMANI Z, WELLING M, CORTES C, et al. The 27th International Conference on Neural Information Processing Systems [C]. Montreal: MIT Press, 2014: 2654 − 2662. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20190595







 下载:
下载: