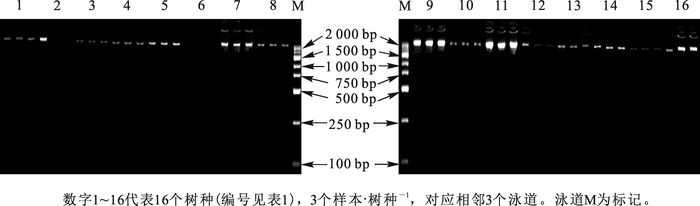
-
樟科Lauraceae植物广泛分布于热带及亚热带地区,全世界约50属2 500~3 000种[1],中国有24属约430种。樟科树木是中国南方重要的经济林木,在林业、轻工、医药、园林绿化中均占有重要地位。楠属Phoebe和樟属Cinnamomum等木材被认为是优质木材,常用作建筑物的梁柱构件、棺椁、家具、箱盒等。樟科木材普遍含有油细胞或黏液细胞,是樟科木材的识别标识;但樟科各个属间及种间,木材结构特征相似,仅通过木材解剖结构特征很难区分。随机扩增多态性(RAPD)与简单序列重复区间扩增多态性(ISSR)是利用聚合酶链式反应(PCR)扩增进行检测的DNA标记[2],具备稳定性好、多态性丰富、成本低、操作简单、样品用量少等优点,因此,利用分子标记技术建立DNA指纹图谱被认为是鉴别树种的有力工具[3]。LANGE等[4]运用RAPD分子标记技术对巨桉Eucalyptu grandis12个无性系进行研究,发现通过其指纹图谱可以有效地鉴定12个巨桉无性系。CASTIGLIONE等[5]利用RAPD分子标记绘制了杨树Populus 32个无性系的指纹图谱,并对它们加以区分。GRAHAM等[6]运用RAPD分子标记技术研究悬钩子属Rubus的13种植物的分类状况。沈永宝等[7]采用ISSR标记技术,仅用2条ISSR引物就将银杏Ginkgo biloba 13个品种进行区分。侯渝嘉等[8]利用ISSR分子标记技术研究了茶树Camellia sinensis种质资源,结果表明:只要用其中任何1条引物即可区分14个茶树品种。李薇等[9]采用ISSR标记技术对6个美洲黑杨Populus deltoids品种进行鉴定,并用1条引物将它们区别。RAPD与ISSR分子标记技术在树种鉴定中被证实具有较强可靠性和稳定性。因此,本研究利用RAPD和ISSR分子标记技术对樟科16个树种进行鉴别,以期为樟科木材精确鉴别提供技术支持。
-
本试验采集南京中山植物园樟科5属16种立木的新鲜枝条,12株·种-1,计192个样品(表 1)。
表 1 实验用材信息
Table 1. Information of tested materials
编号 属名 种名 1 楠属Phoebe 浙江楠P.chekiangensis 2 楠属Phoebe 紫楠P.sheareri 3 楠属Phoebe 丰贞楠P.zhennan 4 楠属Phoebe 闽楠P.bournei 5 楠属Phoebe 湘楠P.hunanensis 6 润楠属Machilus 滇润楠M.pingii 7 润楠属Machilus 龙眼润楠M.oculodracontis 8 润楠属Machilus 薄叶润楠M.leptophylla 9 山胡椒属Lindera 山胡椒L.glauca 10 山胡椒属Lindera 狭叶山胡椒L.angustifolia 11 山胡椒属Lindera 黑壳楠L.megaphylla 12 山胡椒属Lindera 江浙钓樟L.chienii 13 樟属Cinnamomum 浙江樟C.chekiangense 14 樟属Cinnamomum 樟树C.camphora 15 樟属Cinnamomum 大叶樟C.septentrionale 16 月桂属Laurus 月桂L.nobilis -
将去除树皮的枝条切成碎木屑,液氮研磨成粉,准确称量0.1 g木粉,采用改良十六烷基三甲基溴化铵-十二烷基硫酸钠(CTAB-SDS)法[10]提取基因组DNA;紫外分光光度计及质量分数为2%琼脂糖凝胶检测模板DNA的浓度和纯度,置于-20 ℃冰箱中备用。
-
按照文献[11-15]选出ISSR引物42条(表 2),RAPD引物45条(表 3),对提取到的模板DNA进行PCR扩增。RAPD扩增反应体系:10×Taq缓冲液(含Mg2+)2.0 μL;10 mmol·L-1 dNTP Mixture 0.6 μL;10 μmol·L-1 RAPD引物3.0 μL;5×16.67 mkat·L-1 Taq DNA聚合酶0.2 μL;DNA模板2.0 μL;无菌水12.2 μL。PCR程序:94 ℃预变性5 min;94 ℃变性1 min,退火50 s,72 ℃延伸1 min,循环35次;72 ℃保温10 min。设定梯度退火温度34 ℃,36 ℃,38 ℃和40 ℃。ISSR扩增反应体系:10×Taq缓冲液(含Mg2+)2.0 μL;10 mmol·L-1 dNTP Mixture 0.8 μL;10 μmol·L-1 ISSR引物3.0 μL;5×16.67 mkat·L-1 Taq DNA聚合酶0.2 μL;DNA模板2.0 μL;无菌水12.0 μL。PCR程序:94 ℃预变性5 min;94 ℃变性40 s,退火40 s,72 ℃延伸2 min,循环35次;72 ℃保温10 min。设定梯度退火温度50 ℃,52 ℃,54 ℃和56 ℃。
表 2 ISSR引物及其序列
Table 2. ISSR primers and sequences
编号 引物序列3´-5´ 811 GAGAGAGAGAGAGAGAC 812 GAGAGAGAGAGAGAGAA 815 CTCTCTCTCTCTCTCTG 818 CACACACACACACACAG 840 GAGAGAGAGAGAGAGAYT 841 GAGAGAGAGAGAGAGAC 842 GAGAGAGAGAGAGAGAYG 853 TCTCTCTCTCTCTCTCRT 854 TCTCTCTCTCTCTCTCRG 807 AGAGAGAGAGAGAGAGT 856 ACACACACACACACACYA 849 GTGTGTGTGTGTGTGTYA 881 GGGTGGGGTGGGGTG 835 AGAGAGAGAGAGAGAGYC 895 AGAGTTGGTAGCTCTTGATC 837 TATATATATATATATART 844 CTCTCTCTCTCTCTCTRC 836 AGAGAGAGAGAGAGAGYA 838 TATATATATATATATARC 858 TGTGTGTGTGTGTGTGRT 859 TGTGTGTGTGTGTGTGRC 871 TATTATTATTATTATTAT 872 GATAGATAGATAGATA 855 ACACACACACACACACYT 857 ACACACACACACACACYG 843 CTCTCTCTCTCTCTCTRA 830 TGTGTGTGTGTGTGTGG 874 CCCTCCCTCCCTCCCT 823 TCTCTCTCTCTCTCTCC 825 ACACACACACACACACT 808 TATATATATATATATAG 826 ACACACACACACACACC 827 ACACACACACACACACG 828 TGTGTGTGTGTGTGTGA 866 CTCCTCCTCCTCCTCCTC 891 HVHTGTGTGTGTGTGTG 860 TGTGTGTGTGTGTGTGRA 878 GGATGGATGGATGGAT 848 CACACACACACACACARG 873 GACAGACAGACAGACA 834 AGAGAGAGAGAGAGAGYT 880 GGAGAGGAGAGGAGA 表 3 RAPD引物及其序列
Table 3. RAPD primers and sequences
编号 引物序列3´-5´ S2 TCGGCAGCTG S8 GTGACGTAGG S11 CAATCGCCAT S12 TCGGCGATAG S17 GACCGCTTGT S22 TGCCGAGCTG S35 TTCCGAACCC S44 GTGACATGCC S117 GGTGACGCG S150 CACCAGGTGA S397 AGCCTGAGCC S377 CCCAGCTGTG S187 TCCGATGCTG S216 TGGGCGTCAA S226 ACGCCCAGGT S43 GTCGCCGTCA S45 TGAGCGGACA S60 ACCCGGTCAC S66 GAACGGACTC S68 TGGACCGGTG S85 CTGAGACGGA S86 GTGCCTAACC S91 TGCCCGTCGT S126 GGGAATTCGG S130 GGAAGCTTGG S222 AGTCACTCCC S237 ACCGGCTTGT S133 GGCTGCAGAA S247 CCTGCTCATC S285 GGCTGCGACA S446 CCACGGGAAG S461 GTAGCACTCC S3 CATCCCCCTG S21 CAGGCCCTG S24 AATCGGGCTG S28 GTGACGTAGG S31 CAATCGCCGT S40 GTTGCGATCC S62 GTGAGGCGTC S97 ACGACCGACA S221 TGACGCATGG S380 GTGTCGCGAG S146 AAGACCCCTC S167 CAGCGACAAG S184 CACCCCCTTG -
取扩增产物5.0 μL上样到质量分数为2%琼脂糖凝胶中电泳,电泳缓冲液为0.5×TBE,电压160 V,电泳40 min。电泳完毕后放入凝胶成像系统中观察并拍照。
-
分析电泳图,读带时只记录清晰可辨的条带,排除模糊不清的条带。以16个树种为单元,统计同一树种12株同一位置上出现的共有条带,忽略同一树种不同样本间的差异条带,统计多态性位点,计算多态性比率,多态性比率(%)=(总条带数-共有条带数)/总条带数×100%。
-
16个树种均取3个样本的基因组DNA进行电泳检测,发现所有条带清晰无弥散,点样孔清晰(图 1),说明基因组DNA完整性好;紫外分光光度计检测,DNA样品D(260/280)值为1.7~2.0,说明DNA样品纯度较好,满足本试验的要求。
-
RAPD-PCR扩增筛选出8条能扩增出清晰条带且多态性好的引物,并测得其最佳退火温度均是38 ℃(表 4)。共获得清晰可辨条带165条,其中多态性条带153条,多态性比率为92.7%,扩增的DNA片段长度集中在100 ~2 000 bp。平均每条引物扩增出20.6条带,其中19.1条具多态性。引物S91多态性最高,达到100%,引物S221最低,为83.3%。不同引物扩增的产物在多态性水平上的较大差异,表明参试的16个树种在分子水平上差异很大,具有丰富的遗传多样性。
表 4 8条RAPD引物扩增结果
Table 4. Amplified results of 8 RAPD primers
引物序号 退火温度/
℃总条带数/
条多态性条
带/条多态性比
率/ %S22 38 23 21 91.3 S91 38 23 23 100 S35 38 22 21 95.5 S150 38 20 18 90.0 S221 38 18 15 83.3 S66 38 22 21 95.5 S85 38 19 18 94.7 S97 38 18 16 88.9 总计 165 153 平均 20.6 19.1 92.7 扩增结果表明:每条RAPD引物单独使用均可区别这16个树种。以引物S35为例,该引物共扩增出21条多态性条带(图 2),其中楠属扩增出4条共有条带,樟属扩增出5条共有条带。楠属树种扩增出特异性条带450 bp和750 bp,山胡椒属特异性条带为310 bp,樟属特异性条带为2 000 bp和1 000 bp;这些条带可将樟科5属区别开。同属树种间多态性条带差异较小,楠属树种浙江楠(编号1)特异性条带为1 500 bp,紫楠(编号2)特异性条带为1 800 bp,桢楠(编号3)特异性条带为600 bp,湘楠(编号5)特异性条带为2 000 bp。由此可区分楠属5个树种。润楠属中薄叶润楠(编号8)特异性条带为170 bp,龙眼润楠(编号7)特异性条带为1 000 bp;山胡椒属山胡椒(编号9)特异性条带为300 bp,狭叶山胡椒(编号10)特异性条带为1 000 bp,黑壳楠(编号11)特异性条带为2 000 bp;樟属大叶樟(编号15)特异性条带为320 bp,浙江樟(编号13)特异性条带为1 000 bp。由此可知,RAPD引物S35可将16个树种有效区分。
-
ISSR-PCR扩增筛选出6条能够扩增出清晰条带且多态性好的引物,并测得其最佳退火温度(表 5)。共获得清晰可辨条带96条,其中多态性条带86条,平均多态性比率为89.6%,扩增的DNA片段长度集中在100~2 000 bp。平均扩增出条带16条·引物-1,其中14.3条具多态性,引物843多态性最高,为94.4%,引物825多态性最低,为80.0%。
表 5 6条ISSR引物扩增结果
Table 5. Amplified results of 6 ISSR primers
引物序号 退火温度/
℃总条带数 多态性条
带多态性比
率/%815 56 13 12 92.3 825 54 15 12 80.0 834 54 18 16 88.9 840 54 17 16 94.1 848 56 15 13 86.7 843 54 18 17 94.4 总计 96 86 平均 16 14.3 89.6 以引物840扩增结果(图 3)为例,共扩增出16条多态性条带,其中山胡椒属特异性条带在600 bp和750 bp位置,润楠属特异性条带在250 bp和700 bp位置,樟属特异性条带为300 bp和700 bp,月桂属在100 bp处扩增出特异性条带。根据这些特异性条带可区分樟科5属。同属树种间多态性条带差异较小,楠属树种浙江楠(编号1)的特异性条带在1 000 bp位置,紫楠(编号2)特异性条带为450 bp和750 bp,闽楠(编号4)特异性条带为350 bp和400 bp的片段,湘楠(编号5)特异性条带为350 bp和750 bp位置。由此将楠属树种区分开。引物840在山胡椒属的黑壳楠(编号11)和江浙钓樟(编号12)中扩增出相同的条带,不能有效区分这2个树种。
除引物840外,引物815,825,848和843扩增的多态性条带都可区分16个树种。引物840扩增的多态性条带不能有效区分黑壳楠和江浙钓樟,引物834不能有效区分山胡椒和狭叶山胡椒,但这2个引物的组合可区分16个树种。
-
ISSR和RAPD分子标记具备良好的多态性,因而在树种鉴定和指纹图谱研究方面得到了广泛应用。本试验利用8条RAPD引物和6条ISSR引物对樟科16个树种进行DNA扩增,发现2种标记均能扩增出稳定、清晰且多态性好的条带。RAPD分子标记扩增的多态性比率为92.7%,平均每条引物扩增的多态性条带有19.1条;ISSR分子标记扩增的多态性比率为89.6%,平均每条引物扩增的多态性条带有14.3条,通过1个RAPD引物或1~2个ISSR引物扩增的多态性条带可鉴别樟科16个树种。对比结果也发现,RAPD分子标记的多态性和稳定性比ISSR高,与李元春等[16]在山核桃Carya cathayensis中发现RAPD标记的多态性要高于ISSR标记的结果一致;而在马尾松Pinus massoniana[17]和银杏[18]等的研究却发现ISSR标记的多态性要高于RAPD标记,说明不同树种适合不同的分子标记。考虑到2种分子标记的检测水平不同,产生的高效的多态性条带也不同,因此,将这2种标记结合使用会使树种鉴别结果更加准确和全面。
用RAPD分子标记和ISSR分子标记技术鉴定木材,结果准确、耗材少、不受时间和环境的限制,拓宽了木材识别的范围,提高了木材识别的精确性;采用琼脂糖凝胶电泳进行分子鉴定节约了实验经济成本,这不仅在进出口树种鉴定中意义重大,在巨大的木制品消费市场及相关其他领域也具有广泛的应用前景和开发利用价值。
Establishment of RAPD and ISSR markers for wood identification of 16 species of Lauraceae
-
摘要: 采用随机扩增多态性(RAPD)和简单序列重复区间扩增多态性(ISSR)技术鉴别樟科Lauraceae16个树种。用改良十六烷基三甲基溴化铵-十二烷基硫酸钠(CTAB-SDS)法提取樟科16个树种192株(12株·种-1)木质部基因组DNA,用RAPD与ISSR技术对它们进行聚合酶链式反应(PCR)扩增。共筛选出8条RAPD引物与6条ISSR引物,8条RAPD引物共扩增出165条大小为100~2 000 bp的条带,其中多态性条带为153条,多态率为92.7%;6条ISSR引物共扩增出96条大小为100~2 000 bp的条带,其中多态性条带为86条,多态率为89.6%。通过1~2个引物扩增的多态性条带就可鉴别与区分参试的16个树种,为树种鉴定提供技术支持。Abstract: Random amplified polymorphic DNA (RAPD) and inter simple sequence repeats (ISSR) techniques were applied to identify 16 species of Lauraceae. The optimized cetyl trimethylammonium bromide (CTAB)-sodium dodecyl sulfate (SDS) method was used to extract wood DNA of 192 trees in the 16 species. RAPD and ISSR techniques were used to amplify DNA of the 16 species. Results showed that eight RAPD primers and six ISSR primers were screened. Through the eight RAPD primers, 165 bands were obtained, among which 153 were polymorphic for a species level polymorphism loci rate of 92.7%. 96 bands were obtained through the six ISSR primers, among which 86 were polymorphic for a species level polymorphism loci rate of 89.6%. DNA fragment size was 100-2 000 bp. These polymorphism bands amplified by 1 or 2 primers could distinguish the wood of these 16 species in the Lauraceae family, providing technical support for wood identification.
-
表 1 实验用材信息
Table 1. Information of tested materials
编号 属名 种名 1 楠属Phoebe 浙江楠P.chekiangensis 2 楠属Phoebe 紫楠P.sheareri 3 楠属Phoebe 丰贞楠P.zhennan 4 楠属Phoebe 闽楠P.bournei 5 楠属Phoebe 湘楠P.hunanensis 6 润楠属Machilus 滇润楠M.pingii 7 润楠属Machilus 龙眼润楠M.oculodracontis 8 润楠属Machilus 薄叶润楠M.leptophylla 9 山胡椒属Lindera 山胡椒L.glauca 10 山胡椒属Lindera 狭叶山胡椒L.angustifolia 11 山胡椒属Lindera 黑壳楠L.megaphylla 12 山胡椒属Lindera 江浙钓樟L.chienii 13 樟属Cinnamomum 浙江樟C.chekiangense 14 樟属Cinnamomum 樟树C.camphora 15 樟属Cinnamomum 大叶樟C.septentrionale 16 月桂属Laurus 月桂L.nobilis 表 2 ISSR引物及其序列
Table 2. ISSR primers and sequences
编号 引物序列3´-5´ 811 GAGAGAGAGAGAGAGAC 812 GAGAGAGAGAGAGAGAA 815 CTCTCTCTCTCTCTCTG 818 CACACACACACACACAG 840 GAGAGAGAGAGAGAGAYT 841 GAGAGAGAGAGAGAGAC 842 GAGAGAGAGAGAGAGAYG 853 TCTCTCTCTCTCTCTCRT 854 TCTCTCTCTCTCTCTCRG 807 AGAGAGAGAGAGAGAGT 856 ACACACACACACACACYA 849 GTGTGTGTGTGTGTGTYA 881 GGGTGGGGTGGGGTG 835 AGAGAGAGAGAGAGAGYC 895 AGAGTTGGTAGCTCTTGATC 837 TATATATATATATATART 844 CTCTCTCTCTCTCTCTRC 836 AGAGAGAGAGAGAGAGYA 838 TATATATATATATATARC 858 TGTGTGTGTGTGTGTGRT 859 TGTGTGTGTGTGTGTGRC 871 TATTATTATTATTATTAT 872 GATAGATAGATAGATA 855 ACACACACACACACACYT 857 ACACACACACACACACYG 843 CTCTCTCTCTCTCTCTRA 830 TGTGTGTGTGTGTGTGG 874 CCCTCCCTCCCTCCCT 823 TCTCTCTCTCTCTCTCC 825 ACACACACACACACACT 808 TATATATATATATATAG 826 ACACACACACACACACC 827 ACACACACACACACACG 828 TGTGTGTGTGTGTGTGA 866 CTCCTCCTCCTCCTCCTC 891 HVHTGTGTGTGTGTGTG 860 TGTGTGTGTGTGTGTGRA 878 GGATGGATGGATGGAT 848 CACACACACACACACARG 873 GACAGACAGACAGACA 834 AGAGAGAGAGAGAGAGYT 880 GGAGAGGAGAGGAGA 表 3 RAPD引物及其序列
Table 3. RAPD primers and sequences
编号 引物序列3´-5´ S2 TCGGCAGCTG S8 GTGACGTAGG S11 CAATCGCCAT S12 TCGGCGATAG S17 GACCGCTTGT S22 TGCCGAGCTG S35 TTCCGAACCC S44 GTGACATGCC S117 GGTGACGCG S150 CACCAGGTGA S397 AGCCTGAGCC S377 CCCAGCTGTG S187 TCCGATGCTG S216 TGGGCGTCAA S226 ACGCCCAGGT S43 GTCGCCGTCA S45 TGAGCGGACA S60 ACCCGGTCAC S66 GAACGGACTC S68 TGGACCGGTG S85 CTGAGACGGA S86 GTGCCTAACC S91 TGCCCGTCGT S126 GGGAATTCGG S130 GGAAGCTTGG S222 AGTCACTCCC S237 ACCGGCTTGT S133 GGCTGCAGAA S247 CCTGCTCATC S285 GGCTGCGACA S446 CCACGGGAAG S461 GTAGCACTCC S3 CATCCCCCTG S21 CAGGCCCTG S24 AATCGGGCTG S28 GTGACGTAGG S31 CAATCGCCGT S40 GTTGCGATCC S62 GTGAGGCGTC S97 ACGACCGACA S221 TGACGCATGG S380 GTGTCGCGAG S146 AAGACCCCTC S167 CAGCGACAAG S184 CACCCCCTTG 表 4 8条RAPD引物扩增结果
Table 4. Amplified results of 8 RAPD primers
引物序号 退火温度/
℃总条带数/
条多态性条
带/条多态性比
率/ %S22 38 23 21 91.3 S91 38 23 23 100 S35 38 22 21 95.5 S150 38 20 18 90.0 S221 38 18 15 83.3 S66 38 22 21 95.5 S85 38 19 18 94.7 S97 38 18 16 88.9 总计 165 153 平均 20.6 19.1 92.7 表 5 6条ISSR引物扩增结果
Table 5. Amplified results of 6 ISSR primers
引物序号 退火温度/
℃总条带数 多态性条
带多态性比
率/%815 56 13 12 92.3 825 54 15 12 80.0 834 54 18 16 88.9 840 54 17 16 94.1 848 56 15 13 86.7 843 54 18 17 94.4 总计 96 86 平均 16 14.3 89.6 -
[1] 李捷, 李锡文.世界樟科植物系统学研究进展[J].云南植物研究, 2004, 26(1):1-11. LI Jie, LI Xiwen. Advances in Lauraceae systematic research on the world scale [J]. Acta Bot Yunnan, 2004, 26(1): 1-11. [2] ZIETICWIEZ E, RAFALSKI A, LABUDA D. Genome fingerprinting by simple sequence repeats (SSR). anchored polymerase chain reaction amplification [J]. Genomics, 1994, 20(2): 176-183. [3] CEATANO-ANOLLÉS G, GRESSHOFF P M. DNA Markers: Protecols, Applications, and Overriews [M]. NewYork: Wiley-VCH Press, 1997. [4] de LANGE W J, WINGFIELD B D, VILJOEN C D, et al. RAPD fingerprinting to identify Eucalyptus grandis clones [J]. South Afr For J, 1993, 167(1): 47-50. [5] CASTIGLIONE S, WANG G, DAMIANI G, et al. RAPD fingerprints for identification and for taxonomic studies of elite poplar (Populus spp.) clones [J]. Theor Appl Genet, 1993, 87(1/2): 54-59. [6] GRAHAM J, McNICOL R J. An examination of the ability of RAPD markers to determine the relationship within and between Rubus species [J]. Theor Appl Genet, 1995, 90(7/8): 1128-1132. [7] 沈永宝, 施季森, 赵洪亮.利用ISSR DNA标记鉴定主要银杏栽培品种[J].林业科学, 2005, 41(1):202-204. SHEN Yongbao, SHI Jisen, ZHAO Hongliang. Identification on the main cultivated varieties of Ginkgo biloba using ISSR DNA marker [J]. Sci Silv Sin, 2005, 41(1): 202-204. [8] 侯渝嘉, 何桥, 李中林, 等.应用ISSR分子标记对茶树种质资源进行分子鉴定[J].西南农业学报, 2007, 20(6):1272-1276. HOU Yujia, HE Qiao, LI Zhonglin, et al. ISSR applied to the germplasms identification of Camellia sinensis [J]. Southwest China J Agric Sci, 2007, 20(6): 1272-1276. [9] 李薇, 王永进, 樊军锋.美洲黑杨新无性系的ISSR鉴别及SCAR标记转化[J].西北林学院学报, 2014, 29(4):98-102. LI Wei, WANG Yongjin, FAN Junfeng. ISSR identification and transformation of SCAR marker of three new clones of Populus deltoids [J]. J Northwest For Univ, 2014, 29(4): 98-102. [10] 刘金良, 伏建国, 杨晓军, 等.进口针叶木木材的DNA提取与分子鉴定方法[J].中南林业科技大学学报, 2012, 32(8):131-136. LIU Jinliang, FU Jianguo, YANG Xiaojun, et al. DNA extraction from imported coniferous tree wood and identification technology based on molecular genetic tools [J]. J Cent South Univ For Technol, 2012, 32(8): 131-136. [11] 刘玉香, 宋晓琛, 江香梅.润楠ISSR-PCR优化反应体系建立及引物筛选[J].林业科技开发, 2013, 27(5):24-28. LIU Yuxiang, SONG Xiaochen, JIANG Xiangmei. Optimization of ISSR-PCR reaction system and primers screening in Machilus pingii [J]. China For Sci Technol, 2013, 27(5): 24-28. [12] 邢建宏, 陈存及, 张国防, 等.樟树ISSR-PCR反应体系优化研究[J].福建林业科技, 2006, 33(3):96-100. XING Jianhong, CHEN Cunji, ZHANG Guofang, et al. Study on the establishment of ISSR-PCR reaction conditions in Cinnamomum camphora [J]. J Fujian For Sci Technol, 2006, 33(3): 96-103. [13] 陈俊秋, 慈秀芹, 李巧明, 等.樟科濒危植物思茅木姜子遗传多样性的ISSR分析[J].生物多样性, 2006, 14(5):410-418. CHEN Junqiu, CI Xiuqin, LI Qiaoming, et al. Genetic diversity of Litsea szemaois, an endangered species endemic to China, detected by inter-simple sequence repeat (ISSR) [J]. Biodiversity Sci, 2006, 14(5): 410-418. [14] 张炜, 龙汉利, 贾廷彬, 等.桢楠DNA提取和RAPD条件的优化[J].四川林业科技, 2011, 32(4):55-62. ZHANG Wei, LONG Hanli, JIA Tinbin, et al. DNA extraction and optimization of RAPD reaction system for Phoebe zhennan [J]. J Sichuan For Sci Technol, 2011, 32(4): 55-62. [15] 张国防, 陈存及, 邢建宏.樟树RAPD反应体系的优化[J].江西农业大学学报, 2006, 28(3):373-377. ZHANG Guofang, CHEN Cunji, XING Jianhong. Optimalization of RAPD reaction system in Cinnamomum camphora [J]. Acta Agric Univ Jiangxi, 2006, 28(3): 373-377. [16] 李元春, 沈林, 曾燕如.山核桃SRAP体系的建立及与RAPD和ISSR标记的比较[J].浙江农林大学学报, 2011, 28(3):505-512. LI Yuanchun, SHEN Lin, ZENG Yanru. Establishment of a SRAP analysis protocol in Carya cathayensis and a comparison among SRAP, RAPD, ISSR analysis protocols [J]. J Zhejiang A & F Univ, 2011, 28(3): 505-512. [17] 刘爱萍. 中国12个省(区)马尾松主要种质资源的RAPD与ISSR分析[D]. 福州: 福建农林大学, 2007. LIU Aiping. RAPD and ISSR Analysis of Main Pinus massoniana Genetic Resources in 12 Provinces of China [D]. Fuzhou: Fujian Agriculture and Forestry University, 2007. [18] 花喆斌. 银杏群体遗传多样性的RAPD和ISSR分析[D]. 南京: 南京林业大学, 2007. HUA Zhebin. Genetic Diversity in Populations of Ginkgo biloba L. Detected by RAPD and ISSR [D]. Nanjing: Nanjing Forestry University, 2007. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2017.05.023







 下载:
下载: