-
桂花Osmanthus fragrans在中国栽培历史悠久,栽培区域广阔,是中国最重要的集绿化、美化和香化为一体的传统名花和园林树种之一[1-2]。桂花野生资源丰富,现已明确的有150多个品种[3-4],这为桂花种质创新和新品种的选育提供了丰富的遗传变异和优异的基因库[5]。分子标记是进行品种鉴定和亲缘关系分析、分子辅助育种、遗传多样性和繁育系统研究等最常用的技术手段,通常需要有共显性、多态性高、重复性好的引物,因此引物开发显得极其重要[6]。简单重复序列(simple sequence repeats, SSR)是由几个核苷酸为重复单位组成的长达几十个核苷酸的串联重复序列[7],目前,已经报道的桂花SSR引物十分有限,仅ZHANG等[8]开发了29对桂花SSR引物,马寅峰[9]经过筛选获得20对具有多态性的SSR引物,急需开发更多的特异性和多态性高的SSR引物用于桂花遗传分析和品种鉴定等相关研究。表达序列标签-简单重复序列(expressed sequence tag SSR,EST-SSR)技术可避免基因组SSR开发过程中需要构建基因组DNA文库等繁琐步骤,可为功能基因提供可靠的标记,能够充分反映基因组功能区域的相似程度。目前,EST-SSR标记也成为了遗传多样性和品种亲缘关系研究的重要手段[10]。最优的SSR-PCR反应体系和条件,对样本DNA所扩增条带的数量、清晰度以及稳定性甚为重要,进而影响条带多态性的统计与分析[11]。转录组数据是EST-SSR开发利用的有效资源,本研究首先对桂花SSR反应体系进行了优化,同时以不同野生桂花种群为材料,利用桂花转录组数据库大量筛选EST-SSR引物,开发多态性高的EST-SSR引物,并利用筛选得到的引物进行品种鉴定,促进EST-SSR标记技术在桂花种质资源评价和保护、品种鉴定和遗传多样性分析等方面的研究。
-
样本包括浙江龙泉、浙江千岛湖、福建长汀、福建清流、湖南郴州、湖南株洲、贵州瑞安、江西九江等8个桂花野生种群,浙江农林大学桂花资源圃地的7个丹桂品种,以及采自四川农科院园艺所的待鉴定品种。所有样本材料均采集桂花新生枝上第4片完全展开叶,硅胶干燥后,放置-20 ℃冰箱备用。
SSR-PCR反应用的Taq酶,dNTP,镁离子(Mg2+),PCR缓冲液(buffer),电泳用的10×TBE缓冲液,50×TAE溶液的相关药品,无水乙醇,冰乙酸,琼脂糖,丙烯酰胺,甲叉丙烯酰胺,溴酚蓝,6 ×上样缓冲液(loading buffer),标准分子量DNA标准物(marker)等均购自上海生工生物工程公司。
-
本研究采用改良的十六烷基三甲基溴化铵(CTAB)法提取桂花DNA[12],用Nano-Drop1000全自动核酸蛋白快速测仪和质量浓度为1.2%琼脂糖凝胶电泳以及紫外凝胶成像系统检测DNA纯度、浓度和质量。从8个桂花野生种群中随机各选取3份样本DNA作为SSR-PCR反应的模板DNA,选择福建官坊居群中的E4模板DNA用于体系优化以减少模板变化影响体系优化的效果。将所有模板DNA稀释到试验所需的100 mg·L-1,-20 ℃保存备用。
-
以桂花E4为DNA模板,在20 μL的PCR体系中,除2 μL 10 × PCR缓冲液外,其他因素采用L16(45)正交试验设计,对模板DNA含量、Mg2+浓度、dNTP浓度、引物浓度、Taq DNA聚合酶单位数进行5因素4水平梯度试验,共16个正交设计组合(表 1),以确定最佳的桂花EST-SSR反应体系[13]。
表 1 EST-SSR正交试验设计L16(45)
Table 1. EST-SSR orthogonal experimental design L16(45)
组合编号 DNA/ng Mg2+/(mmol·L-1) dNTPs/(mmol·L-1) Taq酶/(×16.67 nkat) 引物/(μmol·L-1) 1 40 1.125 0.15 0.50 0.15 2 40 1.500 0.20 0.75 0.20 3 40 1.875 0.25 1.00 0.25 4 40 2.250 0.30 1.25 0.30 5 60 1.875 0.20 1.25 0.20 6 60 2.250 0.15 1.00 0.15 7 60 1.125 0.30 0.75 0.30 8 60 1.500 0.25 0.50 0.25 9 80 2.250 0.25 0.75 0.25 10 80 1.875 0.30 0.50 0.30 11 80 1.500 0.15 1.25 0.15 12 80 1.125 0.20 1.00 0.20 13 100 1.500 0.30 1.00 0.30 14 100 1.125 0.25 1.25 0.25 15 100 2.250 0.20 0.50 0.20 16 100 1.875 0.15 0.75 0.15 -
利用Biometra PCR仪(德国)进行PCR扩增反应,反应程序如下:94 ℃预变性4 min;94 ℃变性30 s,不同退火时间(TM)为30 s,72 ℃延伸30 s,循环数35;72 ℃延伸7 min。扩增产物利用体积分数为8%非变性聚丙烯酰胺凝胶[28 mL双蒸水,8 mL体积分数为40%丙烯酰胺,4 mL 10 × TBE,320 μL体积分数为10%过硫酸铵,40 μL四甲基乙二胺(TEMED)]电泳检测,以DNA marker B作为参照;电泳仪(北京市六一仪器厂,DYCZ-6C型)电压160 V,电流200 mA,电泳100 min,然后用银染法显示扩增条带。小心拆取凝胶后,依次浸入到固定液及染色液中(20 mL体积分数为10%乙醇,体积分数为0.5%冰乙酸1 mL,硝酸银0.2 g,蒸馏水180 mL),置于摇床上震荡染色12~15 min;去离子水漂洗30 s;显色液(2 g氢氧化钠,0.04 g四硼酸钠、0.6 mL甲醛、200 mL蒸馏水)充分震荡12~15 min,直至条带完全显色。利用凝胶成像系统拍照记录结果,统计并分析扩增条带的多态性。
-
从桂花转录组EST-SSR引物序列数据库中随机挑选150对引物,由生工生物工程(上海)股份有限公司合成,其详细信息参见表 2。利用优化后的最佳反应体系和条件,以8份桂花野生种群的基因组DNA作为模板,对150对SSR引物进行初次筛选,12 g·L-1的琼脂糖凝胶电泳进行扩增产物的有效性检测;再以24份基因组DNA(3份·种群-1)对初筛获得的引物进行复筛,80 g·L-1非变性聚丙烯酰胺凝胶电泳进行多态性检测,最终获得多态性高、重复性好、稳定性高的SSR引物。
表 2 150对随机引物信息表
Table 2. Random primers information of 150 pairs
重复次数 重复类型 总数 5 6 7 8 9 10 11 12 >12 单核苷酸 1 1 2 7 12 二核苷酸 1 1 1 1 3 3 1 10 21 三核苷酸 10 5 2 5 4 2 30 四核苷酸 4 3 1 1 10 五核苷酸 9 1 12 六核苷酸 18 7 7 2 1 36 复合重复 29 总计 41 17 11 9 6 6 4 3 17 150 -
随机挑选3对具有显著多态性的SSR引物,以浙江农林大学桂花苗圃的7个丹桂品种为参照,对四川省常见栽培的14份丹桂样本(SC01~SC14)进行品种鉴定(表 3)。
表 3 品种鉴定材料信息表
Table 3. Material information for cultivars
名称 样本采集地 SC01 成都市街区 SC02 成都市邛崃市 SC03 成都市崇州市区 SC04 成都市大邑县 SC05 成都市都江堰市 SC06 成都市彭州市 SC07 成都市都江堰市 SC08 成都市新都区 SC09 成都市新都区 SC10 成都市新都区 SC11 成都市郫县 SC12 成都市郫县 SC13 成都市温江区 SC14 成都市温江区 ‘朱砂丹桂’ ‘Zhusha Dangui’ 浙江农林大学桂花苗圃 ‘橙红丹桂’ ‘ Chenghong Dangui ’ 浙江农林大学桂花苗圃 ‘状兀红’ ‘ Zhuangyuan Hong ’ 浙江农林大学桂花苗圃 ‘武夷丹桂’ ‘Wuyi Dangui’ 浙江农林大学桂花苗圃 ‘堰虹桂’ ‘ Yanhong Gui ’ 浙江农林大学桂花苗圃 ‘满条红’ ‘ Mantiao Hong ’ 浙江农林大学桂花苗圃 ‘上海丹桂’ ‘ Shanghai Dangui ’ 浙江农林大学桂花苗圃 -
根据条带迁移情况统计目标范围内出现的条带,不同大小的条带分别用“A,B,C,D…”进行分类标记,纯合子带型记为“AA,BB,CC,DD,…”,杂合子标记为“AB,AC,BD,AD,…”,将带型数据输入POPGENE 1.32软件计算遗传距离,然后利用SPSS 19.0的欧氏平均联接法绘制聚类树状图。
-
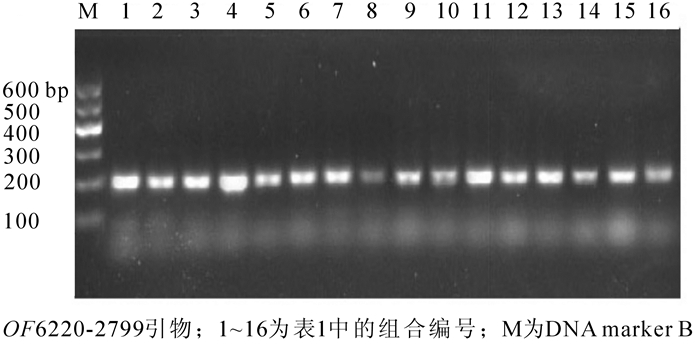
以OF6220-2799扩增结果为例,在SSR-PCR产物琼脂糖凝胶电泳检测结果中,组合8,14,16的扩增条带较弱,其余组合都能扩增出较为清晰的条带,其中组合4的条带最为明亮(图 1)。参照正交试验各试剂的浓度和用量,最终确定桂花EST-SSR最优PCR反应体系为:DNA 40 ng,Mg2+ 2.25 mmol·L-1,引物0.3 μmol·L-1,dNTPs 0.3 mmol·L-1,TaqDNA聚合酶1.25 × 16.67 nkat,并加入2 μL 10 × PCR缓冲液,双蒸水(ddH2O)补充至20 μL。
-
利用优化后的PCR反应体系和条件,对随机选取的150对EST-SSR引物进行初步筛选,共有79对引物扩增出特异性产物,有效扩增率为52.67%,在对应的SSR中,最多的为三、六核苷酸和复合重复类型,最少的为单核苷酸和四核苷酸重复类型(表 4)。
表 4 150对随机EST-SSR引物扩增情况
Table 4. EST-SSR amplification of 150 primers
重复类型 总数 成功扩增/对 比例/% 多态引物/对 多态性比例/% 单核苷酸 12 5 41.67 0 0 二核苷酸 21 7 33.33 0 0 三核苷酸 30 20 66.67 5 16.67 四核苷酸 10 5 50.00 1 10.00 五核苷酸 12 6 50.00 3 25.00 六核苷酸 36 17 47.22 7 19.44 复合重复 29 19 65.52 3 10.34 总体 150 79 52.67 19 12.67 以8个不同野生种群的24份桂花基因组DNA为模板,对初筛所获得的79对引物进行复筛。最终筛选出19对稳定性好、多态性高的引物,占引物总数的12.67%,其中,五核苷酸重复对应的引物多态性最高(25.00%),其次分别为六核苷酸(19.44%)和三核苷酸(16.67%),在单核苷酸和二核苷酸重复类型中没有发现多态性引物(表 4)。筛选得到的19对引物中除了OF8091-2000,OF8233-681,OF10275-1164,OF21405-424在种群内扩增的多态性条带不明显外,其余的15对引物在种群内均能够扩增出丰富的多态性条带(图 2)。
-
利用筛选到的多态性较高的OF8623-1677,OF24299-844,OF28774-2088 3对引物对供试品种进行鉴定(表 5)。扩增结果显示:每对引物都可以产生1或2个等位基因,并且都可以将所有桂花样本有效区分,所有已知桂花品种均有各自独特的带型或带型组合(表 6)。
表 5 3对桂花EST-SSR引物序列
Table 5. Three EST-SSR primer sequences of Osmanthus fragrance
编号 名称 引物序列 退火温度/℃ 重复单元 预期片段大小/bp 9 OF8623-1677 F-TCCACACGCTGAAACTCCTA
R-TCGTATGATGGAGCAGCAAG58 (AGAAA)5 156 13 OF24299-844 F-TACAAAGAAGCCCCACCAAC
R-ATTGAGAGCAACCATTTGCC57 (CCCAAA)5 240 19 OF28774-2088 F-TGACACCATGACTTCCCAGA
R-CAATTGTCGGTGTTGGTTTG57 (TTAGCC)5 174 表 6 品种鉴定带型信息统计表
Table 6. Cultivars with type information statistics
样本 OF8623-1677 OF24299-844 OF28774-2088 SC01 DD AA BC SC02 DD AA BC SC03 DD AA BC SC04 DD AA BC SC05 DD AA BC SC06 CC AC CD SC07 DD 99 BC SC08 99 AD BC SC09 DD AF BC SC10 DD AA BC SC11 DD AA BC SC12 DD 99 BC SC13 DD AA BC SC14 DD AA CC ‘朱砂丹桂’ DD AA BC ‘橙红丹桂’ 99 AE DD ‘状元红’ BB AF CC ‘武夷丹桂’ CC AD CC ‘堰虹桂’ CC AC CD ‘满条红’ BB AF BC ‘上海丹桂’ AA AB AB 说明:相同字母组合代表纯合子,不同字母组合杂合子,99表示无扩增条带 从品种聚类树状图(图 3)可以看出:在相对距离为12的时候,21份供鉴定样本可聚为4类。首先,‘上海丹桂’和‘橙红丹桂’分别单独聚为一类(Ⅰ和Ⅱ),因为这2个品种在3对引物中的带型均比较特别,表现出明显的差异性;第Ⅲ类中,包含了‘朱砂丹桂’和大部分的成都来源的样本,其中除了SC07和SC12在引物OF24299-844中未扩增出条带外,这12份样本在3对引物中均表现出与‘朱砂丹桂’一致的带型,可以判断这些样本为‘朱砂丹桂’;第Ⅳ类中,包含了剩余的4个已知品种和2种待鉴定样本。在相对距离为6时,可将第Ⅳ类分为2组:a组,SC06先与‘堰虹桂’聚为一小支,再与‘武夷丹桂’聚为一支,SC06与‘堰虹桂’在带型上完全一致的,可判断彭州市来源的丹桂为‘堰虹桂’;b组,‘满条红’和‘状元红’先聚为一小支,再与SC08聚为一支,说明SC08与‘满条红’和‘状元红’亲缘关系较近。
-
建立稳定可靠的SSR-PCR反应体系是EST-SSR分子标记开发和利用的基础,SSR体系优化的方法有单因素试验、完全试验或正交试验[14]。单因素试验不能保证各最佳组分的组合即为最佳的反应体系;完全试验却存在试验组合繁多,工作量大,试验周期长等弊端[15-17];正交试验设计则是仅选用几个具有代表性的处理组合进行试验,结果分析简便直观,可操作性强[18]。袁存权等[19]利用L16(45)正交试验设计对刺槐Robinia pseudoacacia的相关序列扩增多态性聚合酶链式反应(SRAP-PCR)体系中的主要成分进行的优化,杨传平等[20]也成功利用L16(45)正交试验设计,优化并建立了一套适用于白桦Betula platyphylla的SSR-PCR反应体系。本研究也利用了正交试验设计,优化并建立了适合桂花的SSR-PCR反应体系。
不同植物的多态性SSR核苷酸重复类型具有很大的差异[21],在蜡梅Chimonanthus praecox中,李响等[22]共从120对引物中筛选获得17对优良的EST-SSR引物,筛出率为14.17%,主要包括二、三和六核苷酸重复类型;徐阳等[23]以杉木Cunninghamia lanceolata为材料,从10个EST-SSR和8个基因组SSR(gSSR)中各筛出4对多态性引物,多态率分别为40.00%和50.00%,所有核苷酸重复类型均有出现。本研究以桂花转录组数据库为基础,从完全随机选取的150对EST-SSR引物中筛选出19对具有稳定多态性的引物,多态性比例为12.67%。其中,五核苷酸(25.00%)、六核苷酸(19.44%)和三核苷酸(16.67%)重复类型的多态性较高,充分说明可以利用桂花的转录组数据库大规模进行桂花EST-SSR引物的开发。
SSR分子标记已经很好地应用于品种鉴定和亲缘关系分析、遗传多样性、交配系统等研究。胡菀等[5]利用SSR分子标记对7个野生桂花群体139个个体的遗传多样性和遗传结构进行了研究,并通过STRUCTURE聚类分析得到不同野生居群间具有较丰富的遗传多样性;ZHANG等[8]开发并获得了29对具有高度多态性的SSR引物,对野生桂花种群和桂花品种分类鉴定进行了相关遗传研究。野生桂花遗传变异丰富,本研究以分布距离相对分散的野生桂花为模板筛选得到的引物,具有很强的研究价值,再利用筛选到的3对高度多态性的EST-SSR引物将遗传差异较小的7个已知丹桂品种进行有效地区分,鉴定了来自于四川成都的14份丹桂样本。结果表明:四川常见的栽培丹桂品种大部分为‘朱砂丹桂’,来源于彭州市丹桂SC06为‘堰虹桂’,而来源于新都区的SC08与‘满条红’和‘状元红’亲缘关系较近。EST-SSR来源于基因的编码区,与功能基因紧密连锁,并且EST的高度保守性使得EST-SSR在同物种的不同品种间乃至不同物种间具有较高的通用性,所以可以对决定重要表型性状的等位基因进行直接鉴定[24-25]。本研究利用桂花转录组开发获得的EST-SSR引物在桂花品种鉴定中具有可靠性和适用性,可为后期桂花遗传多样性和亲缘关系以及遗传连锁图谱的构建等研究奠定基础。
EST-SSR primers and their application in cultivar identification of Osmanthus fragrans
-
摘要: 利用L16(45)正交实验设计,针对模板DNA,镁离子(Mg2+),dNTPs,TaqDNA酶和引物5个因素进行正交优化,建立适用于桂花Osmanthus fragrans表达序列标签-简单重复序列(EST-SSR)的最优扩增反应体系,将其用于桂花转录组EST-SSR多态性引物的筛选,最后利用筛选得到的多态性引物和聚类分析对14份四川省常见的栽培丹桂(SC01~SC14)样本进行品种鉴定。结果表明:桂花EST-SSR最优反应体系包含40 ng模板DNA,2.25 mmol·L-1 Mg2+,0.3 μmol·L-1引物,0.3 mmol·L-1 dNTPs,1.25×16.67 nkat TaqDNA聚合酶,10×PCR Buffer缓冲液2 μL,双蒸水补至20 μL;从150对EST-SSR引物中筛选得到了19对稳定性好、多态性高的SSR引物,多态性从高到低依次为五核苷酸(25.00%),六核苷酸(19.44%)和三核苷酸(16.67%)重复类型。利用筛选得到的3对高度多态性SSR引物(OF8623-1677,OF24299-844和OF28774-2088)对四川省成都市周边常见栽培的14份丹桂(Aurantiacus group)样本进行了有效鉴定。结果表明:14份样本中,有12份可判定为‘朱砂丹桂’‘Zhusha Dangui’,SC06为‘堰红桂’‘Yanhong Gui’,SC08与‘满条红’‘Mantiao Hong’和‘状元红’‘Zhuangyuan Hong’亲缘关系较近。本研究也为今后利用EST-SSR标记对桂花指纹图谱构建、遗传多样性分析、分子育种及品种鉴定与保护等工作奠定基础。Abstract: In this study, expressed sequence tags (ESTs) were identified by EST-simple sequence repeat (SSR) markers. The optimal amplification reaction system for EST-SSR was established by orthogonal design of an L16(45) orthogonal experiment, and it was established for orthogonal design of template DNA, Mg2+, dNTPs, and TaqDNA enzyme and primer. Then, Osmanthus fragrans transcriptase EST-SSR polymorphism primers were screened. Finally, the polymorphic primers and cluster analysis were used to identify the cultivars cultivated in Sichuan Province. Results showed that the EST-SSR optimal reaction system of O. fragrans contained 40 ng template DNA, 2.25 mmol·L-1 Mg2+, 0.3 μmol·L-1 primer, 0.3 mmol·L-1 dNTPs, TaqDNA polymerase 1.25×16.67 nkat, 10×PCR Buffer 2 μL, and ddH2O to 20 μL. A total of 19 SSR primers with high stability and high polymorphism were screened from 150 pairs of EST-SSR primers. The polymorphism was five nucleotides (25.00%), six nucleosides acid (19.44%), and trinucleotide (16.67%). Altogether a total of 14 samplings of O. fragrans cultivars cultivated in Chengdu, Sichuan Province were identified by high sensitivity polymorphic SSR primers. Analysis of genetic diversity, molecular breeding, and identification and protection of cultivars have laid a strong foundation for O. fragrans.
-
Key words:
- botany /
- Osmanthus fragrans /
- EST-SSR /
- system optimization /
- primer screening /
- variety identification
-
表 1 EST-SSR正交试验设计L16(45)
Table 1. EST-SSR orthogonal experimental design L16(45)
组合编号 DNA/ng Mg2+/(mmol·L-1) dNTPs/(mmol·L-1) Taq酶/(×16.67 nkat) 引物/(μmol·L-1) 1 40 1.125 0.15 0.50 0.15 2 40 1.500 0.20 0.75 0.20 3 40 1.875 0.25 1.00 0.25 4 40 2.250 0.30 1.25 0.30 5 60 1.875 0.20 1.25 0.20 6 60 2.250 0.15 1.00 0.15 7 60 1.125 0.30 0.75 0.30 8 60 1.500 0.25 0.50 0.25 9 80 2.250 0.25 0.75 0.25 10 80 1.875 0.30 0.50 0.30 11 80 1.500 0.15 1.25 0.15 12 80 1.125 0.20 1.00 0.20 13 100 1.500 0.30 1.00 0.30 14 100 1.125 0.25 1.25 0.25 15 100 2.250 0.20 0.50 0.20 16 100 1.875 0.15 0.75 0.15 表 2 150对随机引物信息表
Table 2. Random primers information of 150 pairs
重复次数 重复类型 总数 5 6 7 8 9 10 11 12 >12 单核苷酸 1 1 2 7 12 二核苷酸 1 1 1 1 3 3 1 10 21 三核苷酸 10 5 2 5 4 2 30 四核苷酸 4 3 1 1 10 五核苷酸 9 1 12 六核苷酸 18 7 7 2 1 36 复合重复 29 总计 41 17 11 9 6 6 4 3 17 150 表 3 品种鉴定材料信息表
Table 3. Material information for cultivars
名称 样本采集地 SC01 成都市街区 SC02 成都市邛崃市 SC03 成都市崇州市区 SC04 成都市大邑县 SC05 成都市都江堰市 SC06 成都市彭州市 SC07 成都市都江堰市 SC08 成都市新都区 SC09 成都市新都区 SC10 成都市新都区 SC11 成都市郫县 SC12 成都市郫县 SC13 成都市温江区 SC14 成都市温江区 ‘朱砂丹桂’ ‘Zhusha Dangui’ 浙江农林大学桂花苗圃 ‘橙红丹桂’ ‘ Chenghong Dangui ’ 浙江农林大学桂花苗圃 ‘状兀红’ ‘ Zhuangyuan Hong ’ 浙江农林大学桂花苗圃 ‘武夷丹桂’ ‘Wuyi Dangui’ 浙江农林大学桂花苗圃 ‘堰虹桂’ ‘ Yanhong Gui ’ 浙江农林大学桂花苗圃 ‘满条红’ ‘ Mantiao Hong ’ 浙江农林大学桂花苗圃 ‘上海丹桂’ ‘ Shanghai Dangui ’ 浙江农林大学桂花苗圃 表 4 150对随机EST-SSR引物扩增情况
Table 4. EST-SSR amplification of 150 primers
重复类型 总数 成功扩增/对 比例/% 多态引物/对 多态性比例/% 单核苷酸 12 5 41.67 0 0 二核苷酸 21 7 33.33 0 0 三核苷酸 30 20 66.67 5 16.67 四核苷酸 10 5 50.00 1 10.00 五核苷酸 12 6 50.00 3 25.00 六核苷酸 36 17 47.22 7 19.44 复合重复 29 19 65.52 3 10.34 总体 150 79 52.67 19 12.67 表 5 3对桂花EST-SSR引物序列
Table 5. Three EST-SSR primer sequences of Osmanthus fragrance
编号 名称 引物序列 退火温度/℃ 重复单元 预期片段大小/bp 9 OF8623-1677 F-TCCACACGCTGAAACTCCTA
R-TCGTATGATGGAGCAGCAAG58 (AGAAA)5 156 13 OF24299-844 F-TACAAAGAAGCCCCACCAAC
R-ATTGAGAGCAACCATTTGCC57 (CCCAAA)5 240 19 OF28774-2088 F-TGACACCATGACTTCCCAGA
R-CAATTGTCGGTGTTGGTTTG57 (TTAGCC)5 174 表 6 品种鉴定带型信息统计表
Table 6. Cultivars with type information statistics
样本 OF8623-1677 OF24299-844 OF28774-2088 SC01 DD AA BC SC02 DD AA BC SC03 DD AA BC SC04 DD AA BC SC05 DD AA BC SC06 CC AC CD SC07 DD 99 BC SC08 99 AD BC SC09 DD AF BC SC10 DD AA BC SC11 DD AA BC SC12 DD 99 BC SC13 DD AA BC SC14 DD AA CC ‘朱砂丹桂’ DD AA BC ‘橙红丹桂’ 99 AE DD ‘状元红’ BB AF CC ‘武夷丹桂’ CC AD CC ‘堰虹桂’ CC AC CD ‘满条红’ BB AF BC ‘上海丹桂’ AA AB AB 说明:相同字母组合代表纯合子,不同字母组合杂合子,99表示无扩增条带 -
[1] 张美珍, 邱莲卿, 缪柏茂.中国植物志:第61卷[M].北京:科学出版社, 1992. [2] 向其柏, 刘玉莲.中国桂花品种图志[M].杭州:浙江科学技术出版社, 2008:1-377. [3] 臧德奎, 向其柏, 刘玉莲.木犀属品种分类研究[J].林业科学, 2006, 42(5):17-21. ZANG Dekui, XIANG Qibai, LIU Yulian. Notes on cultivar classification in Osmanthus[J]. Sci Silv Sin, 2006, 42(5):17-21. [4] 臧德奎, 向其柏, 刘玉莲, 等.中国桂花的研究历史、现状与桂花品种国际登录[J].植物资源与环境学报, 2003, 12(4):49-53. ZANG Dekui, XIANG Qibai, LIU Yulian, et al. The studying history and the application to international cultivar registration authority of sweet osmanthus (Osmanthus fragrans Lour.)[J]. J Plant Resour Environ, 2003, 12(4):49-53. [5] 胡菀, 罗意, 阳亿, 等.野生桂花的遗传多样性和遗传结构研究[J].园艺学报, 2014, 41(7):1427-1435. HU Wan, LUO Yi, YANG Yi, et al. Genetic diversity and population genetic structure of wild sweet osmanthus revealed by microsatellite markers[J]. Acta Hortic Sin, 2014, 41(7):1427-1435. [6] DUAN Yifan, WANG Xianrong, XIANG Qibai, et al. Genetic diversity of androdioecious Osmanthus fragrans (Oleaceae) cultivars using microsatellite markers[J]. Appl Plant Sci, 2013, 1(6):221-230. [7] 罗冉, 吴委林, 张旸, 等. SSR分子标记在作物遗传育种中的应用[J].基因组学与应用生物学, 2010, 29(1):137-143. LUO Ran, WU Weilin, ZHANG Yang, et al. SSR marker and its application to crop genetics and breeding[J]. Genom Appl Biol, 2010, 29(1):137-143. [8] ZHANG Zhirong, FAN Dengmei, GUO Shiquan, et al. Development of 29 microsatellite markers for Osmanthus fragrans (Oleaceae), a traditional fragrant flowering tree of China[J]. Am J Bot, 2011, 98(12):e356-e359. [9] 马寅峰. 桂花SSR引物的开发和SCoT分子标记体系的建立[D]. 开封: 河南大学, 2015. MA Yanfeng. Development of SSR Primers and Establishment of SCoT Molecular Marker System in Osmanthus fragrans[D]. Kaifeng:Henan University, 2015. [10] ELLIS J R, PASHLEY C H, BURKE J M, et al. High genetic diversity in a rare and endangered sunflower as compared to a common congener[J]. Mol Ecol, 2006, 15(9):2345-2355. [11] KANTETY R V, ROTA M L, MATTHEWS D E, et al. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat[J]. Plant Mol Biol, 2002, 48(5/6):501-510. [12] 徐沂春. 桂花遗传多样性及不同性别花的花芽特性[D]. 杭州: 浙江农林大学, 2013: 32-50. XU Yichun. The Genetic Diversity and Flower Bud Characteristics of Different Sex in Osmanthus fragrans[D]. Hangzhou:Zhejiang A&F University, 2013:32-50. [13] 帕提古丽·麦麦提敏, 张延辉, 李培英, 等.狗牙根EST-SSR反应体系优化及其遗传多样性初步分析[J].新疆农业大学学报, 2014, 37(2):112-118. Patiguli Maimaitimin, ZHANG Yanhui, LI Peiying, et al. Preliminary analysis on optimization of EST-SSR PCR reaction system and genetic diversity for Cynodon dactylon L.[J]. J Xinjiang Agric Univ, 2014, 37(2):112-118. [14] 赵克奇, 董黎, 王少明, 等.刺槐EST-SSR标记PCR反应体系的优化[J].中国农学通报, 2014, 30(22):45-52. ZHAO Keqi, DONG Li, WANG Shaoming, et al. The optimization of EST-SSR PCR reaction system for Robinia pseudoacacia L.[J]. Chin Agric Sci Bull, 2014, 30(22):45-52. [15] 高志红, 章镇, 韩振海, 等.果梅SSR反应体系的优化[J].南京农业大学学报, 2002, 25(4):19-22. GAO Zhihong, ZHANG Zhen, HAN Zhenhai, et al. Optimization on SSR analysis system of Japanese apricot (Prunus mume Sieb. et Zucc.)[J]. J Nanjing Agric Univ, 2002, 25(4):19-22. [16] 尤有利, 王江波, 施维属, 等.荔枝DNA提取及RAPD扩增条件优化[J].生物技术通报, 2010(4):112-115. YOU Youli, WANG Jiangbo, SHI Weishu, et al. Extration of genomic DNA of Litchi chinensis and optimization of the RAPD reaction system[J]. Biotechnol Bull, 2010(4):112-115. [17] 郭大龙, 罗正荣.部分柿属植物SRAP-PCR反应体系的优化[J].果树学报, 2006, 23(1):138-141. GUO Dalong, LUO Zhengrong. Optimization of SRAP-PCR in some Diospyros spp.[J]. J Fruit Sci, 2006, 23(1):138-141. [18] 谢运海, 夏德安, 姜静, 等.利用正交设计优化水曲柳ISSR-PCR反应体系[J].分子植物育种, 2005, 3(3):445-450. XIE Yunhai, XIA De'an, JIANG Jing, et al. Optimizationg for ISSR-PCR system of Fraxinus mandshurica Rupr. using oryhogonal design[J]. Mol Plant Breed, 2005, 3(3):445-450. [19] 袁存权, 李允菲, 杨妮娜, 等.刺槐SRAP-PCR反应体系优化及引物筛选[J].分子植物育种, 2011, 9(25):1182-1188. YUAN Cunquan, LI Yunfei, YANG Nina, et al. Optimization of SRAP-PCR reaction system and selection of primers for Robinia pseudoacacia[J]. Mol Plant Breed, 2011, 9(25):1182-1188. [20] 杨传平, 王艳敏, 魏志刚.利用正交设计优化白桦的SSR-PCR反应体系[J].东北林业大学学报, 2006, 34(6):1-3. YANG Chuanping, WANG Yanmin, WEI Zhigang. Optimization of SSR-PCR system for Betula platyphylla using orthogonal design[J]. J Northeast For Univ, 2006, 34(6):1-3. [21] 王艳敏, 魏志刚, 杨传平.白桦EST-SSR信息分析与标记的开发[J].林业科学, 2008, 44(2):78-84. WANG Yanmin, WEI Zhigang, YANG Chuanping, Data mining for SSRs in ESTs and EST-SSR marker development in Betula platyphylla[J]. Sci Silv Sin, 2008, 44(2):78-84. [22] 李响, 杨楠, 赵凯歌, 等.蜡梅转录组EST-SSR标记开发与引物筛选[J].北京林业大学学报, 2013, 35(1):25-32. LI Xiang, YANG Nan, ZHAO Kaige, et al. Development and primer selection of EST-SSR molecular markers based on transcriptome sequencing of Chimonanthus praecox[J]. J Beijing For Univ, 2013, 35(1):25-32. [23] 徐阳, 陈金慧, 李亚, 等.杉木EST-SSR与基因SSR引物开发[J].南京林业大学学报(自然科学版), 2014, 38(1):9-14. XU Yang, CHEN Jinhui, LI Ya, et al. Development of EST-SSR and genomic-SSR in Chinese fir[J]. J Nanjing For Univ Nat Sci Ed, 2014, 38(1):9-14. [24] CHEN X, SALAMINI F, GEBHARDT C. A potato molecular-function map for carbohydrate metabolism and transport[J]. Theor Appl Genet, 2001, 102(2/3):284-295. [25] 忻雅, 崔海瑞, 张明龙, 等.白菜EST-SSR标记的通用性[J].细胞生物学杂志, 2006, 28(2):248-252. XIN Ya, CUI Hairui, ZHANG Minglong, et al. The transferability of Chinese cabbage EST-SSR markers[J]. Chin J Cell Biol, 2006, 28(2):248-252. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2018.02.015







 下载:
下载: