-
竹类植物属单子叶禾本科Poaceae植物,具重要的经济价值。全世界木本竹类有70多属1 200余种[1]。中国是竹类资源最丰富的国家,素有“竹子王国”之称[2]。虽然已对毛竹Phyllostachys edulis基因组进行了测序[3],但关于竹类植物的研究仍集中在分子标记的开发与应用,功能基因的分离与生物学功能鉴定及高通量的转录组测序等[4-8]。目前,竹类植物中所开发的分子标记基本以未知序列为主,如随机扩增多态性DNA(random amplified polymorphism DNA,RAPD),限制性片段长度多态性(restriction fragment length polymorphism, RFLP),扩增片段长度多态性(amplified fragment length polymorphism, AFLP)等[4],因此,开发出功能基因的分子标记用于比较基因组学及竹类植物中功能基因的挖掘日益重要。在许多植物类群的研究中,低拷贝的核内基因相比能提供更多的信息位点,具备更强的优势[9-12]。Wu等[13]首次基于茄科Solanaceae植物开发出单拷贝直系同源基因(single-copy orthologous genes,COSⅡ)分子标记,并成功用于茄科植物间的同线性研究[14-15]。该标记是基于核基因内保守的直系同源基因开发出来的一种新型分子标记,操作简单、费用低。一般来说,COS标记位点都位于编码区,通常涉及的基因是编码生命必需的酶、辅酶或关键性调控蛋白的基因,具有很高的保守性。COS标记的开发对于比较作图,线性同向序列分析,系统发生学和分子进化等方面的研究具有重要意义[16]。禾本科是种子植物中最有经济价值的。该科中水稻Oryza sativa,高粱Sorghum bicolor和玉米Zea mays等的基因组序列相继公布,基于这些基因组序列,Liu等[17]共鉴定出2~684个COSⅡ基因,并开发出1 072个COSⅡ分子标记,该标记在竹类植物中具有较好的通用性。毛竹基因组序列虽已测序,但仍有部分基因未能获得。因此,为开发更多的适用于禾本科植物通用性分子标记,丰富竹类植物的遗传信息,我们采用同样的方法开发了14对COSⅡ引物,在刚竹属Phyllostachys植物中进行了检测,对部分扩增结果进行了测序,并对其单核苷酸多态性(single nucleotide polymorphism,SNP)位点进行了分析和验证。
-
选取竹亚科Bambusoideae的5个竹种进行试验(表 1)。供试材料都采自浙江农林大学翠竹园。竹叶采集时间为2012年4月,所采集的样本均通过形态学鉴定,采集后用液氮速冻,放入-70 ℃冰箱保存。大肠埃希菌菌株Escherichia coliDH-5α和质粒回收试剂盒购自于生工生物工程(上海)有限公司。载体、聚合酶链式反应(PCR)试剂均购自宝生物工程(大连)有限公司。胶回收试剂盒购自北京鼎国昌盛生物技术有限责任公司。
表 1 供试样本材料
Table 1. Samples used in the study
编号 样本竹种 1 黄秆乌哺鸡竹Phyllostachys vivax f. aureocaulis 2 雷竹Ph.violascens 3 毛竹Ph. edulis 4 绿粉竹Ph.virdiglaucescens 5 紫竹Ph.nigra -
采用十六烷基三甲基溴化铵(CTAB)法提取5个竹种叶片基因组DNA[18]。
-
参照Liu等[17]的方法,基于禾本科植物中COSⅡ基因序列设计了14对引物,引物序列及其在水稻中序列号如表 2。
表 2 引物序列
Table 2. Primer sequences of the 14 COS Ⅱ markers
引物名称 上游引物($ 5' \to 3' $) 下游引物($ 5' \to 3' $) 序列号 Primer 1 TACCCGAAGCGGAGCGCGGCGGTGGT CGCTCCGGCATGTCCTCGAAGAACTT LOC_Os12g34340 Primer 2 ATGAGCTTCGCGGATCTGGAG CGAGCGAGTTGAGGAGGCGGCGGTA LOC_Os02g47440.1 Primer 3 GCACCGCCGACTTCTTCTAC CGATGAGCTTGTGCTGCTT L0C_0s03g60750.1 Primer 4 ATCGCAACGAAAGAAAGAGC GCTTCAACCATAGGCTCAGG LOC_Os04g56070.1 Primer 5 CACTCAAGGTCGATGTGGAT CCTTCTTCCAAAGCAAGCTG LOC_Os05g07632.1 Primer 6 ACTGAATGGCCTGTTGAAGC TGGAATCCCCTCTCAATACC LOC_Os06g14740.1 Primer 7 GGGACCTACAGCAGCCATT ATCACACGGAGTAGGCCATC LOC_Os03g47930.1 Primer 8 CGATGGCATCTACCAAAAGG GGCTGTCAGCGTCCATTAC LOC_Os01g70570.1 Primer 9 GCTGACGAGAACCATGTCAA ATCTCGGTGGCATACCCTCT LOC_Os03g54780.1 Primer 10 GAGAAGCGGGAGGAGTTCAC AGGGACACGACACTGAGTCC LOC_Os10g40130.1 Primer 11 GAGCCCTGACAGTCGTCATA AATCCTTGCAGTCACGCCTA LOC_Os10g41370.1 Primer 12 GACCGTAAGACCTTCCTCGACTTCCT TAGATCGTAATCAGCGTCTGCGA LOC_Os03g46190.2 Primer 13 CCTCTTCGTGAGGTACTACGT AGGAACATGTCCTCCTCG LOC_Os04g38320.1 Primer 14 GAGAACATGGCTAGGCTGGC ACCATGCTCCTGCTAAGTGCCA LOC_Os06g37640.1 -
取各竹种叶片约2.0 g,采用改良CTAB法提取叶片总DNA。PCR反应体系总体积为10.0 μL,其中含50 ng模板DNA;上游、下游引物各0.5 μmol·L-1;200.0 μmol·L-1三磷酸碱基脱氧核苷酸(dNTP);1.0 μmol·L-1 Taq DNA聚合酶;1.0 μL 10×PCR反应缓冲液(内含氯化镁)。采用降落PCR技术,反应条件为:94 ℃预变性5 min;94 ℃变性30 s,59 ℃退火30 s(递减0.3 ℃·循环-1),72 ℃延伸1 min,10个循环;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,10个循环;72 ℃延伸5 min。组合至少重复2次·引物-1,以确保扩增结果的可靠性。扩增产物在体积分数为6%非变性聚丙烯酰胺凝胶上,稳压200 V,电泳80 min。对胶染色,对聚丙烯酰胺凝胶电泳(PAGE)分离后的目的片段确认拍照。
-
以相同迁移位置记作1个扩增位点,记录每对引物在5个材料中PCR扩增获得的位点总数Nt;5个材料中扩增获得的位点数Nb;及多态性位点数Np。在每个引物的相同迁移位点上,以“1”或“0”记录条带信息,其中“1”代表有扩增产物,“0”代表无扩增产物,建立一个数据矩阵。把距离矩阵输入NTedit软件中,用Ntsys模型构建树状聚类图。
-
将PAGE电泳后的目的条带进行割胶回收,将其作为第2次PCR的模板,将2次PCR产物割胶回收再测序,对所得序列采用软件DNAMAN进行比较,分析单核苷酸多态性(SNP)位点。应用内切酶SacⅠ分别对引物1在5个竹种的PCR产物回收进行酶切反应。反应体系及条件为PCR产物6.0 μL,缓冲液1.0 μL,内切酶0.5 μL,双蒸水2.5 μL,混匀,37 ℃水浴12 h,20.0 g·kg-1琼脂糖电泳检测。
-
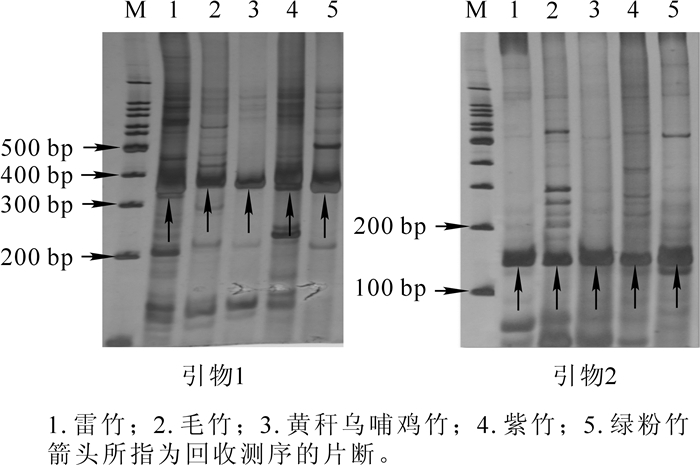
利用新设计的14对引物分别对5个竹种进行PCR扩增。结果显示:12对引物可以在至少3个材料中得到特异性PCR产物,其中7对引物在5个供试材料中均能扩增到产物(表 3)。在可扩增出产物的引物中,有13对引物(92.9%)在供试材料中表现出多态性,仅引物11在供试材料间没有多态性位点。对引物1从雷竹、毛竹、黄秆乌哺鸡竹、紫竹、绿粉竹中扩增的条带进行测序,测序片段的长度除绿粉竹是347 bp,其余都是363 bp(图 1)。绿粉竹与其他竹种间序列大小间的差异主要是由于该基因内含子长度不一造成的。而且毛竹中获得的序列与网站上的序列完全一致。将所测序列与水稻序列进行比对,发现所扩增的序列与水稻序列间的同源性均达到92.77%以上,且在外显子部分高度保守,内含子表现出较大的差异,表明它们均为水稻的同源目标基因(图 2)。对引物2从5个样本中扩增出的条带进行测序,所得片段长度均为132 bp(图 1),分析发现该段序列不包含内含子。同样,从毛竹中获得的序列与美国生物技术信息中心(NCBI)网站上的序列完全一致。对所获得的序列进行同源性分析,发现获得的4条序列与水稻中该基因序列间的差异非常小,彼此之间的同源性均达到97.24%。

图 1 2对引物在5个竹种的扩增结果
Figure 1. PCR products of primer 1 and primer 2 in the 5 bamboo species separated by 6% non-denaturing PAGE gel

图 2 引物1扩增产物的序列比对
Figure 2. Multiple alignment of the Oryza sativa gene (LOC_Os12g0527850) and the sequences of the 5 bamboo species based on the primer 1
表 3 不同引物在5个供试样本中的扩增结果
Table 3. Results obtained by PCR with COS Ⅱ primers in samples tested
引物名称 所在基因的功能或推测功能 Nt Nb Np Primer 1 tetratricopeptide-like helicaldomain-containing protein 6 1~2 6 Primer 2 syntaxin-22-like 6 1~4 6 Primer 3 ribosomal RNA large subunit methyltransferase J 4 1~4 3 Primer 4 COP9 signalosome complex subunit 5b 4 0~3 4 Primer 5 haloacid dehalogenase-like hydrolase family protein 3 0~3 3 Primer 6 NAD binding domain of 6-phosphogluconate dehydrogenase containing protein 2 0~1 2 Primer 7 expressed protein 8 0~8 8 Primer 8 acyltransferase 2 0~2 2 Primer 9 STE_PAK_Ste20_KHSh_GCKh_HPKh.1-STE kinases include homologs to sterile 7, sterile 11, and sterile 20 from yeast 3 0~2 3 Primer 10 UDP-N-acetylmuramoylalanyl-D-glutamate-2,6-diaminopimelate ligase 7 1~6 6 Primer 11 WD repeat-containing protein 8 1 1 0 Primer 12 parafibromin 8 1~8 8 Primer 13 expressed protein 6 1~5 5 Primer 14 expressed protein 1 0~1 1 若按所用14对COSⅡ引物所在基因编码产物的功能进行分类,可将它们大致分为5类,分别为蛋白质修饰、细胞成分组装、代谢、生化过程及其他。从拷贝数来看,这些引物所在基因在水稻中均为单拷贝,但本研究的结果显示,在所扩增的样本中同样表现出单拷贝的特性。有些引物在某些供试材料所扩增出的位点有些具多态性,有些位点不具有多态性。
-
利用14对COS Ⅱ标记引物在这些试验材料中扩增共得到56个多态位点。根据带型的不同,计算试验材料间的遗传距离。根据遗传距离,利用NTsys 2.10e软件,得到5个样本的聚类图(图 3),紫竹与其他4个竹种亲缘关系最远,毛竹与绿粉竹亲缘关系最近。该结果与形态学分类结果吻合。
-
序列比对分析发现,所扩增的序列存在丰富的单核苷酸多态性(SNP)位点。引物1所获序列的编码区(259 bp)存在41个SNP位点,多态性频率为1 SNP/6.3 bp,其中转换型SNP占90%,颠换型SNP占10%(图 2)。对引物1所编码的氨基酸序列比对分析,发现9处SNP引起氨基酸的变异,且第61碱基处的T/C转换,导致雷竹氨基酸序列的提前终止;第151碱基处的T/C转换,导致毛竹氨基酸序列的提前终止。有些SNP位点也会引起酶切位点的变异,如第96碱基处的T/C转换导致毛竹SacⅠ(GAGCTC)酶切位点的消失(图 4)。引物2所获序列(132 bp)共存在7个SNP位点,多态性频率为1 SNP/19 bp,其中2个转换型,5个颠换型,这7个SNP引起2个氨基酸位点的改变(表 4)。

图 4 用SacⅠ酶切检测结果
Figure 4. PCR products of primer in the 5 samples was digested with restriction enzyme SacⅠ
表 4 引物2所扩增片段单核甘酸变异
Table 4. Variation of single nucleotides in the segment of primer 2
样品名称 单核苷酸变异位点 27e 48e 51e 69e 85e 97e 102e 紫竹 G G C C C(Q) G(A) G 黄杆乌哺鸡竹 A A G G C(Q) T(S) T 雷竹 A A G G C(Q) T(S) T 毛竹 A A G G A (K) T(S) T 绿粉竹 A A G G C(Q) T(S) T 说明:下划线表示引起氨基酸变异的SNP位点,括号表示该位点的氨基酸。 -
引物1的扩增产物间存在多个SNP位点,为了验证SNP的可靠性,采用限制性内切酶SacⅠ对引物1在5个竹种的PCR产物进行酶切检测,结果显示:从毛竹中获得的片段不能被内切酶SacⅠ切开,的确在该酶切位点发生了变异,其余竹种的扩增产物在该位点均能被切为2条带,且2条带大小之和与原来的条带大小吻合,电泳检测结果与测序结果一致(图 4)。
-
COSⅡ标记位点是物种进化上相对保守的位点,可用于不同的基因组之间比较作图,构建种间线性遗传图谱和分析遗传关系。本研究参照Liu等[17]的方法,依据禾本科植物中COSⅡ基因序列设计了14对引物。利用这14对引物分别对5个竹种进行PCR扩增,结果显示:11对引物可以在至少3个材料中得到特异性PCR产物,其中7对引物在5个供试材料中均能扩增到产物。选取2对引物的扩增条带进行测序,相同引物扩增出的序列均为水稻的同源目的序列。引物1在5个样本中获得的序列间存在明显差异,除内含子序列差异较大外,在外显子区域也存在丰富的SNP变异位点。这些SNP位点或者引起氨基酸位点变异,或者引起酶切位点变异,甚至会导致氨基酸序列的提前终止,如扩增序列中第96碱基处的差异导致毛竹SacⅠ(GAGCTC)酶切位点;第61和151碱基处的T/C转换,分别导致雷竹与毛竹氨基酸序列的提前终止。我们还对供试材料进行了聚类分析,结果与形态学聚类结果一致,表明开发的14对COSⅡ引物在在刚竹属植物中具有一定的通用性。
SNP是可以鉴定种下等级的最为精确的分子标记,可用于对亲缘关系较近的近缘种及不同基因型的品种进行遗传多样性分析。由于SNP存在于整个基因组中,在许多重要基因座或邻近区,比其他类型分子标记更能提供更多的特殊性标记,可以更直接、快捷地用于分子育种[19]。SNP位点的可靠性一般用PCR-RFLP方法验证,此方法对外形上易混淆种类的鉴定具有准确、快速、价廉和重现性好等的优点。本研究所测的引物1和引物2的扩增产物存在丰富的SNP位点,这一结果与竹类植物基因间存在丰富的SNP位点相吻合[3, 19]。Germano等[20]通过PCR-RFLP方法鉴定出3个云杉Picea近缘种间的多态性。张婷等[21]采用该方法鉴别了药用植物束花石斛Dendrobium chrysanthum,流苏石斛Dendrobium fimbriatum及其形态相似种;曹东伟等[22]用该技术分析了中国樱桃Prunus pseudocerasus分子亲缘地理学研究。本研究亦采用PCR-RFLP方法对引物1所扩增的产物进行了酶切验证,并鉴定出了毛竹和绿粉竹2个相似种。由此,本研究所获得的标记在竹类植物中通用,并有望开发成SNP标记而将不同竹种分开。
Universal genetic markers for the Poaceae family
-
摘要: 为开发更多的适用于禾本科Poaceae植物通用性分子标记, 丰富竹类植物的遗传信息, 依据禾本科单拷贝同源基因(COSⅡ)已知序列的保守区域设计了14对引物, 以此引物对刚竹属Phyllostachys植物进行聚合酶链式反应(PCR)扩增, 并对部分扩增产物进行测序。结果所有引物可以在至少1个材料中得到特异性扩增, 其中7对引物在5个供试材料中均能扩增到产物, 13对引物在5个样本中表现出多态性, 占引物总数的92.9%。对部分扩增产物进行测序, 所测序列均为特异性扩增序列。同一引物在不同样本中的扩增片段间存在单核苷酸多态性(SNP)位点, 部分SNP位点会导致氨基酸序列提前终止或酶切位点变化。对有合适内切酶存在的变异位点进行PCR-RFLP验证, 验证结果与测序结果一致。利用NTsys 2.10e软件, 对5个供试竹种材料进行了亲缘关系分析, 毛竹Phyllostachys edulis与绿粉竹Phyllostachys virdiglaucescens亲缘关系最近, 紫竹Phyllostachys nigra与其他4个竹种亲缘关系最远。所开发的14个COSⅡ引物在刚竹属植物中具一定通用性, 同时挖掘出更多竹类植物中的遗传信息。
-
关键词:
- 植物学 /
- 竹类植物 /
- 通用性分子标记 /
- 单拷贝同源基因(COSⅡ) /
- 单核苷酸多态(SNP) /
- PCR-RFLP
Abstract: To develop more universal molecular markers for grasses, rich genetic information of bamboo plants and 14 pairs of primers were designed according to the conserved regions of the single-copy orthologous (COS Ⅱ) genes of the Poaceae (grass) family. Then, polymerase chain reaction (PCR) was used to test five bamboo accessions. Also, Primer 1 and Primer 2 were used to sequence parts of the amplified products, and single nucleotide polymorphisms (SNP) sites were validated with the PCR-restriction fragment length polymorphism (RFLP) test. Based on different band styles amplified with the 14 primer pairs, genetic distance was calculated with NT sys 2.10e software and a cluster analysis of five species:Phyllostachys edulis, Ph. viridiglaucescens, Ph. nigra, Ph. violascens, and Ph. vivax f. aureocaulis were conducted. Results showed that all primers exhibited specific PCR products in at least one sample. Seven pairs of primers were able to amplify the products in tested samples with 13(92.9%) pairs of primers exhibiting polymorphic PCR products in all samples. Sequencing showed that Primer 1 and Primer 2 were homologous. These sequences were amplified with the same primer and existed at SNP sites. Some SNP sites led to early termination of the amino acid sequence, and some resulted in changes of restriction endonuclease sites. Results of the PCR-RFLP test corresponded with the sequencing. The cluster analysis indicated that Ph. edulis and Ph. viridiglaucescens were closely clustered, and Ph. nigra was the most isolated. Overall, this classification coincided with morphological classification; so, these molecular markers were suitable for application in genetic analyses and other related research with Phyllostachys. -
表 1 供试样本材料
Table 1. Samples used in the study
编号 样本竹种 1 黄秆乌哺鸡竹Phyllostachys vivax f. aureocaulis 2 雷竹Ph.violascens 3 毛竹Ph. edulis 4 绿粉竹Ph.virdiglaucescens 5 紫竹Ph.nigra 表 2 引物序列
Table 2. Primer sequences of the 14 COS Ⅱ markers
引物名称 上游引物($ 5' \to 3' $) 下游引物($ 5' \to 3' $) 序列号 Primer 1 TACCCGAAGCGGAGCGCGGCGGTGGT CGCTCCGGCATGTCCTCGAAGAACTT LOC_Os12g34340 Primer 2 ATGAGCTTCGCGGATCTGGAG CGAGCGAGTTGAGGAGGCGGCGGTA LOC_Os02g47440.1 Primer 3 GCACCGCCGACTTCTTCTAC CGATGAGCTTGTGCTGCTT L0C_0s03g60750.1 Primer 4 ATCGCAACGAAAGAAAGAGC GCTTCAACCATAGGCTCAGG LOC_Os04g56070.1 Primer 5 CACTCAAGGTCGATGTGGAT CCTTCTTCCAAAGCAAGCTG LOC_Os05g07632.1 Primer 6 ACTGAATGGCCTGTTGAAGC TGGAATCCCCTCTCAATACC LOC_Os06g14740.1 Primer 7 GGGACCTACAGCAGCCATT ATCACACGGAGTAGGCCATC LOC_Os03g47930.1 Primer 8 CGATGGCATCTACCAAAAGG GGCTGTCAGCGTCCATTAC LOC_Os01g70570.1 Primer 9 GCTGACGAGAACCATGTCAA ATCTCGGTGGCATACCCTCT LOC_Os03g54780.1 Primer 10 GAGAAGCGGGAGGAGTTCAC AGGGACACGACACTGAGTCC LOC_Os10g40130.1 Primer 11 GAGCCCTGACAGTCGTCATA AATCCTTGCAGTCACGCCTA LOC_Os10g41370.1 Primer 12 GACCGTAAGACCTTCCTCGACTTCCT TAGATCGTAATCAGCGTCTGCGA LOC_Os03g46190.2 Primer 13 CCTCTTCGTGAGGTACTACGT AGGAACATGTCCTCCTCG LOC_Os04g38320.1 Primer 14 GAGAACATGGCTAGGCTGGC ACCATGCTCCTGCTAAGTGCCA LOC_Os06g37640.1 表 3 不同引物在5个供试样本中的扩增结果
Table 3. Results obtained by PCR with COS Ⅱ primers in samples tested
引物名称 所在基因的功能或推测功能 Nt Nb Np Primer 1 tetratricopeptide-like helicaldomain-containing protein 6 1~2 6 Primer 2 syntaxin-22-like 6 1~4 6 Primer 3 ribosomal RNA large subunit methyltransferase J 4 1~4 3 Primer 4 COP9 signalosome complex subunit 5b 4 0~3 4 Primer 5 haloacid dehalogenase-like hydrolase family protein 3 0~3 3 Primer 6 NAD binding domain of 6-phosphogluconate dehydrogenase containing protein 2 0~1 2 Primer 7 expressed protein 8 0~8 8 Primer 8 acyltransferase 2 0~2 2 Primer 9 STE_PAK_Ste20_KHSh_GCKh_HPKh.1-STE kinases include homologs to sterile 7, sterile 11, and sterile 20 from yeast 3 0~2 3 Primer 10 UDP-N-acetylmuramoylalanyl-D-glutamate-2,6-diaminopimelate ligase 7 1~6 6 Primer 11 WD repeat-containing protein 8 1 1 0 Primer 12 parafibromin 8 1~8 8 Primer 13 expressed protein 6 1~5 5 Primer 14 expressed protein 1 0~1 1 表 4 引物2所扩增片段单核甘酸变异
Table 4. Variation of single nucleotides in the segment of primer 2
样品名称 单核苷酸变异位点 27e 48e 51e 69e 85e 97e 102e 紫竹 G G C C C(Q) G(A) G 黄杆乌哺鸡竹 A A G G C(Q) T(S) T 雷竹 A A G G C(Q) T(S) T 毛竹 A A G G A (K) T(S) T 绿粉竹 A A G G C(Q) T(S) T 说明:下划线表示引起氨基酸变异的SNP位点,括号表示该位点的氨基酸。 -
[1] 马乃训, 陈光才, 袁金玲.国产竹类植物生物多样性及保护策略[J].林业科学, 2004, 43(4):103-106. MA Naixun, CHEN Guangcai, YUAN Jinling. Bamboo biodiversity and conservation strategies in China[J]. Sci Silv Sin, 2004, 43(4):103-106. [2] 王树东.浅析竹业发展与打造竹产业循环经济[J].林业科技管理, 2004(3):12-13. WANG Shudong. A discussion on the development of bamboo industry and the establishment of economic circulation of bamboo industry[J]. For Sci & Technol Manage, 2004(3):12-13. [3] PENG Zhenhua, LU Ying, LI Lubin, et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla)[J]. Nat Gen, 2013, 45(4):456-461. [4] 江泽慧.竹类植物基因组学研究进展[J].林业科学, 2012, 48(1):160-166. JIANG Zehui. Progress in bamboo genomics research[J]. Sci Silv Sin, 2012, 48(1):160-166. [5] GAO Zhimin, LI Caili, PENG Zhenhua. Generation and analysis of expressed sequence tags from a normalized cDNA library of young leaf from Ma bamboo (Dendrocalamus latiflorus Munro)[J]. Plant Cell Rep, 2011, 30(11):2045-2057. [6] CAI Zhaoming, ZHANG Yuxiao, ZHANG Lina, et al. Testing four candidate barcoding markers in temperate woody bamboos (Poaceae:Bambusoideae)[J]. J Syst Evol, 2012, 50(6):527-539. [7] ZHANG Yuxiao, ZENG Chunxia, LI Dezhu. Complex evolution in Arundinarieae (Poaceae:Bambusoideae):incongruence between plastid and nuclear GBSSI gene phylogenies[J]. Mol Phyl Evol, 2012, 63(3):777-797. [8] ZHANG Xuemei, ZHAO Lei, ZACGARY L R, et al. De Novo Sequencing and characterization of the floral transcriptome of Dendrocalamus latiflorus (Poaceae:Bambusoideae)[J]. PLoS ONE, 2012, 7(8):e42082. doi:10.1371/journal.pone.0042082. [9] SANG T. Utility of low-copy nuclear gene sequences in plantphylogenetics[J]. Crit Rev Biochem Mol Biol, 2002, 37:121-147. [10] SMALLR L, CRONN R C, WENDEL J F. Use of nuclear genes forphylogeny reconstruction in plants[J]. Aust Syst Bot, 2004, 17:145-170. [11] LI Mingai, WUNDER J, BISSOLI G, et al. Development of COS genes asuniversally amplifiable markers for phylogenetic reconstructions ofclosely related plant species[J]. Cladistics, 2008, 24:727-745. [12] ALVAREZ I, COSTA A, NIETO F G. Selecting single-copy nucleargenes for plant phylogenetics:a preliminary analysis for the Senecioneae (Asteraceae)[J]. J Mol Evol, 2008, 66:276-291. [13] WU Feinan, MUELLER L A, CROUZILLAT D, et al. Combining bioinformatics and phylogenetics to identify large sets of single-copy orthologous genes (COSⅡ) for comparative, evolutionary and systematic studies:a test case in the euasterids plant clade[J]. Theor Appl Genet, 2007, 115:747-755. [14] WU Feinan, EANNETTA N T, XU Yimin, et al. A COSⅡ genetic map of the pepper genome provides a detailed picture of synteny with tomato and newinsights into recent chromosome evolution in the genus Capsicum[J]. Theor Appl Genet, 2009, 118:1279-1293. [15] WU Feinan, EANNETTA N T, XU Yimin, et al. A detailed synteny map of the eggplant genome based on conserved ortholog set II (COSⅡ) markers[J]. Theor Appl Genet, 2009, 118:927-935. [16] FULTON T M, der HOEVEN R V, EANNETTA N T, et al. Identification, analysis, and utilization of conserved ortholog set markers for comparative genomics in higher plants[J]. Plant Cell, 2002, 14:1457-1467. [17] LIU Hailan, GUO Xiaoqin, WU Jiasheng, et al. Development of universal genetic markers based on single-copy orthologous (COSⅡ) genes in Poaceae[J]. Plant Cell Rep, 2013, 32(3):379-388. [18] MURRAY M G, THOMPSON W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Res, 1980, 8(19):4321-4325. [19] 肖国辉, 王弋, 郭小勤, 等.紫竹不同栽培类型PPO基因片段克隆及其SNP分析[J].浙江农林大学学报, 2013, 30(4):511-516. XIAO Guohui, WANG Yi, GUO Xiaoqin, et al. Cloning and SNP analysis of PPO gene among cultivars of Phyllostachys nigra[J]. J Zhejiang A & F Univ, 2013, 30(4):511-516. [20] GENMANO J, KLEIN A S. Species-specific nuclear and chloroplast single nucleotide polymorphisms to distinguish Picea glauca, P. mariana and P. rubens[J]. Theor Appl Genet, 1999, 99:37-49. [21] 张婷, 徐珞珊, 王峥涛, 等.药用植物束花石斛、流苏石斛及其形态相似种的PCR-RFLP鉴别研究[J].药学学报, 2005, 40(5):728-733. ZHANG Ting, XU Luoshan, WANG Zhengtao, et al. Molecular identification of medicinal plants:Dendrobium chrysanthum, Dendrobium fimbriatum and their morphologically allied species by PCR-RFLP analyses[J]. Acta Pharmacol Sin, 2005, 40(5):728-733. [22] 曹东伟, 蔡宇良, 杨娟, 等.中国樱桃的PCR-RFLP分析[J].西北农林科技大学学报:自然科学版, 2007, 35(5):173-178. CAO Dongwei, CAI Yuliang, YANG Juan, et al. PCR-RFLP analysis of Prunus pseudocerasus[J]. J Northwest A & F Univ Nat Sci Ed, 2007, 35(5):173-178. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2014.04.003






 下载:
下载: