-
简单序列重复(SSR)广泛分布于真核生物的核基因组,叶绿体基因组以及线粒体基因组中,常指一类以1~6个核苷酸为重复基元的串联重复DNA序列[1]。因其分布广泛、多态性高且具共显性,SSR标记被广泛开发和应用于种质资源鉴定与遗传多样性分析[2-3]。在植物中,由于叶绿体基因组表现为母系遗传,以半保留方式进行自我复制,几乎不发生重组,保守性远高于核基因组。因此,叶绿体基因组中的SSR标记(chloroplast SSR,cpSSR)可在更高分类水平上进行种间、种内鉴定和遗传进化分析[4]。随着高通量测序技术的发展,大量植物的叶绿体基因组被测序与公布,相应的叶绿体SSR也得到了广泛的开发与应用[5-7]。花椒Zanthoxylum bungeanum是芸香科Rutaceae花椒属Zanthoxylum植物,其果皮晒干后是中国传统的调味料和中草药。在市场上流通的花椒果皮主要来自于花椒Z. bungeanum(又称红花椒或大红袍花椒)和竹叶花椒Z. armatum(又称青花椒),而在日本主要利用日本花椒Z. piperitum(又称胡椒木或朝仓花椒)进行麻味物质和其他药用活性成分的提取分析[8]。在前人的研究中,基于核基因组内部序列差异开发的SCAR(sequence-related amplified polymorphism)标记[9]、ISSR(inter-SSR)标记[10]和EST-SSR(expressed sequence tags-SSR)标记[11]等已利用到花椒属植物的遗传分析中,而cpSSR标记在花椒属植物的开发与应用还未见报道。鉴于此,本研究对花椒cpSSR位点进行筛选,分析其分布特点并利用花椒、竹叶花椒和日本花椒种质资源开发多态性标记,以期为花椒属植物的种质资源鉴定、遗传进化分析等提供可靠的标记资源。
-
基于本研究前期获得的花椒叶绿体基因组序列[12],利用TRF(tandem repeats finder)在线软件(http://tandem.bu.edu/trf/trf.html)搜索单核苷酸重复序列,设置重复数≥10次;利用SSRHunter软件[13]搜索核苷酸重复数位为2,3,4,5的SSR位点,对应重复数设置为5,3,3,3次。合并2种软件的检测结果,统计1~5 bp核苷酸重复所在花椒叶绿体基因组中的位置:长单拷贝序列区(LSC)、短单拷贝序列区(SSC)和2个反向重复序列区(IR)。SSR引物利用Primer3软件()设计。原则为:扩增片段为150~250 bp;引物长度为18~22 bp;退火温度为57~63 ℃;GC质量分数为30%~55%。引物由上海生工生物工程有限公司合成。
-
本研究共选用16份花椒属种质资源进行SSR多态性的筛选,其中包括来自中国四川省和重庆市的5份竹叶花椒种质资源和1份日本花椒种质资源(均采自四川省林业科学研究院生物技术与良种研究所花椒种质圃)以及来自四川、贵州、陕西、山东和山西等5个省的10份花椒种质资源(均采自西北农林科技大学凤县花椒试验示范站)进行多态性的筛选(表 1)。所有种质资源选取3株,取鲜嫩叶片混合,硅胶干燥,常温保存备用。
表 1 供试16份花椒属种质资源来源地
Table 1. Origin of 16 germplasms of Zanthoxylum
种质资源名称 拉丁学名 来源地 ‘重庆九叶青’ Z. armatum
‘Chongqingjiuyeqing’重庆市江津区 ‘重庆荣昌无刺’ Z. armatum
‘Chongqingrongchangwuci’重庆市荣昌区 ‘蓬溪青花椒’ Z. armatum ‘Pengxiqinghuajiao’ 四川省遂宁市 ‘汉源葡萄青椒’ Z. armatum
‘Hanyuanputaoqingjiao’四川省雅安市 ‘狗椒’ Z. armatum ‘Goujiao’ 四川省雅安市 ‘汉源大红袍’ Z. bungeanum
‘Hanyuandahongpao’四川省雅安市 ‘油花椒’ Z. bungeanum ‘Youhuajiao’ 贵州省六盘水市 ‘素花椒’ Z. bungeanum ‘Suhuajiao’ 贵州省六盘水市 ‘凤县大红袍’ Z. bungeanum ‘Fengxiandahongpao’ 陕西省宝鸡市 ‘狮子头’ Z. bungeanum ‘Shizitou’ 陕西省韩城市 ‘韩城大红袍’ Z. bungeanum ‘Hanchengdahongpao’ 陕西省韩城市 ‘党村无刺’ Z. bungeanum ‘Dangcunwuci’ 陕西省韩城市 ‘府谷花椒’ Z. bungeanum ‘Fuguhuajiao’ 陕西省榆林市 ‘山东大红袍’ Z. bungeanum ‘Shandongdahongpao’ 山东省临沂市 ‘山西大红袍’ Z. bungeanum ‘Shanxidahongpao’ 山西省永济市 日本花椒 Z. piperitum 日本兵库县 供试种质资源全基因组DNA利用“植物全基因组DNA提取试剂盒(DP305)”(北京天根生物有限公司)提取,质量分数为1%琼脂糖结合紫外分光光度计检测DNA质量和浓度后稀释至20 mg·L-1,4 ℃保存备用。
-
PCR扩增体系为20 μL,包括10 μL 2× Taq Master Mix(南京诺唯赞生物科技有限公司),模板DNA(20 mg·L-1)5.0 μL,正、反向引物(2 μmol·L-1)各1.5 μL,双蒸水(ddH2O)2.0 μL。PCR扩增程序为:95 ℃预变性5 min;然后95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸8 min后4 ℃保存。取1.5 μL的PCR扩增产物,质量分数为8%的聚丙烯酰胺凝胶电泳(恒定功率100 W,100 min)分离检测,使用pBR322 DNA/MspI Marker作为标准分子量,银染拍照保存。
根据电泳图像,统计扩增条带并构建0~1数据矩阵,即有共迁移条带记为1,无条带则记为0。利用NTSYSpc-2.10软件计算遗传相似系数,采用非加权配对算术平均法(UPGMA)进行遗传相似性聚类。
-
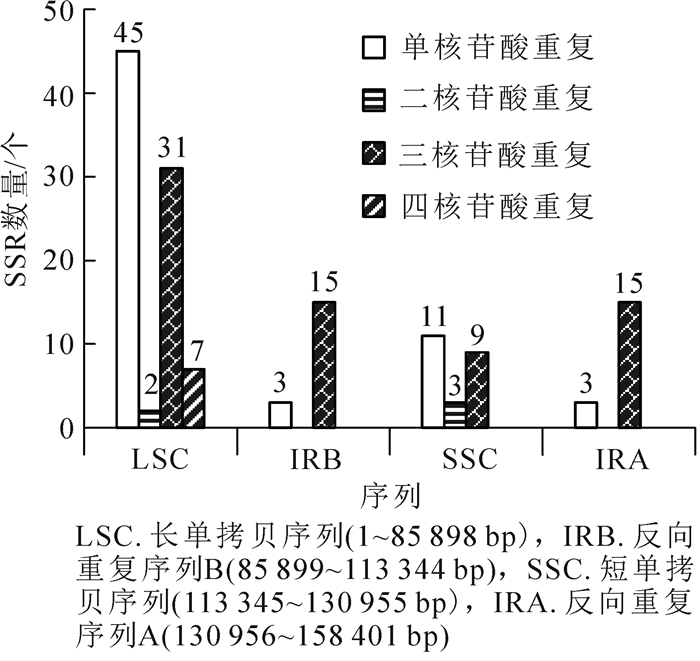
花椒叶绿体基因组全长为158 401 bp,共搜索出了144个SSR位点(图 1和表 2),即在花椒叶绿体基因组中平均每隔1 100.00 bp出现1个SSR位点。

图 1 SSR在花椒叶绿体基因组中的分布特点
Figure 1. Distribution of SSRs in the chloroplast genome of Zanthoxylum bungeanum
表 2 花椒cpSSR的重复类型与次数
Table 2. Repeat types and times of cpSSR in Zanthoxylum bungeanum
重复类型 重复单元 重复数/次 总计/次 3 4 5 6 10~15 >15 单核苷酸 A/T 52 8 60 C/G 2 0 2 二核苷酸 AT/TA 4 1 5 三核苷酸 AAC/GTT 5 5 AAG/CTT 22 3 25 AAT/ATT 20 4 1 25 ACC/GGT 2 2 ACT/AGT 2 2 AGC/CTG 4 4 AGG/CCT 2 2 ATC/ATG 5 5 四核苷酸 AAAT 2 1 3 其他类型 4 4 根据SSR位点在花椒叶绿体基因组中的位置(图 1)可知:LSC区分布85个SSR位点,占SSR总数的59.03%,平均1 010.56 bp出现1个SSR位点;SSC区分布23个SSR位点,占SSR总数的15.97%,平均765.65 bp分布1个SSR位点;在2个IR区(IRA和IRB),均分布18个反向重复的SSR位点,合计占SSR总数的25.00%,平均1 524.72 bp分布1个SSR位点。
数量上,单核苷酸重复和三核苷酸重复分别有62和70个,两者占SSR总数的91.67%,且在LSC区、SSC区和IR区均有分布。二核苷酸重复和四核苷酸重复分别只有5个(3.47%)和7个(4.86%),但二核苷酸重复仅分布于SSC区,而四核苷酸重复仅分布于LSC区。
重复次数上,单核苷酸重复次数多集中在10~15次,共54个位点,占单核苷酸重复总数的87.10%,而重复数>15次的共有8个位点;二核苷酸重复数集中在5次,三核苷酸和四核苷酸重复数集中在3次。根据重复基元类型,单核苷酸重复中A/T重复共60个,而C/G重复位点仅有2个;二核苷酸重复基元均为AT/TA;AAG/CTT和AAT/ATT为三核苷酸重复中的优势重复基元,均为25个,其余重复基元总计为20个;此外,AAAT为四核苷酸重复中的优势重复基元(3个),其余重复基元总计为4个(表 2)。
-
根据SSR位点两侧序列,由Primer3软件设计了30对引物(命名为YLT-01~YLT-30)并合成。其中,YLT-01~YLT-20扩增片段包含的是2个及2个以上的核苷酸重复位点(YLT-01~YLT-12位于LSC;YLT-14~YLT-18位于SSC区;YLT-13、19、20位于IR区),YLT-21~YLT-30扩增片段包含的是单核苷酸重复位点(YLT-21~YLT-27位于LSC区;YLT-30位于SSC区;YLT-28、29位于IR区)。
经PCR扩增筛选,30对引物均能扩增出稳定的目的片段。其中,10对引物在供试种质中呈现多态性,占筛选引物总数的33.33%。如表 3所示:10对引物共检测到24个等位基因。其中,YLT-26包含4个等位基因,YLT-21和YLT-29包含3个等位基因,其余的7个位点(YLT-01,YLT-17,YLT-22,YLT-23,YLT-24,YLT-25和YLT-27)均检测出2个等位基因。在包含2个等位基因的7个位点中,YLT-01,YLT-17,YLT-24,YLT-25和YLT-27能区分出日本花椒,花椒与竹叶花椒为一类(扩增片段大小一致),而YLT-22和YLT-23的扩增结果为:10份花椒种质同一带型,竹叶花椒与日本花椒为同一带型;YLT-21和YLT-29能将花椒、竹叶花椒和日本花椒区分开;YLT-26不仅能区分出花椒、竹叶花椒和日本花椒3个花椒属的不同种,且能区分出竹叶花椒种质中的狗椒。此外,未检测到有标记在10份花椒种质呈现多态性(图 2)。
表 3 多态性SSR引物的基本信息
Table 3. Characterizations of polymorphic SSR primers pairs
引物名称 正向引物(5′→3′) 反向引物(5′→3′) 重复单元 SSR区间位置/
bp片段长度/
bp等位基因数/个 YLT-01 GAAGGGCGTCCATTGTCTAA GAGGAAGCGGAAGCTCTTTT (ATA)3 10 940~10 948 218 2 YLT-17 TGAGACATTCCCAAATCCAA GACCGAAGCTTTTTCGAACT (TA)5…(TA)6 122 229~122 258 177 2 YLT-21 CCGTGTCAACCAATAATCCA ATCCGACTAGTTCCGGGTTC (T)16 1 931~1 946 198 3 YLT-22 CCACAAATGCATTCAGTTCC CAGGCCCGACAGAATAAAAA (A)13 7 436~7 448 177 2 YLT-23 GATTGGTTCGGTTCCAAGAA GGCAATTTGGCGAATAAAAA (A)18 7 924~7 941 215 2 YLT-24 TCGCATTGTAGCGGGTATAG TTTCGAATTGACCCTTGGAT (A)20 9 976~9 995 219 2 YLT-25 CATAGGTCATCGATTCAGCA AAGTCCAGACAAAGCACGGTA (A)15 13 422~13 436 239 2 YLT-26 AACGGAAACGAAGCGATAAA ACGGGAATCCACCAAAAAGT (A)16 28 499~28 514 248 4 YLT-27 ATCTGGGAACGGGGATTAAC CCATCAGAACGGAACGAAAT (T)17 33 511~33 527 157 2 YLT-29 AGCCACTTGTACTGCGAATTT ACGTGCGATTCGATAAAACC (T)11…(A)16 124 632~124 666 207 3 在设计的20对包含2个及2个以上的核苷酸重复位点的引物中,仅有2对引物表现出多态性;而包含单核苷酸重复位点的10对引物中,有8对引物呈现多态性,多态性比例远高于包含多核苷酸重复的位点。
-
基于10个多态性标记的检测结果,利用NTSYSpc-2.10软件对供试的16份花椒属种质进行UPGMA聚类分析。如图 2所示:在遗传相似系数阈值为0.22时,可将16份花椒种质聚为2类,即花椒和竹叶花椒为1类,日本花椒为1类;在遗传相似系数阈值为0.59时,可将花椒和竹叶花椒分为2个小类;而在遗传系数阈值为0.91时,可将竹叶花椒中的狗椒与其他4种竹叶花椒种质区分为2个小类。聚类结果与多态性标记的鉴定结果保持一致。
-
本研究在花椒的叶绿体基因组中共筛选到144个SSR位点,其中三核苷酸重复的数量要高于单核苷酸重复以及二核苷酸和四核苷酸重复的数量,与杨丽等[14]在麻竹Dendrocalamus latiflorus和绿竹Bambusa oldhamii叶绿体基因组中获得的SSR位点的规律一致,但与蒺藜苜蓿Medicao truncatula[15],棉花Gossypium hirsutum[16]和霍山石斛Dendrobium huoshanense[17]等的叶绿体基因组中均以单核苷酸重复为主不同。原则上,碱基重复基元越多,越难形成SSR位点。本研究在三核苷酸重复次数的筛选上,选择重复数大于或等于3次的位点,而大于3次的重复位点仅占三核苷酸重复总数的11.43%(8个)。因此,本研究的三核苷酸重复数量多于单核苷酸重复是由软件参数设置不同引起。此外,花椒cpSSR在LSC和SSC区的分布密度要高于IR区。在花椒叶绿体基因组中,GC质量分数在LSC区,SSC区和IR区中分别为36.87%,33.51%和42.54%[12]。由于GC间有3个氢键相连,打破GC键所需的能量要高于AT间的2个氢键,因此更难造成碱基数量的增加或减少[18]。由此推断,花椒叶绿体基因组中IR区SSR位点相对稀疏的原因与GC质量分数相关。
叶绿体基因组由于保守性高,不涉及基因重组,因此,其中突变位点也更容易保留,基于叶绿体开发的SSR标记除了保留核基因组SSR标记的特点外,其可重复性也更高,在种质资源鉴定中也更具说服力[19]。在本研究获得的10个多态性引物中,有2对引物可以把本研究所选花椒属的3个种区分开,1对引物可以将竹叶花椒种内的‘狗椒’区分出。然而,10对多态性引物均未在10份花椒种质资源中表现出多态性。由此推断,相对于竹叶花椒,花椒种内的种质资源在叶绿体基因组进化上保守性更高。
本研究开发的花椒cpSSR可作为花椒属种间鉴定的有效标记,但要应用于种内鉴定,需进一步筛选种间多态性更高的cpSSR标记或利用EST-SSR、核基因组SSR(Genomic-SSR)等保守性相对较低的标记资源进行区分。
Development of chloroplast SSR markers of Zanthoxylum bungeanum and their generality for interspecies and intraspecies
-
摘要: 叶绿体基因组为母系遗传,保守性高,因此叶绿体基因组中的简单序列重复(SSR)标记可在更高分类水平上进行种质资源鉴定和群体遗传结构分析。利用TRF软件和SSR Hunter软件相结合对花椒Zanthoxylum bungeanum叶绿体基因组中的SSR位点进行筛选。结果显示:花椒叶绿体基因组中共有144个SSR位点,位点间的平均分布距离为1 100.00 bp。基于检测软件设置参数,SSR重复类型主要集中在一、三核苷酸重复,二者占SSR位点总数的91.67%。此外,随机设计合成30对SSR引物对10份花椒种质、5份竹叶花椒Zanthoxylum armatum种质和1份日本花椒Zanthoxylum piperitum种质进行PCR扩增检测,共筛选出10对多态性引物,其中单核苷酸重复位点的多态性高于多核苷酸重复位点。本研究开发的叶绿体SSR标记可有效区分花椒、竹叶花椒和日本花椒,但在花椒种内并未检测出多态性。表明花椒叶绿体基因组的SSR标记可用于花椒属Zanthoxylum不同种间的遗传多样性分析。
-
关键词:
- 植物学 /
- 花椒 /
- 叶绿体基因组 /
- 简单序列重复(SSR) /
- 多态性分析
Abstract: Simple sequence repeat (SSR) molecular markers in the chloroplast genome can be used for germplasm resource identification and population genetic structural analysis at a higher classification level due to the chloroplast genome being of maternal inheritance and high conservation. To screen and develop SSR markers which will be useful for genetic diversity analysis, fingerprinting and germplasms resource identification in the chloroplast genome of Zanthoxylum bungeanum, a traditional spice plant in China, TRF software and SSR Hunter were used to screen the mono-, di-, tri- and tetra-nucleotide repeat motifs and 16 germplasms of Zanthoxylum including 10 individuals of Z. bungeanum, 5 individuals of Zanthoxylum armatum, and 1 individual of Zanthoxylum piperitum were selected to test polymorphism of these detected loci. As a result, 144 SSR loci were identified with an average distribution distance of 1 100.00 bp in the chloroplast genome of Z. bungeanum. Based on parameters set by the detection software, mono-nucleotide and tri-nucleotide repeat motifs were the main repeat types accounting for 91.67% of the total. In addition, 30 SSR primer pairs were randomly designed and synthesized with 10 of them showing polymorphism. The polymorphic markers of mono-nucleotide repeat motifs were more than that of polynucleotide repeat motifs in this study. Furthermore, these cpSSR markers could effectively divide Z. bungeanum, Z. armatum, and Z. piperitum, but no polymorphic marker was found in the 10 individuals of Z. bungeanum. Thus, cpSSR markers of Z. bungeanum could be used for genetic diversity analysis among different interspecies of Zanthoxylum.-
Key words:
- botany /
- Zanthoxylum bungeanum /
- chloroplast genome /
- SSR /
- polymorphism analysis
-
表 1 供试16份花椒属种质资源来源地
Table 1. Origin of 16 germplasms of Zanthoxylum
种质资源名称 拉丁学名 来源地 ‘重庆九叶青’ Z. armatum
‘Chongqingjiuyeqing’重庆市江津区 ‘重庆荣昌无刺’ Z. armatum
‘Chongqingrongchangwuci’重庆市荣昌区 ‘蓬溪青花椒’ Z. armatum ‘Pengxiqinghuajiao’ 四川省遂宁市 ‘汉源葡萄青椒’ Z. armatum
‘Hanyuanputaoqingjiao’四川省雅安市 ‘狗椒’ Z. armatum ‘Goujiao’ 四川省雅安市 ‘汉源大红袍’ Z. bungeanum
‘Hanyuandahongpao’四川省雅安市 ‘油花椒’ Z. bungeanum ‘Youhuajiao’ 贵州省六盘水市 ‘素花椒’ Z. bungeanum ‘Suhuajiao’ 贵州省六盘水市 ‘凤县大红袍’ Z. bungeanum ‘Fengxiandahongpao’ 陕西省宝鸡市 ‘狮子头’ Z. bungeanum ‘Shizitou’ 陕西省韩城市 ‘韩城大红袍’ Z. bungeanum ‘Hanchengdahongpao’ 陕西省韩城市 ‘党村无刺’ Z. bungeanum ‘Dangcunwuci’ 陕西省韩城市 ‘府谷花椒’ Z. bungeanum ‘Fuguhuajiao’ 陕西省榆林市 ‘山东大红袍’ Z. bungeanum ‘Shandongdahongpao’ 山东省临沂市 ‘山西大红袍’ Z. bungeanum ‘Shanxidahongpao’ 山西省永济市 日本花椒 Z. piperitum 日本兵库县 表 2 花椒cpSSR的重复类型与次数
Table 2. Repeat types and times of cpSSR in Zanthoxylum bungeanum
重复类型 重复单元 重复数/次 总计/次 3 4 5 6 10~15 >15 单核苷酸 A/T 52 8 60 C/G 2 0 2 二核苷酸 AT/TA 4 1 5 三核苷酸 AAC/GTT 5 5 AAG/CTT 22 3 25 AAT/ATT 20 4 1 25 ACC/GGT 2 2 ACT/AGT 2 2 AGC/CTG 4 4 AGG/CCT 2 2 ATC/ATG 5 5 四核苷酸 AAAT 2 1 3 其他类型 4 4 表 3 多态性SSR引物的基本信息
Table 3. Characterizations of polymorphic SSR primers pairs
引物名称 正向引物(5′→3′) 反向引物(5′→3′) 重复单元 SSR区间位置/
bp片段长度/
bp等位基因数/个 YLT-01 GAAGGGCGTCCATTGTCTAA GAGGAAGCGGAAGCTCTTTT (ATA)3 10 940~10 948 218 2 YLT-17 TGAGACATTCCCAAATCCAA GACCGAAGCTTTTTCGAACT (TA)5…(TA)6 122 229~122 258 177 2 YLT-21 CCGTGTCAACCAATAATCCA ATCCGACTAGTTCCGGGTTC (T)16 1 931~1 946 198 3 YLT-22 CCACAAATGCATTCAGTTCC CAGGCCCGACAGAATAAAAA (A)13 7 436~7 448 177 2 YLT-23 GATTGGTTCGGTTCCAAGAA GGCAATTTGGCGAATAAAAA (A)18 7 924~7 941 215 2 YLT-24 TCGCATTGTAGCGGGTATAG TTTCGAATTGACCCTTGGAT (A)20 9 976~9 995 219 2 YLT-25 CATAGGTCATCGATTCAGCA AAGTCCAGACAAAGCACGGTA (A)15 13 422~13 436 239 2 YLT-26 AACGGAAACGAAGCGATAAA ACGGGAATCCACCAAAAAGT (A)16 28 499~28 514 248 4 YLT-27 ATCTGGGAACGGGGATTAAC CCATCAGAACGGAACGAAAT (T)17 33 511~33 527 157 2 YLT-29 AGCCACTTGTACTGCGAATTT ACGTGCGATTCGATAAAACC (T)11…(A)16 124 632~124 666 207 3 -
[1] NAVASCUES M, EMERSON B C. Chloroplast microsatellites:measures of genetic diversity and the effect of homoplasy[J]. Mol Ecol, 2005, 14(5):1333-1341. [2] 李军, 董彬, 张超, 等.桂花EST-SSR引物开发及在品种鉴定中的应用[J].浙江农林大学学报, 2018, 35(2):306-313. LI Jun, DONG Bin, ZHANG Chao, et al. EST-SSR primers and their application in cultivar identification of Osmanthus fragrans[J]. J Zhejiang A&F Univ, 2018, 35(2):306-313. [3] WEN Mengfu, WANG Haiyan, XIA Zhiqiang, et al. Developmenrt of EST-SSR and genomic-SSR markers to assess genetic diversity in Jatropha curcas L.[J]. BMC Res Notes, 2010, 3(1):42. [4] 薛建华, 姜莉, 马晓林, 等.莲品种DNA指纹图谱的构建[J].生物多样性, 2016, 24(1):3-11. XUE Jianhua, JIANG Li, MA Xiaolin, et al. Identification of lotus cultivars using DNA fingerprinting[J]. Biodiversity Sci, 2016, 24(1):3-11. [5] ASAF S, WAQAS M, KHAN A L, et al. The complete chloroplast genome of wild rice (Oryza minuta) and its comparison to related species[J]. Front Plant Sci, 2017, 8:304. [6] WANG Yanling, XIE Hongxian, YANG Yi, et al. Chloroplast and mitochondrial microsatellites for Millettia pinnata (Fabaceae) and cross-amplification in related species[J]. Appl Plant Sci, 2017, 5(5):1700034. doi:10.3732/apps.1700034. [7] MOHAMMAD-PANAH N, SHABANIAN N, KHADIVI A, et al. Genetic structure of gall oak (Quercus infectoria) characterized by nuclear and chloroplast SSR markers[J]. Tree Genet Genomes, 2017, 13(3):70. [8] YAMAZAKI E, INAGAKI M, KURITA O, et al. Antioxidant activity of Japanese pepper (Zanthoxylum piperitum D C.) fruit[J]. Food Chem, 2007, 100:171-177. [9] FENG Shijing, YANG Tuxi, LIU Zhenshan, et al. Genetic diversity and relationships of wild and cultivated Zanthoxylum germplasms based on sequence-related amplified polymorphism (SRAP) markers[J]. Genet Resour Crop Evol, 2015, 62:1193-1204. [10] FENG Shijing, YANG Tuxi, LI Xiao, et al. Genetic relationships of Chinese prickly ash as revealed by ISSR markers[J]. Biologia, 2015, 70(1):45-51. [11] 候丽秀, 魏安智, 王丽华, 等.花椒转录组SSR信息分析及其分子标记开发[J].农业生物技术学报, 2018, 26(7):1226-1236. HOU Lixiu, WEI Anzhi, WANG Lihua, et al. Analysis of SSR loci and development of molecular markers in Zanthoxylum bungeanum transcriptome[J]. J Agric Biotechnol, 2018, 26(7):1226-1236. [12] LIU Yulin, WEI Anzhi. The complete chloroplast genome sequence of an economically important plant, Zanthoxylum bungeanum (Rutaceae)[J]. Conserv Genet Resour, 2016, 9(1):25-27. [13] 李强, 万建民. SSR Hunter, 一个本地化的SSR位点搜索软件的开发[J].遗传, 2005, 27(5):808-810. LI Qiang, WAN Jianmin. SSR Hunter:development of a local searching software for SSR sites[J]. Hereditas, 2005, 27(5):808-810. [14] 杨丽, 赵韩生, 彭镇华, 等.竹子叶绿体基因组SSR分子标记的开发及其应用[J].热带亚热带植物学报, 2014, 22(3):263-269. YANG Li, ZHAO Hansheng, PENG Zhenhua, et al. Development and application of SSR molecular markers from the chloroplast genome of bamboo[J]. J Trop Subtrop Bot, 2014, 22(3):263-269. [15] 骈瑞琪, 李伟, 李娜, 等.蒺藜苜蓿叶绿体微卫星分布规律的研究[J].安徽农业科学, 2008, 36(9):3531-3534. PIAN Ruiqi, LI Wei, LI Na, et al. Distribution of microsatellite from complete sequence of Medicao truncatula chloroplast DNA[J]. J Anhui Agric Sci, 2008, 36(9):3531-3534. [16] 蔡小彦, 刘方, 周忠丽, 等.棉花叶绿体基因组全序列微卫星分布规律研究[J].棉花学报, 2015, 27(6):570-575. CAI Xiaoyan, LIU Fang, ZHOU Zhongli, et al. Distribution of microsatellites from the complete sequence of Gossypium hirsutum chloroplast DNA[J]. Cotton Sci, 2015, 27(6):570-575. [17] 刘枫, 赵群, 戴军, 等.霍山石斛cpDNA全序列微卫星分布及分子鉴别研究[J].中药材, 2016, 39(12):2720-2724. LIU Feng, ZHAO Qun, DAI Jun, et al. Microsatellite analysis of cpDNA in Dendrobium huoshanense and its molecular identification[J]. J Chin Med Mater, 2016, 39(12):2720-2724. [18] 童治军, 肖炳光. 3种烟草基因组SSR位点信息分析和标记开发[J].西北植物学报, 2014, 34(8):1549-1558. TONG Zhijun, XIAO Bingguang. Survey of SSR loci information in three tobacco genomes and development of SSR markers[J]. Acta Bot Boreali-Occident Sin, 2014, 34(8):1549-1558. [19] PROVAN J, POWELL W, HOLLINGSWORTH P M. Chloroplast microsatellites:new tools for studies in plant ecology and evolution[J]. Trends Ecol Evol, 2001, 16(3):142-147. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2019.06.023






 下载:
下载: