-
Basic helix-loop-helic (bHLH)转录因子是一类在生物界广泛存在的转录因子家族,通过特定的氨基酸残基与靶基因相互作用,进而调节相关基因的表达[1]。依据进化关系及与DNA的结合模式,将动物中bHLH转录因子分为A~F等6个家族,植物bHLH蛋白大多属于B组[2]。拟南芥Arabidopsis thaliana中有147个bHLH基因,水稻Oryza sativa中有167个bHLH转录因子[3]。CARRETERO-PAULE等[3]对拟南芥、水稻、苔藓和藻类全基因组的bHLH转录因子进行了系统分析,这4类植物中所有638个bHLH基因被分为32个亚家族,主要特征是含有约60个氨基酸的bHLH结构域,可以分为长度15个氨基酸左右的碱性氨基酸区和45个氨基酸左右的α螺旋-环-α螺旋区(HLH区)[4],并且定义了植物bHLH的保守区域,即碱性氨基酸区含有高度保守的H5-E9-R13序列(His5-Glu9-Arg13),这为鉴定新的bHLH转录因子提供了依据[3]。bHLH基因参与植物的生长发育,促进花形态的建成和抑制及种子的萌发,控制气孔细胞分化及保卫细胞形成[5]。bHLH还参与植物适应非生物逆境反应[6]。拟南芥中AtbHLH92表达受盐、干旱、渗透和寒冷等多种胁迫强烈诱导,陆地棉Gossypium hirsutum中分离的GhbHLH1被ABA,干旱和盐胁迫诱导表达[7];过表达水稻OsbHLH001转录因子的拟南芥抗盐及抗冻能力均得到的提高[1]。之外,bHLH还参与生物合成及信号转导。研究发现,bHLH类基因OSB1,ATR2和IND分别参与花青素、色氨酸及赤霉素的合成;PIF3,PIF4和HFR1在光敏色素信号传导中起调控作用;BEE1,BEE2和BEE3在油菜素内酯(brassinolid,BR)的信号传导中起作用[8]。BEE(BR enhanced expression)所编码的蛋白质是属于bHLH超家族的一员,是调控BR信号转导的重要元件[8]。迄今为止对BEE基因的研究积累较少。在拟南芥中,3个BEE基因在BR信号途径中是早期应答受体[8]。拟南芥BEE基因突变体出现植株矮小和开花延迟的表型,且该家族成员存在功能冗余[8]。在木本植物中BEE基因的功能研究未见报道。白桦Betula platyphylla是桦木科Betulaceae桦木属Betula的落叶乔木,天然次生林更新的先锋树种,也是中国重要的经济树种之一。油菜素内酯是对植物生长发育有多方面调节作用的植物激素,对植物生长发育有着多方面的重要影响。本研究以白桦为材料,克隆了3条BpBEE基因,通过生物信息学及实时定量聚合酶链式反应技术,对BpBEE在白桦不同生长阶段的表达变化及响应BR处理的情况进行了研究,旨在深入揭示BpBEE基因功能,探索它在木本植物BR信号途径的作用。
-
基因表达分析材料采自东北林业大学林木遗传育种试验基地10年生的白桦。雌花序出现于4月中下旬, 始花5~10 d后开始受粉, 持续1周左右,雄花序出现于5月底, 8月中旬结束。白桦的雄配子体发育很特殊, 在当年不形成成熟的雄配子体, 直到第2年4月中下旬才发育成熟,称为越冬宿存现象[9]。2013年6月5日至9月20日,隔15 d取材1次,每次均选取相同3株白桦中部的雄花序、叶片、嫩茎和顶芽,分别混样。越冬后第2年4月15日至4月25日,隔5 d采取雄花序和雌花序。植物材料经液氮速冻后,放入-80℃超低温冰箱保存备用。
BR处理以1年生、长势一致的白桦无性系为材料。喷洒0.2 mg·L-1的BR (含质量分数为0.1%的吐温80)溶液5.0 mL,同时设置对照(喷洒质量分数为0.1%吐温80溶液5.0 mL),在处理0,2,4,6,12,24,48和72 h后,分别采集顶芽、叶、木质部和韧皮部。液氮冷冻处理,-80℃冰箱中保存备用。
-
根据研究团队已有的白桦不同组织转录组数据,获得3个BEE基因的全长cDNA序列,分别命名为BpBEE1,BpBEE2,BpBEE3。
利用美国生物技术信息中心(NCBI)在线开放阅读框(ORF)寻找程序确定基因的开放读码框;利用Conserved Domains工具()预测保守区及蛋白质的基本结构域。对蛋白质的分子量和理论等电点进行了在线计算[http://web.expasy.org/protparam/。根据白桦基因组DNA序列(白桦基因组测序、拼接工作已经完成,数据尚未公布)在GSDS (]上对其基因组序列进行外显子和内含子的预测。用BioEdit中的ClustalW进行氨基酸的多序列比对;选取白桦和拟南芥AtBEE基因序列,使用MEGA 5.1以默认参数构建邻接树(neighbor-joining tree,NJ-tree),其中Bootstrap分析进行1 000次重复。
-
采用RNA提取试剂盒(北京百泰克生物技术有限公司,RP3302),按说明书提取白桦不同组织部位的总RNA。用微量紫外检测仪NanoDrop测定提取的RNA样品浓度,用质量分数为1.2%琼脂糖凝胶电泳检测RNA质量。
-
利用PrimeScriptTM RT reagentkit (TaKaRa)进行RNA反转录,反应体系和条件参照试剂盒说明书操作。
-
根据已知的3个BpBEE全长序列设计实时定量引物,同时以白桦α-Tubulin为内标基因(引物序列参见表 1)。以获得的白桦不同生长阶段材料(雌花、雄花、茎、叶和顶芽)及BR处理后不同时间(0,2,4,6,12,24,48和72 h)收集的组织(顶芽、叶、木质部和韧皮部) cDNA稀释10倍作为模板。荧光定量反应体系为SYBR Premix Ex Taq10.0 μL,上、下游引物(10 mmol·L-1)各0.8 μL,Rox DyeⅡ0.4 μL,cDNA 2.0 μL,加水至终体积20.0 μL。反应在ABI PRISM 7500实时定量聚合酶链式反应(PCR)仪上进行,程序为95℃预变性30 s,95℃变性5 s,60℃退火延伸34 s,循环40次,绘制溶解曲线,温度由95℃ 15 s,60℃ 1 min,至95℃ 15 s止,重复3次·反应-1,采用-ΔΔCt算法分析结果。
表 1 定量RT-PCR分析所用引物
Table 1. Primers used in RT-PCR analysis
基因 上游引物序列(5'→3') 下游引物序列(5'→3') BpBEE1 CAGCCAAATGCAAGCATGATGG GGAGCTGGTTGTTGATAGTTCC BpBEE2 GCTAGACAAGCTTCATCAACC TTCCATCTCCTCCACCACC BpBEE3 GTTCAACCAGAATGTGGTGC CTGTGACTGTCAGTAGCTTGG α-Tubulin GCACTGGCCTCCAAGGAT TGGGTCGCTCAATGTCAAGG -
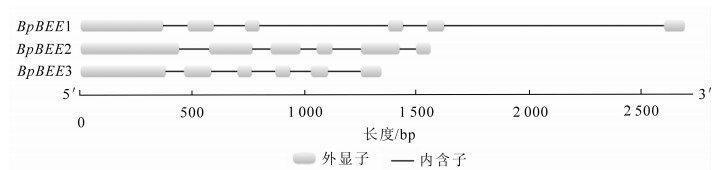
根据白桦基因组信息,对3个基因的基因组序列进行分析,结果BpBEE1,BpBEE2及BpBEE3基因的基因组序列长度差异较大,其中BpBEE1基因组序列最长,达2 700 bp,BpBEE2和BpBEE3基因分别为1 600 bp和1 400 bp,但3个基因的内含子和外显子数目一致,均含有6个外显子5个内含子,且BpBEE1的5个内含子长度(110,140,574,106和982 bp)均比BpBEE2的内含子(134,80,72,127和73 bp)长,BpBEE3内含子长度依次为82,11,104,91和146 bp (图 1)。BpBEE1和BpBEE3编码的氨基酸数目接近,分别为263和269个氨基酸,BpBEE2编码的氨基酸序列较长,为360个氨基酸,理论等电点的范围为5.3~6.5(表 2)。
表 2 3个BpBEE基本理化性质
Table 2. Basic physico-chemical properties of BpBEEs genes
基因 5'UTR/bp 3'UTR/bp ORF长度/bp 氨基酸数目 相对分子质量/kD 理论等电点 BpBEE1 76 407 792 263 29.9 6.42 BpBEE2 73 411 1 083 360 39.9 5.31 BpBEE3 30 167 810 269 30.2 5.93 美国生物技术信息中心BLAST序列分析表明,BpBEE1,BpBEE2和BpBEE3所编码的蛋白具有明显的bHLH蛋白质结构域,包括2个螺旋和1个环状结构,含有特异识别序列E-box (CANNTG)和N-box (CACGC/AG)。多重序列比对结果表明:白桦BpBEE基因所编码的bHLH结构域与bHLH氨基酸序列中的严格保守的位点完全一致,包括碱性结构域中第9位组氨酸,第13位谷氨酸与第16位、第17位的精氨酸,螺旋区域中第10位的亮氨酸(图 2)。白桦BpBEE2与拟南芥AtBEE2基因的氨基酸同源性高达87%,BpBEE3与拟南芥AtBEE1和AtBEE3基因的氨基酸同源性高达为90%,BpBEE1也与拟南芥中AtBEE1和AtBEE3基因的氨基酸同源性达到55%。分子进化树表明,白桦BpBEE1,AtBEE2,AtBEE3基因聚为一类,均与拟南芥AtBEE基因同源性较高。根据已有的bHLH转录因子分类情况,白桦BpBEE基因在bHLH蛋白家族中属于第25亚类(图 3)。
-
利用实时定量PCR技术对3个BpBEE基因在不同时间和不同组织中的表达模式进行分析。结果显示:雌花中(参考组织为2014年4月15日BpBEE1表达量),BpBEE1基因随着雌花的发育表达量逐渐降低,在4月25日达到最低值,下调2-1.8倍。BpBEE2基因呈现上调表达趋势,但是表达量逐渐减少。而BpBEE3呈现先下调后上调的趋势,下调最低峰出现在4月20日(2-2.0倍),而后显著上调(24.0倍)(图 4)。
在雄花发育初期,3个BpBEE的表达丰度下调或不变(参照组织为2014年4月15日BpBEE1基因表达量),在发育中期(7月5日到8月5日)BpBEE2和BpBEE3基因上调表达,上调倍数在22.0~24.0,而越冬后3个基因均成下调表达,尤其以BpBEE3最为明显(图 4)。
茎中3个基因的表达趋势不尽相同(参考组织为2013年6月5日BpBEE1基因表达量)。BpBEE1在发育初期呈现上调趋势,到发育中期依然上调表达但表达量有所减少,到8月20日出现下调最低峰(2-1.0倍),而后显著上调(23.0倍)。BpBEE2基因呈现持续上调趋势,在7月5日出现上调最高峰(25.0倍),随后仍呈现上调表达但是表达量逐渐减少。而BpBEE3基因在整个发育阶段大部分呈现上调趋势,但是变化量小,在6月20日达到下调最低峰(2-1.8倍),在9月5日出现了明显的上调趋势(23.0倍)(图 4)。
在叶中,BpBEE1和BpBEE2基因表达量随着生长持续上调(参考组织为2013年6月5日BpBEE1基因表达量),8月20日达到最高值(27.0~28.0倍),9月5日表达量显著减少。BpBEE3在发育初期呈现下调趋势,随后持续上调(24.5~24.8倍),在9月5日达到下调最低峰(2-2.5倍)(图 4)。
在顶芽中,BpBEE1基因在发育初期表达量较低(参考组织为2013年6月5日BpBEE1基因表达量),在发育的旺盛时期7月5日达到最高峰(22.0倍)随后表达量逐渐降低,在9月5日达到最低点。BpBEE2和BpBEE3的表达规律是相同的,发育初期下调表达,发育的旺盛时期上调表达,发育后期表达量减少甚至下调表达。这表明基因对于顶芽的发育有影响。
-
利用实时定量PCR技术分析了BpBEE1,BpBEE2和BpBEE3对BR激素的响应。结果显示:在芽中BpBEE2和BpBEE3的表达趋势相似,4~48 h均为下调表达(不同组织以0 h取材的BpBEE1基因的表达量为参照),分别在6 h和24 h达到下调的最低值(2-2.0和2-4.5倍)。而BpBEE1与其相反,早期(2 h和4 h)上调表达,在中后期下调表达,上调表达最高值在处理2 h出现(21.8倍)(图 5),这与拟南芥中AtBEE基因是BR信号通路中的早期应答受体的结果相符合[8]。
在叶中,3个BpBEE基因的表达趋势相同,表达丰度均低于处理前。BpBEE1在早期(6 h)表现为下调表达,在中后期的表达呈现波动状,但是在20和2-2.0倍之间波动。BpBEE2出现了较为明显的下调表达,并在48 h时出现了最低值(2-2.0倍)。BpBEE3的表达呈现下调,在6 h达到了最低值(2-6.0倍)(图 5)。
在木质部中,BpBEE1在BR处理后表达量升高,BpBEE1在4 h达到了最高值(22.0倍)。BpBEE2在处理后呈现上调表达,但是表达量均低于处理前。BR处理后BpBEE3下调表达,最低值出现在6 h (2-4.5倍)。
在韧皮部中,BpBEE1和BpBEE3基因表达趋势相似,均为下调表达,分别在48 h和6 h时达到表达最低值(2-3.0倍和2-5.5倍),而BpBEE2在BR处理0 h时为上调表达,处理后呈现下调表达趋势,24 h达到最低值(2-4.0倍)(图 5)。
以上结果表明:BR激素影响BpBEE基因的表达量,在不同组织部位的影响不同。
-
真核生物中bHLH蛋白是一大类转录因子,已在多种生物的基因组中得到鉴定[10]。植物中bHLH家族成员数量众多,仅次于MYB类转录因子家族,在拟南芥中bHLH转录因子超过140个,水稻中则多于160个[5]。根据白桦基因组和转录组数据,分析预测白桦中含有的bHLH转录因子有157个。bHLH蛋白主要特征是含有bHLH结构域,白桦BpBEE均含有62个氨基酸组成的bHLH结构域,并存在高度保守的序列[11],对bHLH与靶DNA的结合密切相关。同时通过序列分析BpBEE能识别E-box (CANNTG)并结合,进而激发基因进行转录[2]。
在进化上,一个保守结构域的内含子/外显子的剪切位点也是十分保守的,所以它对于基因家族的进化研究也是非常重要的证据[12]。在植物的bHLH蛋白超家族中,其内含子的位置十分保守,但数量具有多样化的特点,最常见的是含有2或3个内含子[12],也有个别基因含有更多数目的内含子。例如,拟南芥AtBEE基因含有4~5个内含子和5~6个外显子[11]。白桦BpBEE基因含有的内含子数目一致,均含有5个内含子,故在亲缘关系上比较相近。
进化树分析发现,BpBEE1,BpBEE2和BpBEE3属于bHLH转录因子的第25亚类,与拟南芥中AtBEEs同源性较高[13]。已有研究报道,拟南芥第25亚类中共有17个成员,其中包括AtBEE1,AtBEE2,AtBEE3和AtCIB1基因,这些基因参与激素信号途径和器官的发育[11]。AtBEEs基因表达受BR和脱落酸(ABA)的诱导,在喷洒植物生长调节物质BR后,AtBEE基因的表达量明显高于未处理前,拟南芥幼苗的胚轴也有一定程度的增长;而ABA的效果与BR相反,在喷洒植物生长调节物质后AtBEE基因的表达量降低[8]。AtCIB1基因所编码的蛋白质可以调控光诱导基因的表达,从而控制植物的成花转变[14]。拟南芥隐花色素CRY2与bHLH转录因子CIB1发生互作,CIB1能够和成花素FT基因启动子区的E-box (CANNTG)结合,从而促进FT表达,而FT编码一个可移动的转录因子,从叶片转移到茎端分生组织促进开花基因的表达,最终促进光周期控制的开花[15]。在本研究中,3个BpBEE基因在不同组织部位的表达呈现出一定程度的相似性,预测其在功能上有一定的冗余,但也有很多不同点。在生长旺盛时期,3个BpBEE基因在叶、顶芽和茎中均呈现上调表达;在雄花的减数分裂时期,BpBEE2和BpBEE3基因呈现上调表达,故我们认为白桦BpBEE基因参与到了植物的生长发育过程中。在进行BR激素处理之后,3个BpBEE基因的表达量都明显的变化,在处理早期0~6 h期间,BpBEE1和BpBEE2基因的表达量有明显的变化,表明BpBEE基因参与对BR激素应答,BpBEE1与BpBEE2表现为早期应答反应。在拟南芥中AtBEEs基因作为转录因子调控多个基因,例如BRI1,BIMs,BIN2和BZR2基因的表达从而调节BR信号途径[16]。本研究的3个基因与其同源性较高,BpBEE基因是否具有相同的调节功能,及对下游基因及表达调控机制仍然需要进一步研究揭示。
Cloning and expression of three BpBEE genes in Betula platyphylla
-
摘要: 根据已有的白桦Betula platyphylla茎尖、叶片及木质部等45个转录组信息,获得3条白桦BEE基因的全长cDNA序列,分别命名为BpBEE1,BpBEE2和BpBEE3。对3个BpBEE基因进行了生物信息学分析和实时定量聚合酶链式反应分析,结果表明:3个基因具有典型的结合DNA及特异识别序列E-box和N-box,属于bHLH类转录因子。这3个基因与拟南芥Arabidopsis thaliana中AtBEE基因亲缘关系较近,属于bHLH超家族第25亚类。在白桦生长季内,BpBEE1,BpBEE2和BpBEE3在不同组织中均有表达,叶中BpBEE基因表达量明显高于其他部位。在生长旺盛时期,茎、叶和顶芽中BpBEE基因均呈现上调表达,表明BpBEE基因可能参与到白桦的生长过程。油菜素内酯(BR)激素处理之后,BpBEE1在早期(2 h和4 h)的芽和木质部中呈显著上调(>2倍);BpBEE2在早期(2 h)的木质部和芽显著上调外,其他处理时间也呈现不同程度的上调。表明3个白桦BpBEE基因参与BR激素应答,且BpBEE1和BpBEE2呈现早期应答响应。Abstract: Based on the transcriptome sequences from 45 libraries of stem tips, leaves, and xylem of Betula platyphylla (birch), three BEE genes (BpBEE1, BpBEE2, and BpBEE3), were obtained bioinformatics and expression patterns of three genes were analyses. Results showed that three BpBEE genes belonged to basic helix-loop-helix (bHLH) transcription factor superfamily, and contained a typical DNA binding domain and E-box/N-box specificity sites. These genes were highly homologous to AtBEE genes, and belonged to the 25th subclass in the bHLH superfamily. During the growing season, the RNA level of all three BpBEE genes were highestin leaves than in others tissues. In the vigorous growing stage, All BpBEE genes were up-regulated in leaves and buds. With the brassinosteroids (BR) treatment, BpBEE1 was up-regulated in buds and xylem (>2 times) in the early stages (2 h and 4 h); whereas, BpBEE2 was up-regulated in the xylem and buds in the early stage (2 h). These results suggested that the three BpBEE genes could be involved in the growth process of birch, and response for BR hormone, BpBEE1 and BpBEE2 showeds early reply responses, especially.
-
表 1 定量RT-PCR分析所用引物
Table 1. Primers used in RT-PCR analysis
基因 上游引物序列(5'→3') 下游引物序列(5'→3') BpBEE1 CAGCCAAATGCAAGCATGATGG GGAGCTGGTTGTTGATAGTTCC BpBEE2 GCTAGACAAGCTTCATCAACC TTCCATCTCCTCCACCACC BpBEE3 GTTCAACCAGAATGTGGTGC CTGTGACTGTCAGTAGCTTGG α-Tubulin GCACTGGCCTCCAAGGAT TGGGTCGCTCAATGTCAAGG 表 2 3个BpBEE基本理化性质
Table 2. Basic physico-chemical properties of BpBEEs genes
基因 5'UTR/bp 3'UTR/bp ORF长度/bp 氨基酸数目 相对分子质量/kD 理论等电点 BpBEE1 76 407 792 263 29.9 6.42 BpBEE2 73 411 1 083 360 39.9 5.31 BpBEE3 30 167 810 269 30.2 5.93 -
[1] 刘晓月, 王文生, 傅彬英, 等.植物bHLH转录因子家族的功能研究进展[J].生物技术进展, 2011, 1(6):391-397. LIU Xiaoyue, WANG Wensheng, FU Binying, et al. Research progress of plant bHLH transcription factor family[J]. Curr Biotechnol, 2011, 1(6):391-397. [2] 王勇, 陈克平, 姚勤. bHLH转录因子家族研究进展[J].遗传, 2008, 30(7):821-830. WANG Yong, CHEN Keping, YAO Qin. Progress of studies on bHLH transcription factor families[J]. Hereditas, 2008, 30(7):821-830. [3] CARRETERO-PAULET L, GALSTYAN A, ROIG-VILLANOVA I, et al. Genome-wide classification and evolutionary analysis of the bHLH family of transcription factors in arabidopsis, poplar, rice, moss, and algae[J]. Plant Physiol, 2010, 153(3):1398-1412. [4] 杨鹏程, 周波, 李玉花.植物花青素合成相关的bHLH转录因子[J].植物生理学报, 2012, 48(8):747-758. YANG Pengcheng, ZHOU Bo, LI Yuhua. The bHLH transcription factors involved in anthocyanin biosynthesis in plants[J]. Plant Physiol J, 2012, 48(8):747-758. [5] 刘文文, 李文学.植物bHLH转录因子研究进展[J].生物技术进展, 2013, 3(1):7-11. LIU Wenwen, LI Wenxue. Progress of plant bHLH transcription factor[J]. Curr Biotechnol, 2013, 3(1):7-11. [6] 刘文文. bHLH122提高植物抗逆能力的分子机制初探[D].北京:中国农业科学院, 2013. LIU Wenwen. Molecular Mechanism Study of bHLH122 in Improving Plant Tolerance to Stresses[D]. Beijing:Chinese Academy of Agricultural Sciences, 2013. [7] 王艳敏, 白卉, 曹焱. bHLH转录因子研究进展及其在植物抗逆中的应用[J].安徽农业科学, 2015, 43(21):34-35. WANG Yanmin, BAI Hui, CAO Yan. Research progress of bHLH transcription factor and application in plant abiotic stress tolerance[J]. J Anhui Agric Sci, 2015, 43(21):34-35. [8] FRIEDRICHSEN D M, NEMHAUSER J, MURAMITSU T, et al. Three redundant brassinosteroid early response genes encode putative bHLH transcription factors required for normal growth[J]. Genetics, 2002, 162(3):1445-1456. [9] 刘雪梅, 杨传平.白桦雌雄花发育周期的时序特征[J].林业科学, 2006, 42(12):28-32. LIU Xuemei, YANG Chuanping. Temporal characteristics of developmental cycles of remale and male flowers in Betula platyphylla in northeastern China[J]. Sci Silv Sin, 2006, 42(12):28-32. [10] 张全琪, 朱家红, 倪燕妹, 等.植物bHLH转录因子的结构特点及其生物学功能[J].热带亚热带植物学报, 2011, 19(1):84-90. ZHANG Quanqi, ZHU Jiahong, NI Yanmei, et al. The structure and function of plant bHLH transcription factor[J]. J Trop Subtrop Bot, 2011, 19(1):84-90. [11] TOLEDOORTIZ G, HUQ E, QUAIL P H. The Arabidopsis basic/helix-loop-helix transcription factor family[J]. Plant Cell, 2003, 15(8):1749-1770. [12] 段学鹏.水稻和拟南芥基因组bHLH转录因子超家族生物信息学研究[D].上海:上海大学, 2006. DUAN Xuepeng. Genome-Wide Bioinformatics Research of bHLH Transcription Factor Superfamily in Rice and Arabidopsis thaliana[D]. Shanghai:University of Shanghai, 2006. [13] LI Xiaoxing, DUAN Xuepeng, JIANG Haixiong, et al. Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice and Arabidopsis[J]. Plant Physiol, 2006, 141(4):1167-1184. [14] 杨立文, 付建新, 亓帅, 等.高等植物开花时间的蓝光调控:隐花色素介导的光信号传导[J].分子植物育种, 2015, 13(2):450-460. YANG Liwen, FU Jianxin, QI Shuai, et al. Blue light regulating flowering time in higher plants:cryptochromes meated signal transduction[J]. Mol Plant Breed, 2015, 13(2):450-460. [15] 贾贞, 赵菲佚, 吴存祥. FT及其同源基因在植物发育调控中的多功能效应[J].西北植物学报, 2011, 31(12):2558-2564. JIA Zhen, ZHAO Feiyi, WU Cunxiang. Pleiotropic effect of FT and its orthologues on plant development[J]. Acta Bot Boreal-Occident Sin, 2011, 31(12):2558-2564. [16] CIFUENTES-ESQUIVEL N, BOU-TORRENT J, GALSTYAN A, et al. The bHLH proteins BEE and BIM positively modulate the shade avoidance syndrome in Arabidopsis seedlings[J]. Plant J Cell Mol Biol, 2013, 75(6):989-1002. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2017.01.019







 下载:
下载: