-
沙棘Hippophae rhamnoides别名醋柳、沙枣,为胡颓子科Elaeagnaceae沙棘属Hippophae多年生落叶灌木或小乔木。根据SUN等[1]和BARTISH等[2]的系统分类,沙棘属共有7个种,11个亚种;其中有9个亚种广泛分布于亚欧温带地区。沙棘果实中维生素C和黄酮含量较高,种子中富含脂肪油,枝叶富含高蛋白,这些生物活性物质是美容产品、抗败血酸药物和营养补充剂等的重要组成成分[3];沙棘的根系能产生根瘤,具有固氮功能,从而可达到改善土壤的作用;又由于其能适应干旱的环境,可以防风固沙,因此可用于生态恢复和土壤保护。因此沙棘是集生态、经济和社会效益为一体的多用途树种[3-4]。中国是沙棘属植物类群分布最多的国家,包括7个种,5个亚种;其沙棘属的资源蕴藏量也最大,沙棘天然林分布达67.54万hm2[4];而中国的青藏高原地区被认为是沙棘的起源中心[5]。目前,中国沙棘种植总面积达200.00万hm2,约占世界总面积的90%以上[6]。但当前广泛用于栽培的大果沙棘品种主要是从俄罗斯引进的,人工选育自蒙古沙棘H. rhamnoides subsp. mongolica亚种的天然远缘杂交种[7]。中国沙棘自然资源的开发和利用对其人工培育有重要意义;而对沙棘遗传多样性的系统研究是沙棘新品种选育的基础之一。目前,已有利用分子标记研究沙棘属植物的系统发育关系的报道[1, 8],探讨了沙棘种下不同亚种的起源过程[5],分析了沙棘个别亚种,如中国沙棘H. rhamnoides subsp. sinensis,云南沙棘H. rhamnoides subsp. yunnanensis的遗传多样性,但是对于沙棘种下多个亚种的多样性及分化的研究很少。本研究利用简单序列重复(SSR)标记比较分析了沙棘中分布最广的7个亚种的遗传多样性和遗传分化,分析了当前广泛栽培的26个大果沙棘品种的遗传多样性和可能来源,以期为沙棘新品种选育提供基础。
-
从野外采集中国沙棘,云南沙棘,中亚沙棘H. rhamnoides subsp. turkestanica,蒙古沙棘,高加索沙棘H. rhamnoides subsp. caucasica,海滨沙棘H. rhamnoides subsp. rhamnoides和溪生沙棘H. rhamnoides subsp. fluviatilis共7个沙棘亚种叶片样品,其中:蒙古沙棘20个个体,其他沙棘亚种各10个个体;收集在中国林业科学研究院温室播种的高加索沙棘幼苗10株;从黑龙江农业科学院浆果研究所、中国林业科学研究院沙漠实验中心收集26个大果沙棘品种的叶片。样品采集见表 1。叶片采集后用变色硅胶干燥处理带回实验室,采用植物全基因组DNA提取试剂盒(北京天根生物公司)提取总DNA。
表 1 7个沙棘亚种与26个大果沙棘品种采样信息
Table 1. Sampling information of the 7 subspecies and 26 cultivars of Hippophae rhamnoides
中文名 简写 样本数 采样点 中国沙棘 ZG 10 中国青海西宁大通县、门源、祁连山等 云南沙棘 YN 10 中国四川红原、西藏林芝工布江达、米林等 中亚沙棘 ZY 10 中国新疆乌恰县城、吉根乡等 蒙古沙棘 MG 20 中国新疆哈巴河县、吉木乃县、布尔津县、阿勒泰喀拉希力克乡等 溪生沙棘 XS 10 瑞士苏黎世、法国弗莱堡、德国汉堡等 高加索沙棘 GJS 10 各地混合种子播种 海滨沙棘 HB 10 丹麦、罗马尼亚等 大果沙棘品种 CV 26 中国黑龙江农业科学科院浆果研究所、中国林业科学研究院沙漠林业实验中心 -
所用的14个微卫星标记选自李珊珊等[9]从沙棘转录组和基因组中开发的,引物信息见表 2。为简化实验,PCR扩增采用将2~4对携带不同荧光标记(Fam, Hex, Rox和Tamra,生工生物工程股份有限公司,上海),且扩增产物长度不同的微卫星引物混合后,在同一PCR管中进行。扩增反应体系为30 μL,分别包括2×T5 Super PCR Mix(PAGE)(擎科新业生物技术有限公司,北京),正向和反向引物分别0.2 μmol·L-1和模板DNA约50 ng。热循环反应在ABI System 9700PCR扩增仪(Applied Biosystems, Life Technologies)上进行,程序如下:94 ℃预变性4 min,10个循环的94 ℃变性30 s,60 ℃(每个循环降1 ℃)退火30 s和72 ℃延伸45 s,20个循环的94 ℃变性30 s,50 ℃退火30 s和72 ℃延伸45 s,最后72 ℃ 10 min。
表 2 14个微卫星位点基本信息
Table 2. Information of the 14 microsatellite loci
引物名称* 引物序列(5′→3′) 重复基序 退火温度/℃ 等位基因数 等位基因大小/bp 引物来源 eHs008 F: ACTCATGCCCATCACCTTTT (AT)6 55 8 213~239 李珊珊等[9] R: GCTCGTTGCCACTGTTACAA eHs032 F: GCAGTCCGGACAGTCAGAAA (GTT)7 55 5 248~260 李珊珊等[9] R: CCAAAGCAAAACCACGCAGA eHs047 F: GTGCAAAAACCAGGAGTGCC (TAG)6 55 6 154~169 李珊珊等[9] R: TACAACCCTGCTACCCCCAT eHs146 F: ACAAGCGTGTGAGGATTCTT (CTA)5 55 9 109~133 李珊珊等[9] R: CGATGAGAGGCCGGCATATT eHs150 F: TTGCCACCTCGATTGGTACC (TGT)6 55 8 215~239 李珊珊等[9] R: GACTCACACTGCACATGAGGA eHs195 F: TCCTCCTGACTGTCTCGACC (TA)7 55 17 219~265 李珊珊等[9] R: CCCTAGTGAAGCTCAGTTTTGC eHs219 F: CTCCTGACAAAACTGTTGCA (CAC)6 55 5 208~220 李珊珊等[9] R: TATGGGGTCCCAACTCCCTT eHs296 F: TGAACGCTCATGATCAAAACGT (TGA)5 55 6 208~223 李珊珊等[9] R: CAGCTGGTGGAAGTTGAGGT gHs155 F: GTCTCTTCCCAAGCTCACATGC (TC)6 55 6 132~142 本研究 R: TGGGCCAAAATGTACTGGATA gHs238 F: CCCACTCCATAATCACTCTC (TC)6 55 14 127~153 本研究 R: TGAGAAAATTGGAGTCAAGTG gHs242 F: CGTAATAATCGGCCCCTAGATAA (AT)8 55 18 132~180 本研究 R: ATGCACCATATGCCTTCAAATAT gHs304 F: TTCTGTGAGTGCTGGGTTTAGCTT (CT)6 55 11 125~165 本研究 R: GCTTATTGCGGAGAAGAAGAACAA gHs317 F: TTCCAGATTCCATTACCCAAATTC (AG)15 55 16 141~175 本研究 R: TCTCCTACCAGGCCAACATAAAAA gHs321 F: TTTGTGATCCTTGTGCTTCATTTG (AAT)5 55 4 150~159 本研究 R: GGTTTTACAATGTGTCCTCATCATC 说明:*eHs开始的引物为EST-SSR标记,gHs开始的引物为基因组SSR PCR产物交由擎科新业生物技术有限公司进行变性[0.5 μL产物与9.0 μL的甲酰胺,0.5 μL的内标Liz500(Applied Biosystems, Life Technologies)混合后93 ℃变性5 min],然后在ABI 3730测序仪上进行毛细管电泳。最后利用Genemapper 4.0软件(Applied Biosystems, Life Technologies)读取电泳结果并分析得到每个个体基因型。每一批数据从PCR到毛细管电泳,都以大果沙棘品种‘向阳’为阳性对照。
-
采用软件GenALEX 6.503[10],针对每个沙棘亚种和所有品种,计算如下遗传多样性指数:等位基因个数(NA),有效等位基因个数(Ne),Shannon信息指数(I),观测杂合度(HO),期望杂合度(HE)和固定指数(F)。利用微卫星分析软件MSA 4.05[11]计算亚种两两之间的遗传分化系数(FST)[12],并进行10 000次重抽样后统计显著性,检验各亚种间分化是否显著偏离于0。
-
基于个体多位点微卫星基因型,利用MSA软件[11]计算个体之间的遗传距离(DC)[13];然后基于此遗传距离,采用PHYLIP 3.695[14]软件包中的NEIGHBOR子程序构建系统发育树,自展值设为500次。
-
利用贝叶斯聚类软件STRUCTURE 2.3.4[15]确定沙棘各亚种的分组,并鉴定大果沙棘品种的来源。分析采用混合模型(admixture model),考虑各亚种的等位基因频率相关(allele frequencies correlated),分组数K值设置为1~10,每个K值重复计算10次,每次的运算进行1 000 000次迭代和100 000次BURNIN。利用Structure Harvester[16]计算STRUCTURE运算结果中各K值所对应的似然值lnP(D)与模型值ΔK,根据lnP(D)和ΔK最大的原则[17]选择合适的分组数。
-
沙棘7个亚种的14个微卫星位点平均NA为2.643~4.643,NE为1.869~3.065,I为0.559~1.053,HO为2.460~0.477,HE为0.293~0.523,F为0.002~0.300。其中:中国沙棘的遗传多样性为所有沙棘亚种中最高,有着最高的NE,I,HO和HE(表 3),其次为云南沙棘,但云南沙棘的F远高于中国沙棘,显示云南沙棘个体间差异较大。高加索沙棘的NE,I,HE和F在所有沙棘亚种中最低,表明其遗传多样性水平最低,次低为海滨沙棘。总体来看,亚洲分布的中国沙棘、云南沙棘、蒙古沙棘和中亚沙棘遗传多样性普遍高于欧洲分布的溪生沙棘、海滨沙棘和高加索沙棘。26个大果沙棘品种的等位基因个数最多,可能与其个体数最多有关,但是其NE,I,HO,HE与各沙棘亚种的值相比都属于中等。
表 3 7个沙棘亚种与26个沙棘品种的遗传多样性
Table 3. Genetic diversity of the 7 subspecies and 26 cultivars of Hippophae rhamnoides
亚种编号 等位基因个数NA 有效等位基因个数NE Shannon信息指数I 观测杂合度HO 期望杂合度HE 固定指数F ZG 4.286 3.065 1.053 0.477 0.523 0.043 YN 4.643 3.052 1.046 0.343 0.515 0.300 ZY 3.500 2.241 0.864 0.417 0.476 0.148 MG 4.143 2.094 0.841 0.324 0.437 0.198 XS 3.214 2.217 0.752 0.386 0.403 0.042 GJS 2.643 1.869 0.559 0.266 0.293 0.042 HB 3.000 1.920 0.620 0.246 0.316 0.191 CV 4.714 2.267 0.896 0.429 0.444 0.002 说明:编号所代表的亚种名见表 1 沙棘亚种两两之间的FST都显著大于0(表 4),表明各亚种间分化明显。其中:蒙古沙棘和中亚沙棘之间分化最低(FST=0.274),次低为中国沙棘和云南沙棘之间(FST=0.283);中国沙棘和高加索沙棘之间遗传分化最高(FST=0.535)。亚洲分布的沙棘亚种与欧洲分布的沙棘亚种之间分化普遍高于这2个地区内部亚种之间的分化。
表 4 基于14个SSR位点的沙棘亚种间遗传分化系数
Table 4. Subspecies pairwise FST based on 14 SSR loci
亚种(品种) ZG YN ZY MG XS GJS ZG YN 0.283*** ZY 0.412*** 0.369*** MG 0.434*** 0.368*** 0.274*** XS 0.456*** 0.424*** 0.386*** 0.447*** GJS 0.535*** 0.513*** 0.378*** 0.483*** 0.427*** HB 0.511*** 0.468*** 0.432*** 0.462*** 0.345*** 0.354*** 说明:编号所代表的亚种名见表 1。***表示P<0.001 -
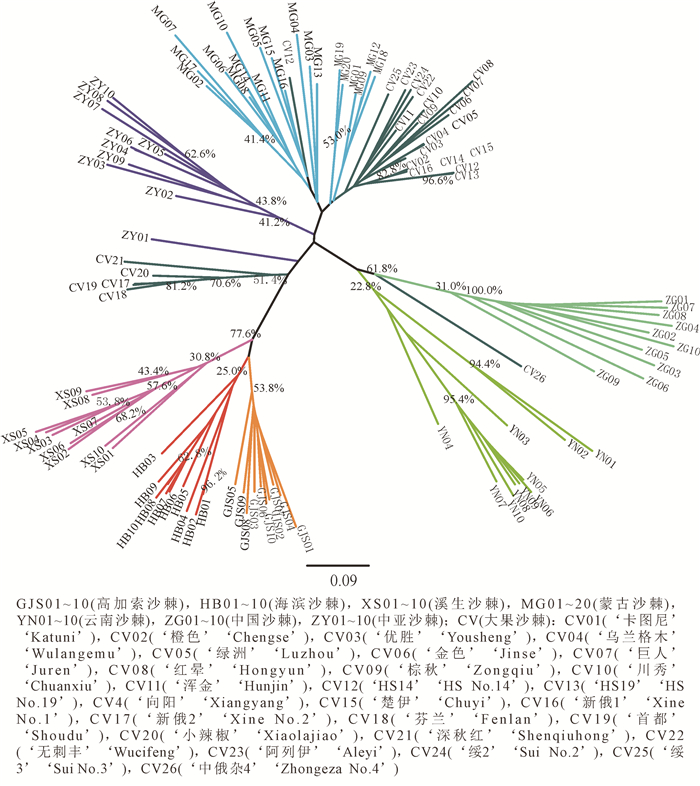
系统发育树结果显示:除大果沙棘品种外,所有沙棘个体分成7个分支,与亚种分类基本一致。仅1个中亚沙棘个体列到亚种分支以外,但各分支的支持率并不高。这7个分支可以分成2个大支:欧洲分布的高加索沙棘、溪生沙棘和海滨沙棘以77.6%的支持率聚为一大支;亚洲分布的中国沙棘、云南沙棘、中亚沙棘和蒙古沙棘聚为另一大支(图 1)。大果沙棘品种主要分成2个大支:大部分的品种个体聚成一支,然后与蒙古沙棘亚种野生个体分支聚在一起;另有5个品种(‘新俄2’‘Xine No.2’,‘芬兰’‘Fenlan’,‘首都’‘Shoudu’,‘小辣椒’‘Xiaolajiao’,‘深秋红’‘Shenqiuhong’)以超过50%的支持率聚为一支,位于欧洲分布的沙棘亚种分支和亚洲分布的沙棘亚种分支之间。大果沙棘品种‘中俄杂4’‘Zhongeza No.4’位于中国沙棘和云南沙棘分支的基部。
-
贝叶斯聚类分析发现:当K=7时,lnP(D)迅速增加;当K>7时lnP(D)的增加趋于缓慢。可见,K=7是一个明显的拐点(图 2A)。因此,lnP(K)支持所有的沙棘亚种和品种分为7组。而从ΔK与K的关系可以看出:在K=3处,ΔK有1个显著的峰值(图 2B),因而支持所有沙棘个体划分为3组。第1组由中国沙棘和云南沙棘组成,第2组主要由蒙古沙棘和大部分大果沙棘品种组成,第3组由溪生沙棘、高加索沙棘和海滨沙棘组成。中亚沙棘的遗传成分主要来源于第2组,并包含少量的第3组和更少的第1组遗传成分。大果沙棘品种‘新俄2’‘芬兰’‘首都’‘小辣椒’和‘深秋红’的遗传组成中第2组和第3组各约占50%;大果沙棘品种‘巨人’‘Juren’的遗传组成主要来自第2组,第3组约占20%;大果沙棘‘中俄杂4’的遗传组成中第1组和第2组各约占50%。当所有个体划分为7组时,中国沙棘、云南沙棘、中亚沙棘、蒙古沙棘、溪生沙棘、高加索沙棘和大部分大果沙棘品种各自组成一组,但云南沙棘包含少量其他各组的成分,中亚沙棘和蒙古沙棘包含大果沙棘组的成分,溪生沙棘包含高加索沙棘和云南沙棘的遗传成分;海滨沙棘由溪生沙棘和高加索沙棘的遗传成分共同构成,大果沙棘品种‘新俄2’‘芬兰’‘首都’‘小辣椒’和‘深秋红’的遗传成分一半来自大果沙棘组,另一半由溪生沙棘和高加索沙棘组共同构成,大果沙棘‘中俄杂4’则中国沙棘和大果沙棘组各约占50%。
-
廉永善等[18]根据形态特征分析认为:沙棘种下的9个亚种可归为3组:中国沙棘、云南沙棘和卧龙沙棘H. rhamnoides subsp. wolongensis分为第1组;蒙古沙棘、高加索沙棘和中亚沙棘为第2组;喀尔巴千山沙棘H. rhamnoides subsp. carpatica,海滨沙棘和溪生沙棘为第3组。马玉花等[8]基于ITS序列建立的系统树认为沙棘9个亚种分为2个组:高加索沙棘、海滨沙棘、喀尔巴千山沙棘、中亚沙棘、云南沙棘和卧龙沙棘为一组;蒙古沙棘、溪生沙棘与中国沙棘为另一组。本研究基于14个微卫星位点的基因型构建的系统发育树可以看出:7个沙棘亚种主要分成2个组:欧洲分布的海滨沙棘、溪生沙棘和高加索沙棘为一组;亚洲分布的中国沙棘、云南沙棘、蒙古沙棘和中亚沙棘为另一组。组间不同亚种之间的遗传分化系数也明显高于组内不同亚种间。这说明跨欧亚大陆的地理隔离已造成欧洲和亚洲分布的沙棘亚种之间更为明显的分化。贝叶斯分析显示:这7个沙棘亚种可分为3个组:中国沙棘和云南沙棘分为第1组;蒙古沙棘和中亚沙棘为第2组;海滨沙棘、溪生沙棘和高加索沙棘为第3组。该分组结果与廉永善等[18]根据形态特征的分组更为相近,只是在高加索沙棘的归属上稍有差别。高加索沙棘可能在欧洲分布的时间较短,导致高加索沙棘与溪生沙棘、海滨沙棘的基因交流更广泛,但在形态学上,高加索沙棘还未能从蒙古沙棘和中亚沙棘区分开。
本研究还发现:在系统发育树中,沙棘个体大都按亚种聚类,即来自同一亚种的个体聚为一支;在贝叶斯聚类分析中,当分7组时,除海滨沙棘由溪生沙棘组和高加索沙棘组共同构成外,其他亚种都各自分成一组。这说明沙棘各亚种间存在明显的遗传分化,与两两沙棘亚种间较高的遗传分化系数相符。基因交流的限制[19]和对各自生境的适应性进化都可能造成沙棘亚种间明显的遗传分化。然而,贝叶斯聚类分析显示:各沙棘亚种间可能存在少量的杂交和基因渐渗;系统发育树中每个亚种的分支支持率并不高,也说明种间杂交可能存在。这与JIA等[5]利用cpDNA片段以及ITS序列对沙棘种下9个亚种的分析结果一致。然而,要进一步确认各亚种间杂交和基因渐渗的方向和程度,还需要更多样品和基因组范围内更多位点的详细分析。
-
JIA等[5]推测沙棘主要起源于中国青藏高原地区,中国东部和欧洲分布的沙棘亚种都是从该地区扩散而来。本研究发现:中国分布的中国沙棘和云南沙棘的遗传多样性水平远高于欧洲分布的溪生沙棘、高加索沙棘和海滨沙棘,这与沙棘起源于中国青藏高原地区的假说相符。相比分布广泛的中国沙棘,云南沙棘仅分布在西藏、四川、青海和云南交界的狭长地带,分布范围较小,较高的遗传多样性可能与其分布邻近起源中心有关。云南沙棘分布区地形复杂,环境变化多端,因此,该亚种各个体也可能因适应不同的环境而积累了较高的遗传变异[19]。云南沙棘有着最高的固定指数F,也说明其个体间遗传变异较大。
分析样品的数量和采集方式也可能会影响沙棘亚种遗传多样性的评估。在本研究中,除蒙古沙棘为20个个体外,其他沙棘均为10个个体。因此当亚种间进行比较时,不存在样品数量的偏差。另外,除高加索沙棘取自种子的播种苗外,其他沙棘亚种的个体都采自2~4个野外种群,这也将减小地理种群的偏差。当然,要准确评估沙棘各亚种内的遗传多样性水平,还需整个分布区更全面的采样。
-
基于14个位点的微卫星基因型构建的系统发育树中,大部分大果沙棘品种都和采自中国新疆阿勒泰地区的蒙古沙棘亚种聚在一起;贝叶斯聚类分析将所有沙棘个体划分为3组时(K=3),大果沙棘品种也和蒙古沙棘亚种为一组,这与大果沙棘人工选育自蒙古沙棘亚种的记录相符。然而,系统发育树和贝叶斯聚类分析都能将大果沙棘品种和中国新疆阿勒泰地区的蒙古沙棘个体分开,说明用来品种选育的俄罗斯蒙古沙棘种群与中国新疆阿勒泰地区的蒙古沙棘种群存在明显的遗传分化。系统发育树和贝叶斯聚类分析表明:本研究所分析的大果沙棘品种并非都选育自蒙古沙棘亚种,品种‘新俄2’‘芬兰’‘首都’‘小辣椒’和‘深秋红’可能是蒙古沙棘和欧洲分布的沙棘亚种杂交的结果。
本研究还发现:所收集的26个大果沙棘品种多样性稍高于中国新疆阿勒泰地区的蒙古沙棘亚种野生种群,与本研究的7个沙棘亚种相比属于中等偏上水平,表明用于栽培的大果沙棘品种具有丰富的遗传变异。刘雨娜等[20]曾利用RAPD标记分析了24个大果沙棘品种得出同样的结论。大果沙棘品种来源于蒙古沙棘的远缘杂交后代,这自然会提高大果沙棘的多样性水平;其次,本研究表明:少量大果沙棘品种具有欧洲分布的其他沙棘亚种的遗传成分,说明这些沙棘可能是蒙古沙棘和欧洲其他沙棘种杂交的结果,这也提高了大果沙棘的多样性水平。虽然大果沙棘品种有着丰富的遗传变异,但其多样性仍低于从野外采集的少量中国沙棘和云南沙棘个体。这说明,中国丰富的沙棘自然资源可为沙棘新品种的选育提供重要的遗传基础。
Genetic variation of 7 subspecies and 26 important cultivars in Hippophae rhamnoides
-
摘要: 利用14个微卫星标记分析了沙棘Hippophae rhamnoides 7个亚种和26个大果沙棘品种的遗传多样性,采用系统发育树和贝叶斯聚类方法分析了这些个体的聚类情况。结果显示:中国沙棘H. rhamnoides subsp. sinensis和云南沙棘H. rhamnoides subsp. yunnanensis遗传多样性水平最高,高加索沙棘H. rhamnoides subsp. caucasica最低。系统发育树将所有个体聚为2个大支:亚洲分支和欧洲分支。大部分大果沙棘品种与亚洲的蒙古沙棘H. rhamnoides subsp. mongolica聚为1支,少量个体位于亚洲分支和欧洲分支之间。贝叶斯聚类分析将所有个体划分为3组或7组:分3组时,中国沙棘和云南沙棘为一组,蒙古沙棘和大部分大果沙棘品种为一组,溪生沙棘H. rhamnoides subsp. fluviatilis,高加索沙棘和海滨沙棘H. rhamnoides subsp. rhamnoides为一组;分7组时,除海滨沙棘由溪生沙棘组和高加索沙棘组共同构成外,其他亚种和大果沙棘品种各自为一组,并发现少量组间混合基因型。表明沙棘种下亚种间分化明显,亚洲和欧洲分布的沙棘亚种间的分化尤为明显;大果沙棘品种大部分源于蒙古沙棘亚种,个别品种可能为蒙古沙棘和欧洲沙棘亚种间的杂交后代。Abstract: Hippophae rhamnoides have important ecological, economic and social benefits, so it is essential to understand the genetic variation of the species. Genetic diversity on 14 microsatellite loci was estimated for seven H. rhamnoides subspecies and 26 big grain sea buckthorn cultivars. The clustering of these individuals was analyzed by phylogenetic tree and Bayes cluster. Results indicated that H. rhamnoides subsp. sinensis had the highest genetic diversity, followed by H. rhamnoides subsp. yunnanensis; H. rhamnoides subsp. caucasica had the lowest genetic diversity. The phylogenetic tree based on individual genotypic distance classified all individuals into two big clades according to distribution, the Asian clade and the European clade. Most big grain sea buckthorn cultivars clustered with H. rhamnoides subsp. mongolica individuals collected from the Asian clade with a few individuals located between the two clades. The Bayesian cluster analysis found that all individuals could be classified into three or seven groups. For three groups, group one consisted of H. rhamnoides subsp. yunnanensis and H. rhamnoides subsp. sinensis; group two had the most cultivars and H. rhamnoides subsp. mongolica; and group three was H. rhamnoides subsp. fluviatilis, H. rhamnoides subsp. caucasica, and H. rhamnoides subsp. rhamnoides. For seven groups, all subspecies were classified into a different group with a few hybrids, and only H. rhamnoides subsp. rhamnoides showed a hybrid ancestry between H. rhamnoides subsp. fluviatilis and H. rhamnoides subsp. caucasica. There were also a few groups with mixed genotypes. Thus, genetic divergence among these seven H. rhamnoides subspecies was important, especially between subspecies distributed in different continents; whereas, big grain sea buckthorn cultivars were mostly selected from H. rhamnoides subsp. mongolica with a few of hybrid origin from H. rhamnoides subsp. mongolica and subspecies in Europe.
-
表 1 7个沙棘亚种与26个大果沙棘品种采样信息
Table 1. Sampling information of the 7 subspecies and 26 cultivars of Hippophae rhamnoides
中文名 简写 样本数 采样点 中国沙棘 ZG 10 中国青海西宁大通县、门源、祁连山等 云南沙棘 YN 10 中国四川红原、西藏林芝工布江达、米林等 中亚沙棘 ZY 10 中国新疆乌恰县城、吉根乡等 蒙古沙棘 MG 20 中国新疆哈巴河县、吉木乃县、布尔津县、阿勒泰喀拉希力克乡等 溪生沙棘 XS 10 瑞士苏黎世、法国弗莱堡、德国汉堡等 高加索沙棘 GJS 10 各地混合种子播种 海滨沙棘 HB 10 丹麦、罗马尼亚等 大果沙棘品种 CV 26 中国黑龙江农业科学科院浆果研究所、中国林业科学研究院沙漠林业实验中心 表 2 14个微卫星位点基本信息
Table 2. Information of the 14 microsatellite loci
引物名称* 引物序列(5′→3′) 重复基序 退火温度/℃ 等位基因数 等位基因大小/bp 引物来源 eHs008 F: ACTCATGCCCATCACCTTTT (AT)6 55 8 213~239 李珊珊等[9] R: GCTCGTTGCCACTGTTACAA eHs032 F: GCAGTCCGGACAGTCAGAAA (GTT)7 55 5 248~260 李珊珊等[9] R: CCAAAGCAAAACCACGCAGA eHs047 F: GTGCAAAAACCAGGAGTGCC (TAG)6 55 6 154~169 李珊珊等[9] R: TACAACCCTGCTACCCCCAT eHs146 F: ACAAGCGTGTGAGGATTCTT (CTA)5 55 9 109~133 李珊珊等[9] R: CGATGAGAGGCCGGCATATT eHs150 F: TTGCCACCTCGATTGGTACC (TGT)6 55 8 215~239 李珊珊等[9] R: GACTCACACTGCACATGAGGA eHs195 F: TCCTCCTGACTGTCTCGACC (TA)7 55 17 219~265 李珊珊等[9] R: CCCTAGTGAAGCTCAGTTTTGC eHs219 F: CTCCTGACAAAACTGTTGCA (CAC)6 55 5 208~220 李珊珊等[9] R: TATGGGGTCCCAACTCCCTT eHs296 F: TGAACGCTCATGATCAAAACGT (TGA)5 55 6 208~223 李珊珊等[9] R: CAGCTGGTGGAAGTTGAGGT gHs155 F: GTCTCTTCCCAAGCTCACATGC (TC)6 55 6 132~142 本研究 R: TGGGCCAAAATGTACTGGATA gHs238 F: CCCACTCCATAATCACTCTC (TC)6 55 14 127~153 本研究 R: TGAGAAAATTGGAGTCAAGTG gHs242 F: CGTAATAATCGGCCCCTAGATAA (AT)8 55 18 132~180 本研究 R: ATGCACCATATGCCTTCAAATAT gHs304 F: TTCTGTGAGTGCTGGGTTTAGCTT (CT)6 55 11 125~165 本研究 R: GCTTATTGCGGAGAAGAAGAACAA gHs317 F: TTCCAGATTCCATTACCCAAATTC (AG)15 55 16 141~175 本研究 R: TCTCCTACCAGGCCAACATAAAAA gHs321 F: TTTGTGATCCTTGTGCTTCATTTG (AAT)5 55 4 150~159 本研究 R: GGTTTTACAATGTGTCCTCATCATC 说明:*eHs开始的引物为EST-SSR标记,gHs开始的引物为基因组SSR 表 3 7个沙棘亚种与26个沙棘品种的遗传多样性
Table 3. Genetic diversity of the 7 subspecies and 26 cultivars of Hippophae rhamnoides
亚种编号 等位基因个数NA 有效等位基因个数NE Shannon信息指数I 观测杂合度HO 期望杂合度HE 固定指数F ZG 4.286 3.065 1.053 0.477 0.523 0.043 YN 4.643 3.052 1.046 0.343 0.515 0.300 ZY 3.500 2.241 0.864 0.417 0.476 0.148 MG 4.143 2.094 0.841 0.324 0.437 0.198 XS 3.214 2.217 0.752 0.386 0.403 0.042 GJS 2.643 1.869 0.559 0.266 0.293 0.042 HB 3.000 1.920 0.620 0.246 0.316 0.191 CV 4.714 2.267 0.896 0.429 0.444 0.002 说明:编号所代表的亚种名见表 1 表 4 基于14个SSR位点的沙棘亚种间遗传分化系数
Table 4. Subspecies pairwise FST based on 14 SSR loci
亚种(品种) ZG YN ZY MG XS GJS ZG YN 0.283*** ZY 0.412*** 0.369*** MG 0.434*** 0.368*** 0.274*** XS 0.456*** 0.424*** 0.386*** 0.447*** GJS 0.535*** 0.513*** 0.378*** 0.483*** 0.427*** HB 0.511*** 0.468*** 0.432*** 0.462*** 0.345*** 0.354*** 说明:编号所代表的亚种名见表 1。***表示P<0.001 -
[1] SUN K, CHEN X L, MA R J, et al. Molecular phylogenetics of Hippophae L.(Elaeagnaceae) based on the internal transcribed spacer (ITS) sequences of nrDNA[J]. Plant Syst Evol, 2002, 235(1/4):121-134. [2] BARTISH I V, JEPPSSON N, NYBOM H, et al. Phylogeny of Hippophae (Elaeagnaceae) inferred from parsimony analysis of chloroplast DNA and morphology[J]. Syst Bot, 2002, 27(1):41-54. [3] HAKEEM K R, ÖZTÜRK M, ALTAY V, et al. An alternative potential natural genetic resource: sea buckthorn[Elaeagnus rhamnoides (syn.: Hippophae rhamnoides)] [C]//ÖZTÜRK M, HAKEE M KR, ASHRAF M, et al. Global Perspectives on Underutilized Crops. Cham: Springer International Publishing, 2018: 25-82. [4] 齐虹凌, 于泽源, 李兴国.沙棘研究概述[J].沙棘, 2005, 18(2):37-41. QI Hongling, YU Zeyuan, LI Xingguo. Summary of studies on seabuckthorn[J]. Hippophae, 2005, 18(2):37-41. [5] JIA Dongrui, ABBOTT R J, LIU Tengliang, et al. Out of the Qinghai-Tibet Plateau:evidence for the origin and dispersal of Eurasian temperate plants from a phylogeographic study of Hippophae rhamnoides (Elaeagnaceae)[J]. New Phytol, 2012, 194(4):1123-1133. [6] 林赫杰, 陈钰.沙棘研究现状、开发利用及发展前景[J].天津农业科学, 2010, 16(2):128-130. LIN Haojie, CHEN Yu. Research status, development and prospects of seabuckthorn[J]. Tianjin Agric Sci, 2010, 16(2):128-130. [7] 黄铨, 于倬德.沙棘研究[M].北京:科学出版社, 2006. [8] 马玉花, 冶贵生, 向前胜, 等.基于ITS序列探讨沙棘属植物的系统发育关系[J].应用生态学报, 2014, 25(10):2985-2990. MA Yuhua, YE Guisheng, XIANG Qiansheng, et al. Phylogenetic relationships of seabuckthorn based on ITS sequences[J]. Chin J Appl Ecol, 2014, 25(10):2985-2990. [9] 李珊珊, 曾艳飞, 何彩云, 等.基于沙棘转录组序列开发EST-SSR分子标记[J].林业科学研究, 2017, 30(1):69-74. LI Shanshan, ZENG Yanfei, HE Caiyun, et al. Development of EST-SSR markers based on seabuckthorn transcriptomic sequences[J]. For Res, 2017, 30(1):69-74. [10] PEAKALL R, SMOUSE P E. GenAlEx 6.5:genetic analysis in Excel. Population genetic software for teaching and research:an update[J]. Bioinformatics, 2012, 28(19):2537-2539. [11] DIERINGER D, SCHLOTTERER C. MICROSATELLITE ANALYSER (MSA):a platform independent analysis tool for large microsatellite data sets[J]. Mol Ecol Notes, 2003, 3(1):167-169. [12] WEIR B S, COCKERHAM C C. Estimating F-Statistics for the analysis of population structure[J]. Evolution, 1984, 38(6):1358-1370. [13] CAVALLI S L L, EAWARDS A W F. Phylogenetic analysis:models and estimation procedures[J]. Am J Human Genet, 1967, 19(1/3):233-257. [14] FELSENSTEIN J. PHYLIP: phylogeny inference package, Version 3.695[CP/OL]. Seattle: Department of Genome Sciences, University of Washington, 2013. [15] FALUSH D, STEPHENS, M, PRITCHARD J K. Inference of population structure using multilocus genotype data:dominant markers and null alleles[J]. Mol Ecol Notes, 2007, 7(4):574-578. [16] EARL D A. STRUCTURE HARVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method[J]. Conserv Genet Resour, 2012, 4(2):359-361. [17] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters in individuals using the software STRUCTURE:a simlation study[J]. Mol Ecol, 2005, 14(8):2611-2620. [18] 廉永善, 陈学林.沙棘属植物的系统分类[J].沙棘, 1996, 9(1):15-24. LIAN Yongshan, CHEN Xuelin. Systematic classification of seabuckthorn plants[J]. Hippophae, 1996, 9(1):15-24. [19] 陈纹.沙棘属几个中国特有类群的遗传多样性研究[D].兰州: 西北师范大学, 2004. CHEN Wen. The Study of Genetic Diversity of Three Endemic Taxa of Hippophae in China[D]. Lanzhou: Northwest Normal University, 2004. [20] 刘雨娜, 于泽源, 李兴国. 24个大果沙棘品种的RAPD分析[J].果树学报, 2007, 24(2):230-233. LIU Yuna, YU Zeyuan, LI Xingguo. RAPD analysis of 24 big grain seabuckthorns (Hippophae rhamnoides)[J]. J Fruit Sci, 2007, 24(2):230-233. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2019.04.006







 下载:
下载: