-
类黄酮物质参与植物多种生理活动,不仅能为植物生长发育提供帮助,同时还在植物抵抗逆境胁迫方面发挥作用[1]。黄烷酮3-羟化酶(F3H)作为类黄酮代谢途径上游的关键酶之一,负责催化形成二氢黄酮醇,之后经过花青素和黄酮醇合成途径形成各种类黄酮衍生物[2]。F3H在基因及蛋白结构上高度保守,其底物特异性较强,需要在2-氧化戊二酸盐、分子氧、亚铁离子(Fe2+)和抗坏血酸盐的辅助下发挥作用,所以在分类上也被归为2-酮戊二酸依赖性双加氧酶(2-ODD)家族[3−4]。目前,在草莓Fragaria vesca、红花Carthamus tinctorius、拟南芥Arabidopsis thaliana、茶Camellia sinensis、黄花红砂Reaumuria trigyna、独行菜Lepidium apetalum和铁皮石斛Dendrobium officinale等植物中相继报道了关于类黄酮合成关键酶基因F3H的调控研究[5−11]。在功能上,F3H基因对植物的颜色着色有重要作用。苹果 Malus domestica MdLUX和MdPCL-like通过其启动子区域DNA低甲基化以及激活MdF3H,促进果皮中的花青素合成[12]。在草莓中,RNAi介导的F3H基因沉默导致花青素及黄酮醇显著降低,产生无色的草莓果实[5]。此外,F3H基因表达受到多种植物激素的正调控,其转录水平的增加也能提高植物对非生物和生物胁迫的耐受能力[13]。番茄 Solanum lycopersicum SlF3HL能在低温胁迫中刺激转基因烟草 Nicotiana tabacum合成类黄酮物质,使烟草耐冷性提高[14]。CtF3H在不同表型和化学型的红花中作用不同,在茉莉酸甲酯(MeJA)刺激下,CtF3H在花橙黄色型红花中高表达,并与醌式查尔酮和黄酮醇的积累有关,但在花白色型红花中低表达且不影响黄酮醇的积累[6]。
掌叶覆盆子Rubus chingii是中国特色的药食同源植物,也是浙江省新“浙八味”之一,具有补肝益肾、固精缩尿等功效[15]。相关研究表明掌叶覆盆子富含20多种黄酮类活性成分,具有抗菌抗炎等多种药理活性[16−17]。然而掌叶覆盆子类黄酮生物合成及转录调控机制尚不清晰。本研究克隆得到1个掌叶覆盆子类黄酮生物合成途径关键酶基因RcF3H的全长序列,并对其进行生物信息学分析,同时利用转录组和实时荧光定量PCR (RT-qPCR)技术分析RcF3H基因在不同组织、果实发育期以及MeJA刺激下的表达特征,为后期解析RcF3H对掌叶覆盆子类黄酮代谢的调控机制提供参考。
-
掌叶覆盆子植物材料来自杭州市富阳环山掌叶覆盆子种植基地(29.93°N,119.95°E)。4个发育阶段(青果、青转黄果、黄果、红果)的掌叶覆盆子果实于2022年4—5月陆续采收。
激素MeJA处理:于2022年4月,选择生长状态良好的1年生掌叶覆盆子植株,使用100 μmol·L−1的MeJA溶液喷施叶片的正、反两面做胁迫处理(MJ),以清水为对照组(ck)。每2 d喷施1次,处理7 d后,采集每组植株从上往下的第2张叶片作为样本。各3个重复,液氮速冻处理后放于−80 ℃低温冰箱保存。
-
使用FastPure® Universal Plant Total RNA Isolation Kit提取试剂盒(RC411),提取掌叶覆盆子植物样本总RNA。使用分光光度计(NanoDrop 2000)检测提取掌叶覆盆子总RNA浓度。使用Evo M-MLV反转录试剂盒Ⅱ(AG11711)反转录合成掌叶覆盆子cDNA。总RNA和cDNA分别放入−80和−20 ℃低温冰箱保存备用。
-
根据掌叶覆盆子基因组文件[18]得到RcF3H基因的参考序列。使用Primer Premier 6.0设计RcF3H基因的扩增引物(表1)。引物合成由浙江尚亚生物技术有限公司完成。PCR扩增体系:ddH2O 9.5 μL,上下游引物各1 μL (10 μmol·L−1),掌叶覆盆子cDNA模板1 μL,12.5 μL PrimeSTAR Max premix(R045A)。PCR扩增程序:94 ℃ 3 min;98 ℃ 10 s,58 ℃ 15 s,72 ℃ 70 s,35个循环;72 ℃ 5 min;4 ℃ 保温。反应结束后将PCR扩增产物使用琼脂糖凝胶电泳检测并使用产物纯化试剂盒(DC301)回收。最后将RcF3H基因片段连接到pMD19-T载体上(D102A),热激法转化大肠埃希菌Escherichia coli DH5α感受态,挑取阳性单克隆进行菌液PCR鉴定,由浙江尚亚生物技术有限公司测序分析。
表 1 引物序列
Table 1. Primer sequence for this study
引物名称 引物序列(5′→3′) 引物用途 β-actin-F ATCCACGAGACTACATACAACTCC 内参基因 β-actin-R CTGTCTGCAATACCAGGGAAC RcF3H-F ATGGCTCCTACACCTACTAC 序列扩增 RcF3H-R AGCAAAAATACCATCCACTT RcF3H-qPCR-F CAAAGTGGCCTACAACCAATTC 荧光定量 RcF3H-qPCR-R CCTCGACAATCTTCTTGCAAATC -
使用Gene Structure Display Server网站分析RcF3H基因结构,并使用IBS 2.0作图。使用Expasy ProtParam tool网站预测掌叶覆盆子RcF3H蛋白的分子量、等电点等基本理化性质;通过Plant-mPLoc网站预测RcF3H蛋白的亚细胞定位情况。使用Expasy Protscale网站分析掌叶覆盆子RcF3H蛋白亲疏水性;使用美国国家生物技术信息中心(NCBI)中的CD-Search分析RcF3H蛋白的保守结构域。
使用SOPMA网站预测RcF3H蛋白的二级结构,利用Swiss-Model网站预测RcF3H蛋白的三维结构。从NCBI数据库的Protein Blast中下载与RcF3H有较高同源性的蔷薇科Rosaceae植物的F3H蛋白序列,使用DNAMAN软件进行多序列比对;使用MEGA 7软件将拟南芥、水稻Oryza sativa等单、双子叶其他科属植物的F3H蛋白序列采用邻接法(neighbor-joining)构建系统发育树(bootstrap值设置为1 000,其余参数默认),并利用ChiPlot作图。使用PlantCARE网站获得RcF3H基因前2 000 bp的启动子序列,检索得到顺式作用元件[19−20]。
-
参考掌叶覆盆子的组织转录组数据[18],分析不同组织下RcF3H基因相对表达量,并使用TBtools 软件绘制热图。
-
以反转录合成的掌叶覆盆子cDNA为材料,使用Primer Premier 6.0设计RcF3H基因的RT-qPCR引物(表1)。引物由浙江尚亚生物技术有限公司合成。RT-qPCR反应方法参照文献[21],使用iTaq™ universal SYBR® Green supermix (1725121)以及荧光定量PCR仪(ABI 7500)完成。RT-qPCR反应体系:iTaq™ universal SYBR® Green supermix 5 μL,上、下游引物各0.5 μL (10 mmol·L−1),cDNA模板1 μL (50 mg·L−1),ddH2O 3 μL。RT-qPCR反应程序:95 ℃ 30 s;95 ℃ 10 s,60 ℃ 30 s ,40个循环。结果以掌叶覆盆子β-actin为内参基因,采用2–∆∆Ct法[22]计算RcF3H基因相对表达量。
-
数据使用Excel 2021进行统计分析。使用SPSS 26进行单向方差分析以及t检验,使用GraphPad Prism 8作图。
-
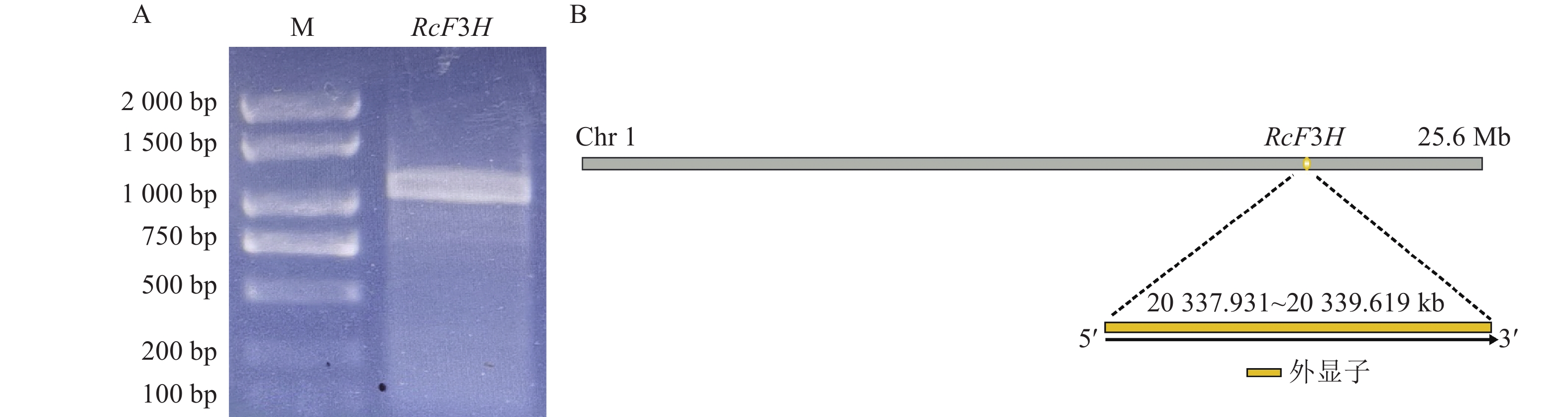
以反转录合成的掌叶覆盆子cDNA为模板,成功扩增得到目的基因片段(图1A)。对基因片段进行切胶回收,连接转化至DH5α感受态,12 h后对阳性单克隆菌液进行PCR验证并测序分析。结果显示:目的片段长度为1 098 bp,将其命名为RcF3H。RcF3H基因定位于第1条染色体内,由1个外显子构成,共编码了365个氨基酸(图2)。
-
RcF3H蛋白分子式为C1831H2900N498O553S13,相对分子量为65.16 kDa;理论等电点为5.64;脂肪系数为83.32;不稳定系数为38.22,属于稳定蛋白。亚细胞定位预测表明:该蛋白定位在细胞质上。
蛋白亲疏水性分析表明:RcF3H蛋白属于亲水性蛋白;蛋白平均亲水指数为−0.448,大部分区域表现出亲水性,最大疏水分值(−2.756)位于肽链第145位氨基酸,最大亲水分值(2.022)位于肽链第44位氨基酸(图3)。
蛋白结构域分析发现:RcF3H蛋白属于2-酮戊二酸依赖性双加氧酶家族,拥有2OG-FeⅡ_0xy区域以及PLN02515和Pcb C等2个保守结构域(图4)。
-
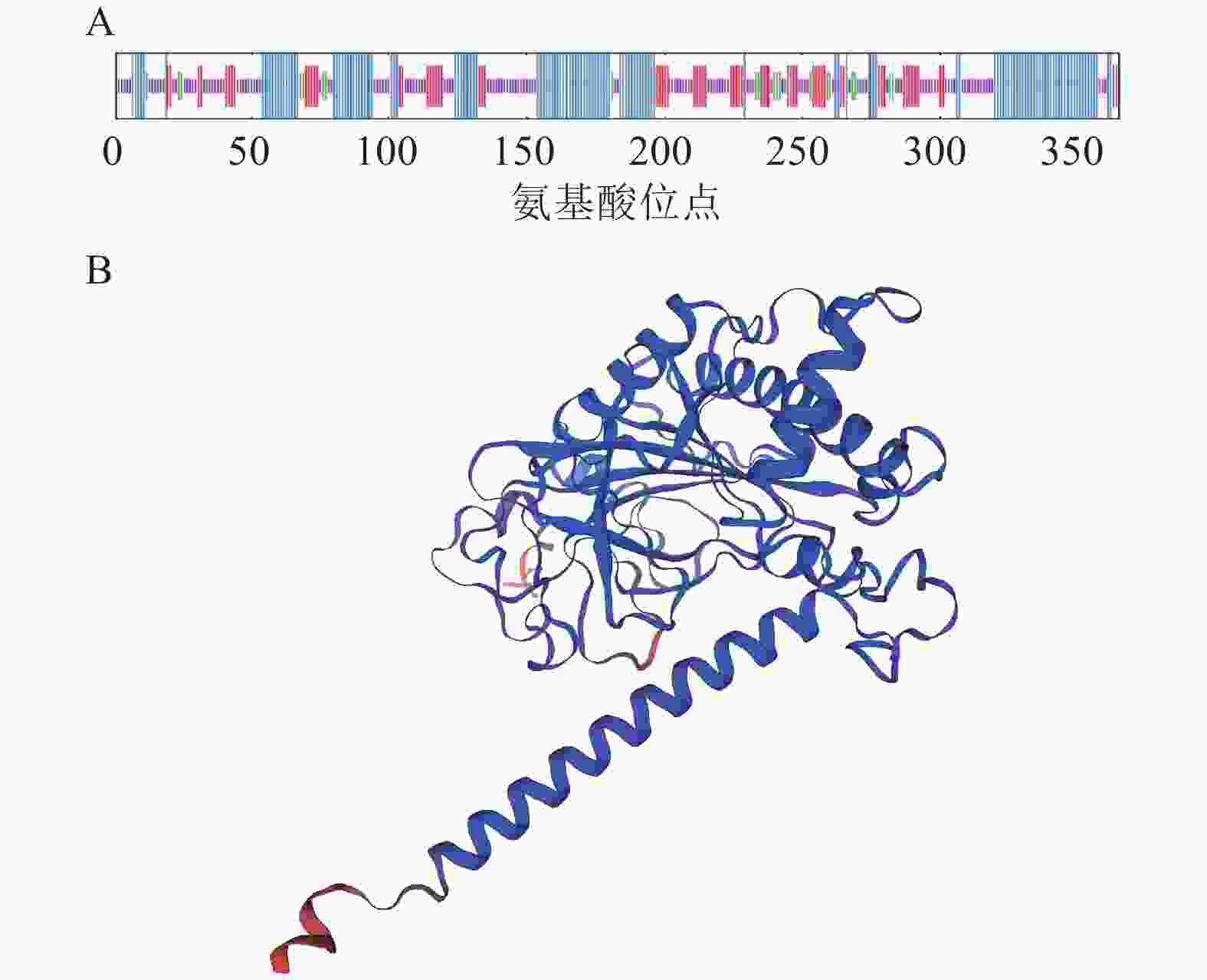
RcF3H蛋白的二级结构预测发现:该蛋白存在α-螺旋、β-折叠、直链延伸和无规则卷曲4种结构,其中以α-螺旋和无规则卷曲占比最大,分别占37.53%和38.63%,而直链延伸和β-折叠分别占18.36%和5.48% (图5)。此外,以石榴Punica granatum蛋白(编号:A0A218XVH5.1)为模板,对RcF3H蛋白的三级结构进行建模。建模的三级结构覆盖率为92.31%,序列一致性为85.75%。该结果与二级结构预测结果一致。
-
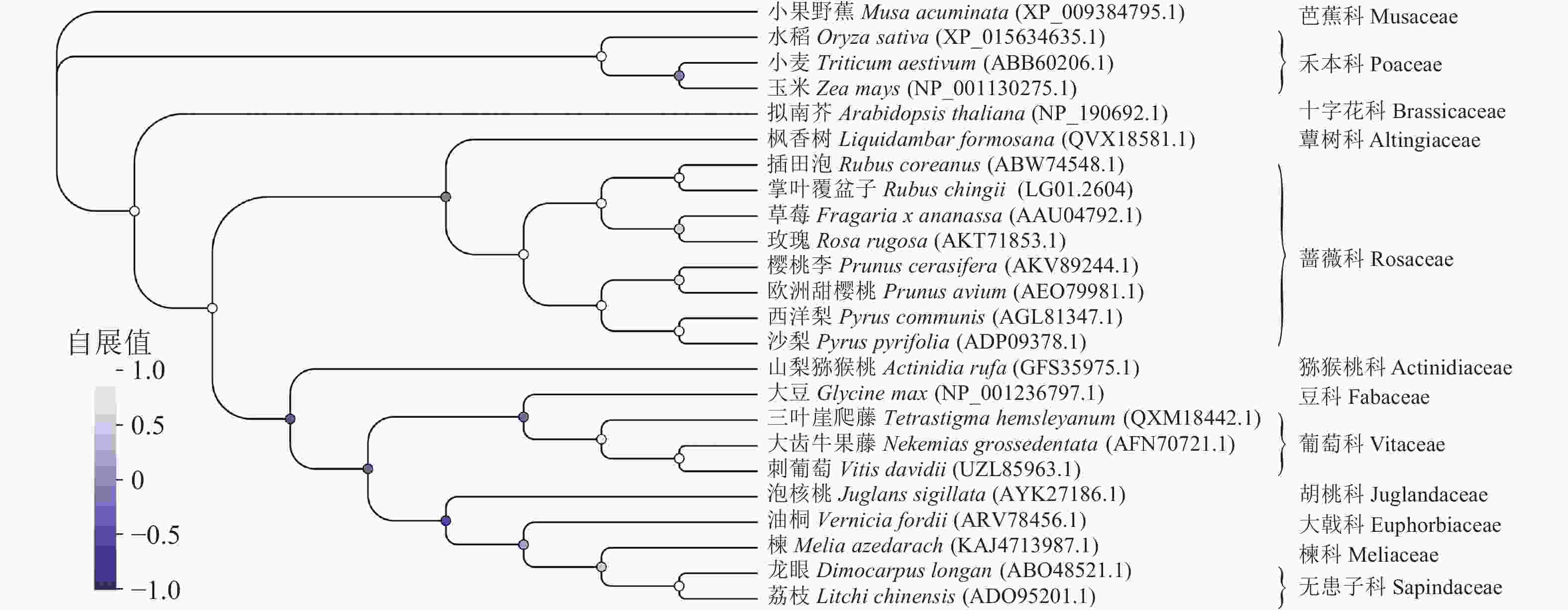
多序列比对分析发现:蔷薇科F3H蛋白序列具有很高的相似度,其中RcF3H与插田泡Rubus coreanus、草莓和玫瑰Rosa rugosa的相似性超过95%,与樱桃李Prunus cerasifera、欧洲甜樱桃Prunus avium和西洋梨Pyrus communis相似性接近90% (图6)。系统进化树分析发现:RcF3H与蔷薇科插田泡蛋白(编号:ABW74548.1)的亲缘关系最密切。同科属的F3H蛋白在系统进化上处于同一分支。与单子叶植物相比,掌叶覆盆子RcF3H与双子叶植物关系更密切,属于同一亚组(图7)。以上结果说明掌叶覆盆子RcF3H蛋白在生物进化过程中较为保守。
-
RcF3H基因启动子中具有大量相对保守的CAAT-box和TATA-box。根据顺式作用元件功能的不同,可以将其分为植物生长发育、激素响应以及胁迫响应3个类别(表2)。RcF3H基因启动子顺式作用元件有2个植物生长发育的顺式调控元件;在激素响应中茉莉酸响应元件数量最多,赤霉素和生长素次之。此外,RcF3H基因在胁迫响应上包含有多数响应光照元件以及少量应对厌氧、缺氧、防御和应激、低温以及干旱的顺式作用元件。以上结果表明:RcF3H基因在辅助掌叶覆盆子响应激素和应对胁迫上发挥了重要作用,且可能受外界刺激后调控合成类黄酮物质。
表 2 掌叶覆盆子RcF3H基因启动子顺式作用元件分析
Table 2. Analysis of cis-regulatory elements of RcF3H promoter in R. chingii
类别 元件名称 数量 功能 植物生长发育 Circadian 1 参与昼夜节律控制的顺式调控元件 O2-site 2 玉米蛋白代谢调控的顺式调控元件 激素响应 AuxRE 1 生长素反应元件的一部分 CGTCA-motif 1 参与茉莉酸反应性的顺式作用元件 TGACG-motif 1 参与茉莉酸反应性的顺式作用元件 MYC 6 激素响应元件 P-box 1 赤霉素响应元件 Myb 1 MYB识别位点 MYB 1 MYB识别位点 MYB-like sequence 1 MYB识别位点 胁迫响应 ARE 3 对厌氧诱导必要的顺式调节元件 GC-motif 2 参与缺氧特异性诱导性的增强子元件 LTR 1 参与低温反应的顺式元件 MBS 1 MYB结合位点参与干旱诱导 STRE 1 胁迫响应元件 as-1 1 胁迫响应元件 TC-rich repeats 1 参与防御和应激反应的顺式作用元件 Box 4 2 参与光反应的保守DNA模块 GATA-motif 1 光响应元件的一部分 GT1-motif 3 光响应元件 Sp1 1 光响应元件 W box 1 诱导子响应元件 -
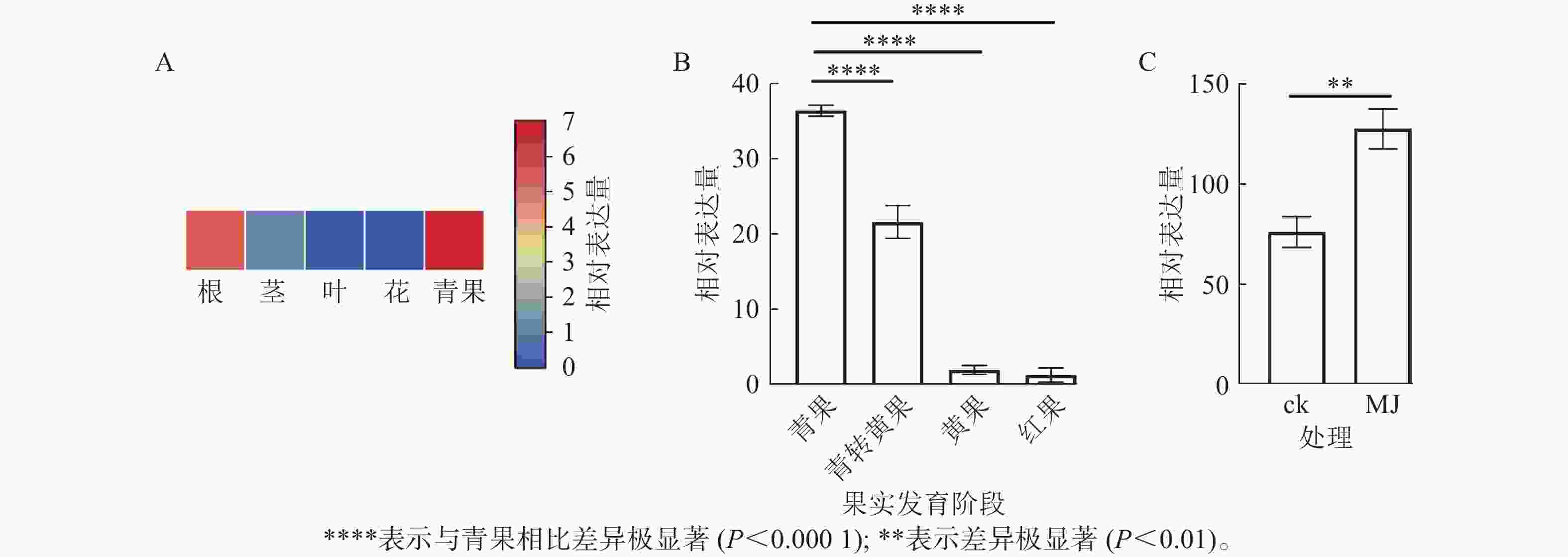
由图8A可知:RcF3H基因在掌叶覆盆子的根、茎、叶和果实中表达;其中青果中的相对表达量最高,分别是根的1.18倍,茎的5.05倍,叶片的46.00倍。表明RcF3H可能与掌叶覆盆子果实的生长发育有关。进一步分析在果实发育成熟过程中RcF3H基因的相对表达量。由图8B可知:RcF3H基因表达在青果期表达量最高,并在发育成熟过程表达量逐渐下降,该趋势与类黄酮质量分数变化一致[23]。启动子顺式作用元件分析发现:掌叶覆盆子RcF3H基因前2 000 bp启动子上存在响应MeJA的顺式作用元件(表2)。结合RT-qPCR技术发现:掌叶覆盆子RcF3H基因经过MeJA诱导处理后基因表达明显上升(图8C)。
-
本研究从掌叶覆盆子cDNA中克隆得到了完整的RcF3H基因序列,该基因由1条外显子构成,全长1 098 bp,编码了365个氨基酸。蛋白序列比对后发现:掌叶覆盆子RcF3H蛋白与蔷薇科插田泡等6种植物F3H的同源关系较近。蛋白质是细胞的功能分子,基因功能的主要执行者。掌叶覆盆子RcF3H拥有 2OG-Fe(Ⅱ)-Oxy结构以及2个典型的PLN02515和Pcb C保守结构域,亚细胞定位预测该蛋白位于细胞质中。一些蛋白质需要转移至特定的位置后才能发挥作用,信号肽能引导新合成的蛋白质向分泌通路转移[24]。RcF3H蛋白为稳定的亲水性蛋白,蛋白二级和三级结构分析发现:RcF3H主要以α-螺旋和无规则卷曲缠绕形成。系统进化树分析发现:RcF3H蛋白与单子叶植物亲缘关系较远,与双子叶植物亲缘关系较近且处于同一亚组,说明在进化过程中RcF3H基因较保守。
转录调控对基因表达起着很大的作用,主要通过基因的启动子及其相关的顺式作用元件控制[25]。对RcF3H启动子分析发现:其拥有茉莉酸、赤霉素和生长素等响应元件。RcF3H基因在MeJA处理7 d后表达量上调了1.68倍。在拟南芥中,茉莉酸和脱落酸能够调控类黄酮生物合成中AtF3H的表达[26]。MeJA处理绿熟期的樱桃番茄S. lycopersicum ‘Xin Taiyang’果实后,促进了类黄酮的积累,其类黄酮代谢途径中PAL1、C4H以及F3H等基因表达水平上调[27]。掌叶覆盆子RcF3H启动子还存在逆境胁迫响应有关的作用元件。黄花红砂在受到UV-B和干旱胁迫后,其RsF3H酶活性增加,类黄酮生物合成产物积累增多[28]。说明RcF3H基因可能在激素和逆境胁迫上发挥作用,调节类黄酮物质的合成来提高对环境适应性。
进一步探究掌叶覆盆子RcF3H表达与类黄酮的关系。RcF3H在掌叶覆盆子的根、茎、叶和果实中表达且具有组织特异性。RcF3H在青果中表达量最高,并在果实成熟过程中表达量逐渐下降,推测RcF3H可能与果实发育有关。随着掌叶覆盆子果实发育成熟,椴树苷和山奈酚-3-O-芸香糖苷等各种黄酮化合物会逐渐减少[23],这与RcF3H表达变化趋势一致,推测RcF3H可能促进类黄酮的积累。一些研究报道了MeJA能促进类黄酮的生物合成[26−27]。掌叶覆盆子RcF3H能响应外源MeJA的刺激,其表达量相较于对照组上升了1.68倍,而MeJA促进掌叶覆盆子类黄酮代谢未见报道。综上所述,掌叶覆盆子RcF3H在多个组织器官中发挥生物学功能,并能影响类黄酮的积累。此外,具体的生物学功能验证以及调控机制解析也有待进一步研究。
-
本研究在掌叶覆盆子中鉴定克隆了2-酮戊二酸依赖性双加氧酶家族基因RcF3H,该基因蛋白定位于细胞质,与双子叶植物同源性较高。该基因启动子上存在大量与激素和逆境响应相关的元件。RcF3H在多个器官中均有表达且具有组织特异性,并随果实成熟表达量逐渐下降。外源MeJA的刺激处理可提高RcF3H基因的表达。本研究初步解析了RcF3H在掌叶覆盆子类黄酮物质积累中的作用,为进一步研究其分子机制提供参考。
Cloning and expression analysis of RcF3H in Rubus chingii
-
摘要:
目的 黄烷酮3-羟化酶(F3H)是植物类黄酮合成的关键酶。研究掌叶覆盆子Rubus chingii RcF3H基因与类黄酮代谢的关系并分析其生物学功能,可为进一步探究RcF3H在掌叶覆盆子类黄酮物质积累过程中的作用机制提供参考。 方法 在掌叶覆盆子中克隆RcF3H基因,并进行生物信息学分析,包括理化性质、同源性比对、系统进化树和启动子顺式作用元件等。检测RcF3H基因在不同组织器官、果实生长时期以及在外源茉莉酸甲酯(MeJA)诱导下的表达水平。 结果 2-酮戊二酸依赖性双加氧酶(2-ODD)家族基因RcF3H位于第一条染色体,片段长度为1 098 bp,由1个外显子构成,共编码了365个氨基酸。RcF3H属于亲水性稳定蛋白,亚细胞预测定位在细胞质上。RcF3H蛋白的二、三级结构主要由α-螺旋和无规则卷曲组成,与插田泡Rubus coreanus及拟南芥 Arabidopsis thaliana等双子叶植物的亲缘关系最近。RcF3H基因启动子区域含有多个顺式作用元件,功能集中于响应激素和应对胁迫等方面。RcF3H基因在掌叶覆盆子果实中表达量较高,有明显的组织特异性。RcF3H基因表达变化与类黄酮物质在掌叶覆盆子果实中积累规律一致,并在100 μmol·L−1 MeJA处理下表达量上升。 结论 RcF3H 响应外源MeJA刺激,并可能作为掌叶覆盆子类黄酮生物合成的正向调节因子,影响类黄酮的积累。图8表2参28 Abstract:Objective Flavanone 3-hydroxylase (F3H) is a key enzyme in the synthesis of plant flavonoids. This study aims to investigate the relationship between RcF3H gene and flavonoid metabolism in Rubus chingii and analyze its biological function, so as to provide reference for further exploring the mechanism of RcF3H in the process of flavonoid accumulation in R. chingii. Method RcF3H gene was cloned from R. chingii, and bioinformatics analysis was performed, including physicochemical properties, homology comparison, phylogenetic tree and promoter cis-acting elements. At the same time, this study detected the expression level of RcF3H gene in different tissues, fruit growth period and exogenous methyl jasmonate (MeJA) induction. Result RcF3H gene, belonging to the 2-oxoglutarate-dependent dioxygenase superfamily, was located on the first chromosome, and its fragment length was 1098 bp. RcF3H gene consisted of 1 exon and encoded 365 amino acids. RcF3H belonged to the hydrophilic stable protein. Subcellular localization predicted that the protein was located in the cytoplasm. The secondary and tertiary structure of RcF3H was mainly composed of α-helix and irregular curl, which had the closest genetic relationship with dicotyledons such as Rubus coreanus and Arabidopsis thaliana. Analysis of promoter cis-acting elements showed that the promoter region of RcF3H gene contained multiple cis-acting elements, and its function mainly focused on responding to hormones and stress. RcF3H gene was highly expressed in fruits, and had obvious tissue specificity. In addition, the expression of RcF3H gene in different ripening stages of fruits was consistent with the accumulation of flavonoids, and the expression increased under the treatment of 100 μmol·L−1 MeJA. These results suggested that RcF3H gene is the key enzyme to promoting flavonoid synthesis.Conclusion RcF3H gene responds to MeJA stimulation and may act as a positive regulator of flavonoid biosynthesis in R. chingii, affecting flavonoid accumulation. [Ch, 8 fig. 2 tab. 28 ref.] -
Key words:
- Rubus chingii /
- flavanone 3-hydroxylase /
- gene cloning /
- expression analysis /
- flavonoids
-
表 1 引物序列
Table 1. Primer sequence for this study
引物名称 引物序列(5′→3′) 引物用途 β-actin-F ATCCACGAGACTACATACAACTCC 内参基因 β-actin-R CTGTCTGCAATACCAGGGAAC RcF3H-F ATGGCTCCTACACCTACTAC 序列扩增 RcF3H-R AGCAAAAATACCATCCACTT RcF3H-qPCR-F CAAAGTGGCCTACAACCAATTC 荧光定量 RcF3H-qPCR-R CCTCGACAATCTTCTTGCAAATC 表 2 掌叶覆盆子RcF3H基因启动子顺式作用元件分析
Table 2. Analysis of cis-regulatory elements of RcF3H promoter in R. chingii
类别 元件名称 数量 功能 植物生长发育 Circadian 1 参与昼夜节律控制的顺式调控元件 O2-site 2 玉米蛋白代谢调控的顺式调控元件 激素响应 AuxRE 1 生长素反应元件的一部分 CGTCA-motif 1 参与茉莉酸反应性的顺式作用元件 TGACG-motif 1 参与茉莉酸反应性的顺式作用元件 MYC 6 激素响应元件 P-box 1 赤霉素响应元件 Myb 1 MYB识别位点 MYB 1 MYB识别位点 MYB-like sequence 1 MYB识别位点 胁迫响应 ARE 3 对厌氧诱导必要的顺式调节元件 GC-motif 2 参与缺氧特异性诱导性的增强子元件 LTR 1 参与低温反应的顺式元件 MBS 1 MYB结合位点参与干旱诱导 STRE 1 胁迫响应元件 as-1 1 胁迫响应元件 TC-rich repeats 1 参与防御和应激反应的顺式作用元件 Box 4 2 参与光反应的保守DNA模块 GATA-motif 1 光响应元件的一部分 GT1-motif 3 光响应元件 Sp1 1 光响应元件 W box 1 诱导子响应元件 -
[1] MATHESIUS U. Flavonoid functions in plants and their interactions with other organisms [J/OL]. Plants, 2018, 7 (2): 30[2024-04-05]. doi: 10.3390/plants7020030. [2] MA Gang, ZHANG Lancui, YAMAMOTO R, et al. Molecular characterization of a flavanone 3-hydroxylase gene from citrus fruit reveals its crucial roles in anthocyanin accumulation [J/OL]. BMC Plant Biology, 2023, 23 (1): 233[2024-04-05]. doi: 10.1186/s12870-023-04173-3. [3] AVNEESH K, BALJINDER S, KASHMIR S. Functional characterization of flavanone 3-hydroxylase gene from Phyllanthus emblica (L. ) [J]. Journal of Plant Biochemistry and Biotechnology, 2015, 24(4): 453 − 460. [4] 段玥彤, 王鹏年, 张春宝, 等. 植物黄烷酮-3-羟化酶基因研究进展[J]. 生物技术通报, 2022, 38(6): 27 − 33. DUAN Yuetong, WANG Pengnian, ZHANG Chunbao, et al. Research progress in plant flavanone-3-hydroxylase gene [J]. Biotechnology Bulletin, 2022, 38(6): 27 − 33. [5] FEI Jiang, WANG Jiayi, JIA Haifeng, et al. RNAi-Mediated silencing of the flavanone 3-hydroxylase gene and its effect on flavonoid biosynthesis in strawberry fruit [J]. Journal of Plant Growth Regulation, 2013, 32(1): 182 − 190. [6] TU Yanhua, LIU Fei, GUO Dandan, et al. Molecular characterization of flavanone 3-hydroxylase gene and flavonoid accumulation in two chemotyped safflower lines in response to methyl jasmonate stimulation [J/OL]. BMC Plant Biology, 2016, 16 (1): 132[2024-04-05]. doi: 10.1186/s12870-016-0813-5. [7] BONG G K, JEONG H K, JIYOUNG K, et al. Accumulation of flavonols in response to ultraviolet-B irradiation in soybean is related to induction of flavanone 3-beta-hydroxylase and flavonol synthase [J]. Molecules and Cells, 2008, 25(2): 247 − 252. [8] SINGH K, RANI A, KUMAR S, et al. An early gene of the flavonoid pathway, flavanone 3-hydroxylase, exhibits a positive relationship with the concentration of catechins in tea (Camellia sinensis) [J]. Tree Physiology, 2008, 28(9): 1349 − 1356. [9] ZHANG Huirong, ZHAO Lixia, WANG Jia, et al. Cloning and functional analysis of two flavanone-3-hydroxylase genes from Reaumuria trigyna [J]. Acta Physiologiae Plantarum, 2014, 36(5): 1221 − 1229. [10] 赵乐, 马利刚, 张金燕, 等. 独行菜LaF3H基因克隆、序列分析及原核表达[J]. 中草药, 2018, 49(23): 5626 − 5632. ZHAO Le, MA Ligang, ZHANG Jinyan, et al. Cloning, sequence analysis, and prokaryotic expression of LaF3H gene from Lepidium apetalum [J]. Chinese Traditional and Herbal Drugs, 2018, 49(23): 5626 − 5632. [11] SI Can, DONG Wei, da SILVA J A T, et al. Functional analysis of flavanone 3-hydroxylase (F3H) from Dendrobium officinale, which confers abiotic stress tolerance [J]. Horticultural Plant Journal, 2023, 9(2): 356 − 364. [12] LI Wenfang, NING Gaixing, ZUO Cunwu, et al. MYB_SH[AL]QKY[RF] transcription factors MdLUX and MdPCL-like promote anthocyanin accumulation through DNA hypomethylation and MdF3H activation in apple [J]. Tree Physiology, 2020, 41 (5): 836 − 848. [13] ALMUTH H, DINESHKUMAR K, CHHANA U, et al. Flavanone-3-hydroxylase plays an important role in the biosynthesis of spruce phenolic defenses against bark beetles and their fungal associates [J/OL]. Frontiers in Plant Science, 2019, 10 : 208[2024-04-05]. doi: 10.3389/fpls.2019.00208. [14] MENG Chen, ZHANG Song, DENG Yongsheng, et al. Overexpression of a tomato flavanone 3-hydroxylase-like protein gene improves chilling tolerance in tobacco [J]. Plant Physiology and Biochemistry, 2015, 96: 388 − 400. [15] SHENG Jiayun, WANG Siqi, LIU Kaohua, et al. Rubus chingii Hu: an overview of botany, traditional uses, phytochemistry, and pharmacology [J]. Chinese Journal of Natural Medicines, 2020, 18 (6): 401 − 416. [16] HE Beihui, DAI Linghao, JIN Li, et al. Bioactive components, pharmacological effects, and drug development of traditional herbal medicine Rubus chingii Hu (Fu-Pen-Zi) [J/OL]. Frontiers in Nutrition, 2023, 9 : 1052504[2024-04-05]. doi: 10.3389/fpls.2019.00208. [17] 李瑶晨, 范紫佩, 杨静, 等. 野生蔬菜功能性成分及其生物活性研究进展[J]. 浙江农林大学学报, 2022, 39(4): 913 − 922. LI Yaochen, FAN Zipei, YANG Jing, et al. Research progress on functional components and biological activities of wild edible vegetables [J]. Journal of Zhejiang A&F University, 2022, 39(4): 913 − 922. [18] WANG Longji, LEI Ting, HAN Guomin, et al. The chromosome-scale reference genome of Rubus chingii Hu provides insight into the biosynthetic pathway of hydrolyzable tannins [J]. The Plant Journal, 2021, 107(5): 1466 − 1477. [19] 卓娟, 侯丹, 林新春. 毛竹PhebHLH6基因克隆及表达分析 [J]. 浙江农林大学学报, 2023, 40(4): 731 − 737. ZHUO Juan, HOU Dan, LIN Xinchun. Cloning and expression analysis of PhebHLH6 gene from Phyllostachys edulis [J]. Journal of Zhejiang A&F University, 2023, 40(4): 731 − 737. [20] 尚林雪, 王群, 张国哲, 等. 紫薇LiCMB1基因的克隆及表达特性分析[J]. 浙江农林大学学报, 2023, 40(2): 330 − 337. SHANG Linxue, WANG Qun, ZHANG Guozhe, et al. Cloning and expression characteristics of LiCMB1 gene in Lagerstroemia indica [J]. Journal of Zhejiang A&F University, 2023, 40(2): 330 − 337. [21] 郑飞雄, 陈俊宇, 江林琪, 等. 掌叶覆盆子4CL基因家族鉴定及表达分析[J]. 农业生物技术学报., 2024, 32(2): 311 − 321. ZHENG Feixiong, CHEN Junyu, JIANG Linqi, et al. Genome-wide identification and expression analysis of 4CL gene family in Rubus chingii Hu [J]. Journal of Agricultural Biotechnology, 2024, 32(2): 311 − 321. [22] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the \begin{document}$2^{-\Delta\Delta Ct} $\end{document} [23] LI Xiaobai, JIANG Jingyong, CHEN Zhen, et al. Transcriptomic, proteomic and metabolomic analysis of flavonoid biosynthesis during fruit maturation in Rubus chingii Hu [J/OL]. Frontiers in Plant Science, 2021, 12 : 706667[2024-04-05]. doi: 10.3389/fpls.2021.706667. [24] 苏甜, 李子薇, 张新梅, 等. 黄瓜CsD27基因的克隆、生物信息学及表达分析[J/OL]. 分子植物育种, 2024-03-27[2024-04-05]. https://link.cnki.net/urlid/46.1068.S.20240326.1103.004. SU Tian, LI Ziwei, ZHANG Xinmei, et al. The cDNA cloning, bioinformatics and expression analysis of CsD27 gene in Cucumis sativus L. [J/OL]. Molecular Plant Breeding, 2024-03-27[2024-04-05]. https://link.cnki.net/46.1068.S.20240326.1103.004. [25] ZOU Cheng, SUN Kelian, MACKALUSO J D, et al. Cis-regulatory code of stress-responsive transcription in Arabidopsis thaliana [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108 (36): 14992 − 14997. [26] LORETI E, POVERO G, NOVI G, et al. Gibberellins, jasmonate and abscisic acid modulate the sucrose-induced expression of anthocyanin biosynthetic genes in Arabidopsis [J]. The New Phytologist, 2008, 179(4): 1004 − 1016. [27] TAO Xiaoya, WU Qiong, LI Jiayin, et al. Exogenous methyl jasmonate regulates phenolic compounds biosynthesis during postharvest tomato ripening [J/OL]. Postharvest Biology and Technology, 2022, 184 : 111760[2024-04-05]. doi: 10.1016/j.postharvbio.2021.111760. [28] LIU Meiling, LI Xinrong, LIU Yubing, et al. Regulation of flavanone 3-hydroxylase gene involved in the flavonoid biosynthesis pathway in response to UV-B radiation and drought stress in the desert plant, Reaumuria soongorica [J]. Plant Physiology and Biochemistry, 2013, 73: 161 − 167. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20240326







 下载:
下载: