-
黄酮类化合物又名类黄酮(flavonoids),是植物中分布最广、数量最多的一类重要次生代谢物质。根据结构差异和取代基的不同,黄酮类化合物主要可分为:黄酮醇(flavonols)、黄酮(flavones)、黄烷酮(flavanones)、花色素/花青素(anthocyanidins/anthocyanins)、异黄酮(isoflavones)以及黄烷醇/儿茶素(flavanols/catechins)等[1]。其中,黄酮醇是类黄酮中数量最多、分布最广泛的一类,已发现约1 700多种[2]。
黄酮醇作为植物中广泛存在的一类次生代谢物,在调控植物生长发育、花色形成及逆境响应等过程中扮演着关键角色。与此同时,黄酮醇也具有重要的药理活性,可通过调节多种生理过程发挥抗过敏、抗炎及抗癌等作用[3]。鉴于黄酮醇在植物生理与人类健康领域的重要作用,解析黄酮醇生物合成的调控机制已成为当前的研究热点。其中,MYB转录因子因其在苯丙烷代谢通路中的核心作用而备受关注。MYB转录因子是植物中数量最多、功能最多的转录因子家族之一。它们通过直接或者与其他转录因子形成复合体等方式,调控黄酮醇合成关键基因的表达,以控制黄酮醇的生物合成。正是通过这一调控网络,MYB得以广泛影响植物的多种生理过程,包括黄酮醇的积累、特定生长发育阶段的调控、细胞内信号传导以及对生物与非生物胁迫的响应等。
本研究综述了黄酮醇生物合成途径及其调控机制,重点总结了MYB转录因子的结构与功能、参与黄酮醇生物合成调控的方式以及环境胁迫对MYB转录因子调控黄酮醇生物合成的影响,阐述了MYB转录因子如何协同环境信号(光照、气温、其他非生物胁迫)与内部发育信号精细调控黄酮醇合成,以期为深入解析黄酮醇代谢调控网络、花色形成机制及植物非生物胁迫相应机制等提供理论基础。
-
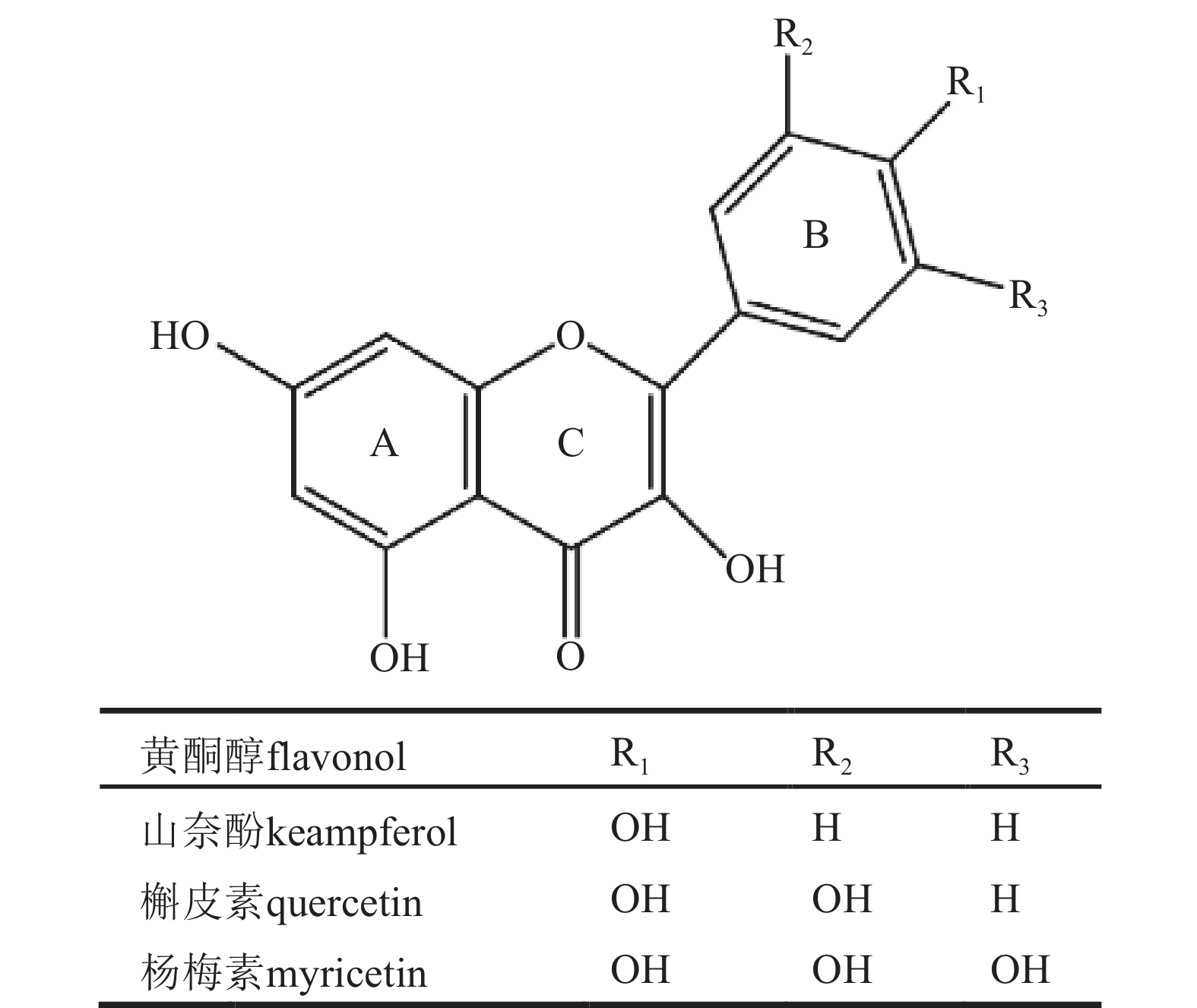
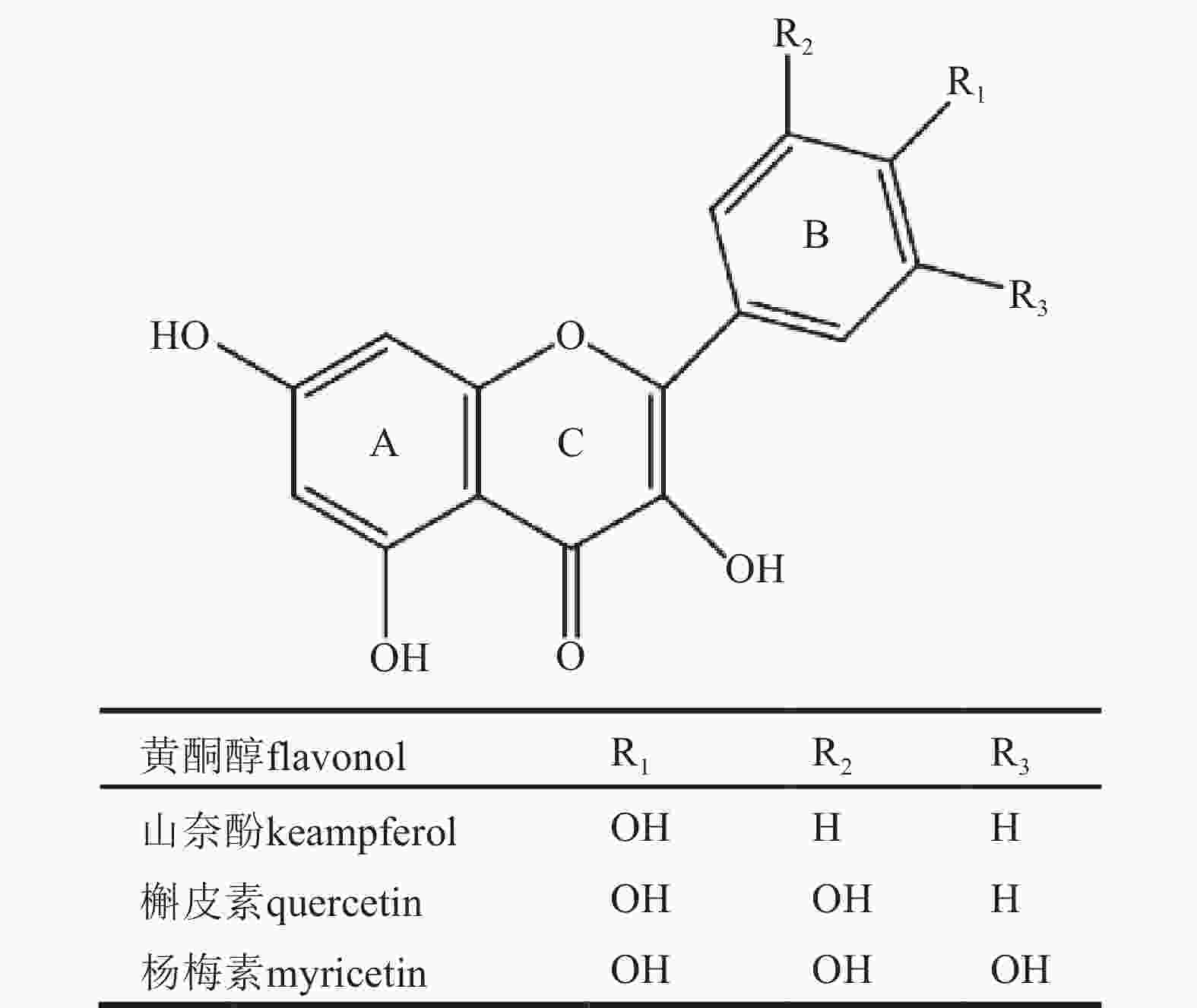
黄酮醇具有由15个碳原子(C6-C3-C6)构成的核心骨架,包含2个苯环(A环和B环)以及连接这2个苯环的1个含氧杂环(C环)。其C环的C2-C3位是双键(C=C)、C4位是羰基(C=O),最关键的是C环的C3位有1个羟基(—OH),这是其区别于其他黄酮类化合物最明显的特征。目前,已鉴定的天然黄酮醇苷元包括山奈酚(kaempferol)、槲皮素(quercetin)、杨梅素(myricetin)、杜鹃黄素(azaleatin)、漆黄素(fisetin)、柚皮黄素(naringenin)、鼠李素(rhamnetin)、桑色素(morin)等10余种,其中山奈酚、槲皮素及杨梅素最为常见,三者在结构上的主要区别在于B环的羟基化程度依次增加[4](图1)。在植物体内,黄酮醇极少以游离苷元形式存在,主要通过各种糖苷化修饰来增加其水溶性和结构多样性。糖配体可由C-糖苷键或O-糖苷键与黄酮醇苷元连接,连接的糖基主要包括葡萄糖、鼠李糖、半乳糖等单糖及由其构成的双糖、三糖甚至更复杂的寡糖链。此外,黄酮醇还能进一步发生酰基化、羟基化、甲基化等多种修饰[5−6]。黄酮醇3-羟基与4-羰基的邻位配位结构、B环取代基的数目与模式,以及其丰富的糖基化等结构修饰,共同构成了黄酮醇种类繁多、结构多变的特点。这些结构多样性赋予了黄酮醇一系列广泛而高效的生物学功能和药理活性。
黄酮醇在植物中普遍分布,其功能贯穿植物的整个生命周期。多种植物都含有丰富的黄酮醇,但其含量因植物种类和组织部位的不同而存在显著差异。通常,黄酮醇富集于植物表皮、叶片等暴露于阳光的部位。例如刺山柑Capparis spinosa和茶叶Camellia sinensis等植物的黄酮醇含量极高;而洋葱Allium cepa、韭菜A. tuberosum、西兰花Brassica oleracea var. italica、蔓越莓Vaccinium macrocarpon、蓝莓V. corymbosum等植物也含有较丰富的黄酮醇类物质,是人们日常生活中黄酮醇的重要摄取来源[7]。黄酮醇在调控植物花色形成、植物生长发育及响应环境胁迫等关键生理过程中发挥关键作用。黄酮醇作为植物花色呈现的“调色师”,能够通过代谢竞争等调控植物的花色,参与植物色素的变化和稳定性[8]。黄酮醇、黄酮醇苷等化合物的特征吸收峰位于紫外区(240~280 nm及350~385 nm),而在可见光区则基本无吸收,这一特性决定了它们通常呈现接近无色或淡黄色。在美洲莲Nelumbo lutea中检测到的主要类黄酮物质为黄酮醇类化合物,其中槲皮素-3-O-葡萄糖醛酸苷(quercetin-3-O-glucuronide)含量尤为丰富,约占所测总黄酮质量分数的90%~95%,是导致花瓣呈现黄色的主要色素成分[9]。对棉花Gossypium arboreum花瓣的色素分析表明:花瓣呈现的淡黄色或奶油色主要与黄酮醇类化合物的积累有关,以槲皮素-3-葡萄糖苷(quercetin-3-glucoside)和槲皮素-3-芸香糖苷(quercetin-3-rutinoside)等为主[10]。另外,黄酮醇也可以作为辅助色素,与花色苷协同调控花色。烟草Nicotiana tabacum中过表达黄酮醇合酶基因(FLS)可催化更多的二氢黄酮醇合成黄酮醇,竞争花青素合成途径的共同底物,导致花青素积累减少,从而使花色变淡[11]。作为重要的次生代谢物质,黄酮醇广泛存在于植物的各类组织与不同发育阶段,通过多种分子机制参与调控植物的生长发育及逆境响应过程。在生长发育方面,黄酮醇可通过调节生长素转运蛋白活性,维持根系中生长素分布的梯度稳定性,从而保障根系正常发育;在拟南芥Arabidopsis thaliana突变体中的研究表明:黄酮醇可通过影响生长素前体及其代谢物含量,调控胚胎生长与根系形态建成[12−13]。在逆境响应方面,黄酮醇在拟南芥中通过HY5-MYB12/111转录调控级联,发挥吸收紫外辐射与抗氧化的双重功能,是植物响应紫外线B (UV-B)胁迫的关键代谢产物[14];此外,黄酮醇还能调节抗坏血酸代谢,减轻叶绿体光损伤,维持光磷酸化途径的正常运转[15]。上述功能共同体现了黄酮醇在植物生命活动与环境适应中的重要作用。
在药理方面,黄酮醇已被广泛认为是促进人体健康和预防多种慢性疾病(心血管病、神经退行性疾病、2型糖尿病等)的有效活性物质[16−18]。黄酮醇经肠道菌群代谢后,产生短链脂肪酸(short-chain fatty acids, SCFAs)和酚酸衍生物[如3,4-二羟基苯乙酸(3,4-dihydroxyphenylacetic acid, DOPAC)],具有抗炎、调节肠道屏障功能和改善胰岛素敏感性的作用[19]。
-
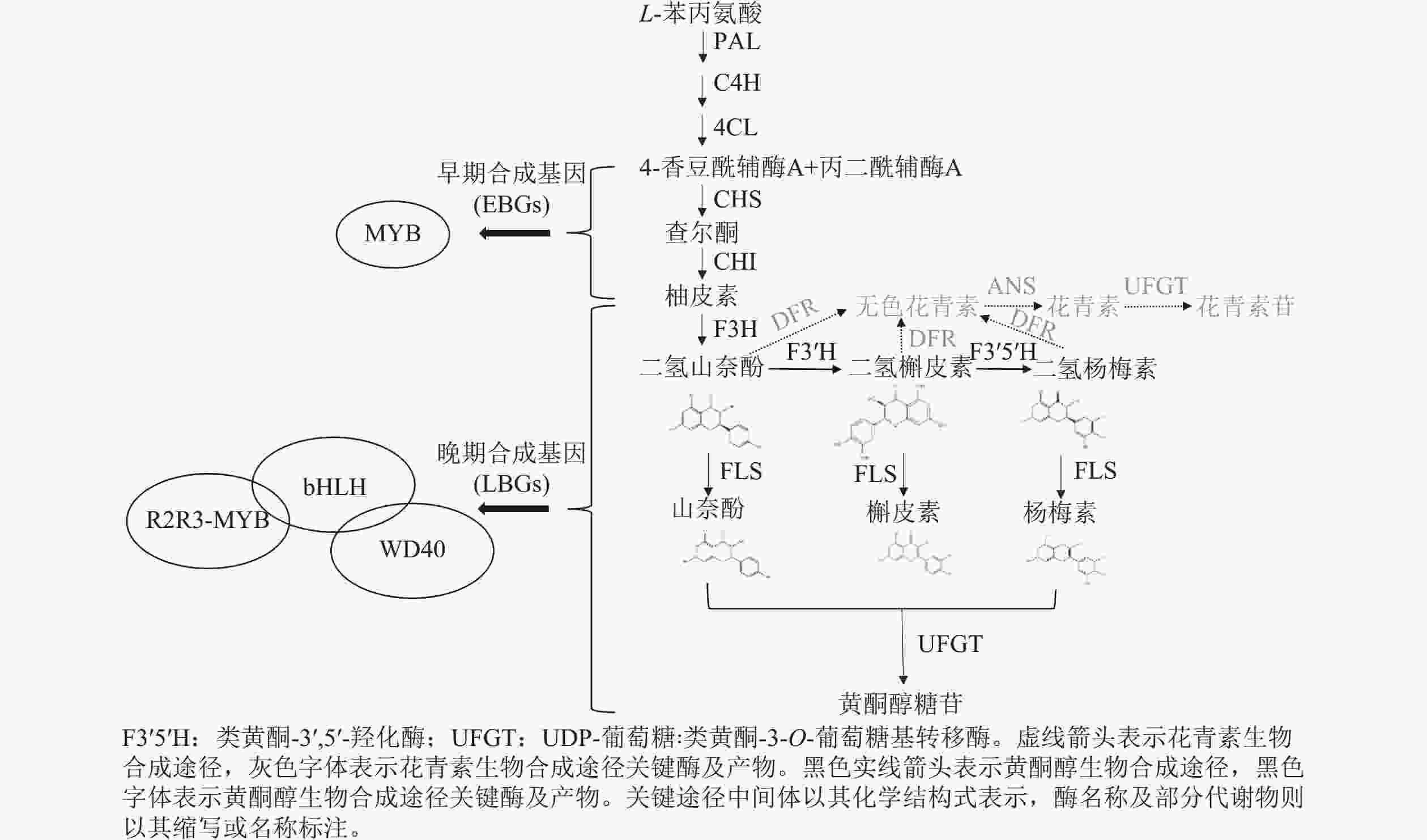
植物中黄酮醇等苯丙烷代谢产物的生物合成途径(图2)及其关键结构基因高度保守。苯丙烷生物合成途径以L-苯丙氨酸(3-phenyl-L-alanine)为起点,经由苯丙氨酸解氨酶(phenylalanine ammonia-lyase, PAL)、肉桂酸-4-羟化酶(cinnamic acid-4-hydroxylase, C4H)和4-香豆酸∶CoA连接酶(4-coumarate∶coenzyme A ligase, 4CL)的催化,生成4-香豆酰-CoA (p-coumaroyl-CoA)[20]。4-香豆酰-CoA经查尔酮合酶(chalcone synthase, CHS)、查尔酮异构酶(chalcone isomerase, CHI)、黄烷酮3-羟化酶(flavanone 3-hydroxylase, F3H)的催化形成二氢山奈酚(dihydrokaempferol, DHK)[21]。二氢山奈酚可以在类黄酮-3′-羟化酶(flavonoid 3′-hydroxylase, F3′H)和类黄酮-3′,5′-羟化酶(flavonoid 3′,5′-hydroxylase, F3′5′H)的作用下分别生成二氢槲皮素(dihydroquercetin, DHQ)和二氢杨梅素(dihydromyricetin, DHM),二氢山奈酚、二氢槲皮素和二氢杨梅素可以在二氢黄酮醇还原酶(dihydroflavonol 4-reductase)的催化下向花青素类物质的方向合成,也可以在二氢黄酮醇合酶的作用下直接形成山奈酚、槲皮素、杨梅素等黄酮醇物质[22]。在高等植物中,编码类黄酮合成关键酶的基因可分为2类:早期合成基因(early biosynthesis genes, EBGs),主要包括PAL、4CL、CHS、CHI、F3H;晚期合成基因(late biosynthesis genes, LBGs),主要包括FLS、F3'H、F3′5′H、UFGT。EBGs和LBGs的调节单元之间主要区别在于:早期合成基因主要受R2R3-MYB转录因子的独立调控,而晚期合成基因则主要由R2R3-MYB、bHLH和WD40重复蛋白组成的转录因子复合物调控[23]。
-
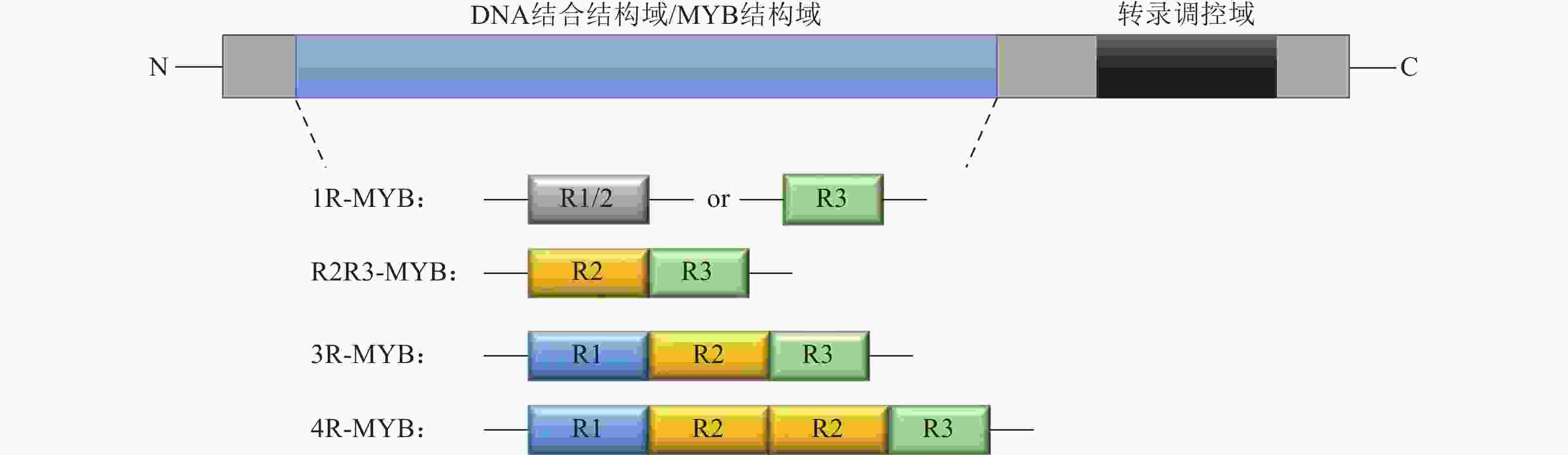
在拟南芥中,鉴定出1 600多个转录因子,约占整个基因组的6%[24]。其中MYB家族成员约占拟南芥转录因子家族总数的9%。MYB转录因子因其在N端保守的MYB结构域而命名,根据结合结构域所包含的R结构数目,将MYB转录因子分为4个亚类:1R-MYB、R2R3-MYB、3R-MYB和4R-MYB(图3)[25]。MYB转录因子具有广泛的生物学功能,参与植物的许多生命过程,如调节植物生长发育、参与细胞形态发生、调节初级和次级代谢反应、响应非生物和生物胁迫[26]。1R-MYB转录因子包含1个MYB结构域,主要参与次级代谢,包括苯丙烷生物合成、生物钟的调控以及营养器官的发育等[27−28];R2R3-MYB转录因子在MYB结合结构域中包含2个保守的R2和R3重复序列,同时在C末端可变区内包含1个调控结构域(激活或抑制功能),成员众多、功能多样,广泛参与细胞分化、次生代谢、环境胁迫以及抵御病虫害的侵袭[29];3R-MYB转录因子保守结构域由R1、R2和R3组成,主要存在于大部分真核生物中,参与调控细胞周期的动态变化和细胞分化[30];4R-MYB转录因子的保守结构域由4个R1/R2重复序列组成,目前在植物中发现的该亚家族基因数目还很少[31]。
近年来,植物中MYB转录因子对黄酮醇生物合成的调控已被广泛研究(表1)。MYB转录因子通过多种分子机制调控黄酮醇的生物合成,包括直接调控黄酮醇生物合成基因的表达、与其他类型转录因子形成复合调控模块,以及在环境因素介导下特异性诱导黄酮醇的合成积累。
表 1 植物中参与黄酮醇生物合成调控的MYB转录因子及其功能
Table 1. Representative MYB transcription factors involved in flavonol biosynthesis regulation across different plant species and their functions
物种 MYB转录因子 功能 靶基因 调控作用 参考文献 香蕉 Musa acuminata MaMYBFA 正向调节FLS,增加果实中黄酮醇含量 MaFLS2 正调控 [32] 鹰嘴豆 Cicer arietinum CaMYB39 促进黄酮醇的积累,增加黄酮醇含量 FLS 正调控 [33] 非洲菊 Gerbera hybrida GhMYB1a 促进山柰酚型黄酮醇积累,是形成典型黄色和象牙白花色品种的关键因子 GhCHS, GhF3H, GhFLS 正调控 [34] 棉花 Gossypium arboreum GaYP 上调黄酮醇合成,是花瓣产生黄色的必要基因 GaFLS 正调控 [35] 芍药 Paeonia qiui PqMYBF1 激活黄酮醇通路,将代谢通量从花青素引导至黄酮醇,导致花瓣中黄酮醇大量积累而花青素减少 PqCHS, PqF3H PqFLS 正调控 [36] 水仙 Narcissus tazetta NtMYB12 特异性促进副冠(花冠)中黄酮醇的合成,对副冠的黄色形成至关重要 NtFLS 正调控 [37] 水仙 NtMYB8 促进花冠中黄酮醇的合成,对副冠的黄色形成至关重要 NtDFR 正调控 [38] 番茄 Solanum lycopersicum SlMYB12 主要在果实表皮调控黄酮醇积累,影响果实颜色和抗氧化能力 SlCHS1, SlCHS2, SlFLS 正调控 [39] 草莓 Fragaria × ananassa FaMYB5/FaMYB10-like 在果实中部分调控黄酮醇积累,与FaMYB-10 (主控花青素)功能重叠,共同影响果实
颜色FaFLS 正调控 [40] 烟草 Nicotiana tabacum NTMYB12 上调相关合成基因 AtFLS, AtCHS, AtCHI 正调控 [41–42] 葡萄 Vitis vinifera VvMYBF1
VviMYBF1特异性调控果皮和叶片中黄酮醇苷合成。黄酮醇积累不仅影响品质,还在紫外线胁迫下保护光合组织,增强抗逆性 VvFLS4, VvFLS5,
VviFLS1, VviFLS4正调控 [43–44] 菊花 Chrysanthemum × morifolium CmMYB3a
CmMYB3b响应紫外线等环境信号,通过增加黄酮醇合成以保护植物免受紫外伤害 AtCHS, AtCHI,
AtF3H, AtFLS正调控 [45] 茶 Camellia sinensis CsMYBL2 调控黄酮醇的生物合成,影响茶叶品质 CsFLS, CsUGT 正调控 [46] 桃 Prunus persica PpMYB15
PpMYBF1主要在果实中表达,调控黄酮醇合成,影响果实的营养品质和抗氧化能力 PpCHS1, PpCHI1, PpF3H, PpFLS1 正调控 [47] 梨 Pyrus bretschneideri PbMYB9 结合果实中相关基因的启动子诱导黄酮醇的合成,影响果实的营养品质 PbUFGT1 正调控 [48] 海棠 Malus MdMYBB8 激活果实中MdFLS启动子,增加黄酮醇生物合成,影响果实的营养品质 MdFLS 正调控 [49] 拟南芥 Arabidopsis
thalianaMYB21 特异性调控雄蕊(花粉)中黄酮醇的积累。
对于花粉育性和正常花粉管萌发至关重要FLS 正调控 [50] 淫羊藿 Epimedium sagittatum EsMYB9 调控黄酮醇合成,可能与其药用活性成分的积累有关 NtFLS 正调控 [51] 三叶青 Tetrastigma hemsleyanum ThMYBPAR 激活内源黄酮醇生物合成基因的表达,增加黄酮含量 CHS, FLS 正调控 [52] 银杏 Ginkgo biloba GbMYB36 调控黄酮醇合成,银杏叶黄酮醇苷是著名的心脑血管药物活性成分 GbF3′H 正调控 [53] 白菜 Brassica rapa ssp. chinensis BCMYB111 以通过直接与黄酮醇生物合成相关的启动子结合来调节黄酮醇的合成 BcF3H, BcFLS1 正调控 [54] -
MYB转录因子通过直接靶向调控PAL、CHS、FLS等黄酮醇生物合成途径中的关键酶基因的转录水平,控制黄酮醇的生物合成。MYB转录因子的调控可以分为激活和抑制,这主要取决于其C末端结构域的序列特征。该区域通常包含特定的效应模块,负责招募转录共激活因子或共抑制因子复合物,从而介导对下游靶基因的正向或负向调控[55]。拟南芥中AtMYB11、AtMYB12和AtMYB111共同调控黄酮醇的生物合成。它们通过直接、特异性地激活黄酮醇合成途径的早期关键基因(PAL、CHS、CHI、F3H、FLS)的表达来实现这一功能[56]。在拟南芥幼苗中,AtMYB12和AtMYB111的表达具有组织特异性。AtMYB12主要在根中调控黄酮醇的合成与积累,而AtMYB111则主要在子叶中表达[57]。PARK等[58]研究发现:在荞麦Fagopyrum esculentum中AtMYB12不仅激活了黄酮醇合成途径的特异基因,还能显著提高上游苯丙烷途径基因PAL和4CL的表达。葡萄中MYB类转录因子VvMYBF1可以激活FLS1的表达,并成功恢复该突变体根中黄酮醇合成缺失的表型[59]。ZHAI等[48]研究表明:梨Pyrus brestchneideri的PbMYB12b通过激活下游基因PbCHSb和PbFLS的表达,促进槲皮素苷和异鼠李糖苷的生物合成。在朝鲜蓟Cynara cardunculus var. scolymus中发现:CcMYB12能直接结合黄酮醇合成途径关键酶基因(如FLS、CHI、F3H)启动子中的AC-Ⅱ顺式作用元件,导致黄酮醇含量显著升高[60]。玫瑰Rosa rugosa中的R2R3-MYB转录因子RrMYB12和RrMYB111可以激活CHS和FLS基因,增强黄酮醇代谢通路,使得花色淡化[61]。大豆Glycine max中GmMYB3a调控参与异黄酮、类黄酮和苯丙氨酸代谢的下游基因,尤其是关键的查尔酮合酶基因CHS7和CHS8,可以调节异黄酮生物合成[62]。
除正调控外,MYB转录因子也能参与负调控黄酮醇的积累。在大豆中GmMYB77抑制异黄酮合成途径的GmIFS和GmCHS,显著降低大豆中的类黄酮含量[63]。同时也有研究发现:大豆中的GmMYB100可以显著降低转基因大豆毛状根以及转基因拟南芥中的黄酮醇含量[64]。
-
除了直接调控结构基因的转录活性,MYB转录因子通常与bHLH、WD40等相互作用形成复合体共同调控黄酮醇的生物合成。黄酮醇途径中结构基因的表达是受转录因子调控的,其中涉及较多的转录因子是由MYB、bHLH和WD40所组成的MBW三元复合体(图4)。MBW三元复合体中MYB转录因子主要负责特异性结合靶基因启动子区的MYB结合元件(MRE),决定调控的特异性(如黄酮醇途径的MYB因子:AtMYB12)。bHLH作为桥梁蛋白,通过其碱性螺旋-环-螺旋结构域与MYB及WD40蛋白互作。WD40作为支架蛋白,通过β-螺旋桨结构稳定复合体构象,增强MYB-bHLH结合稳定性[65]。
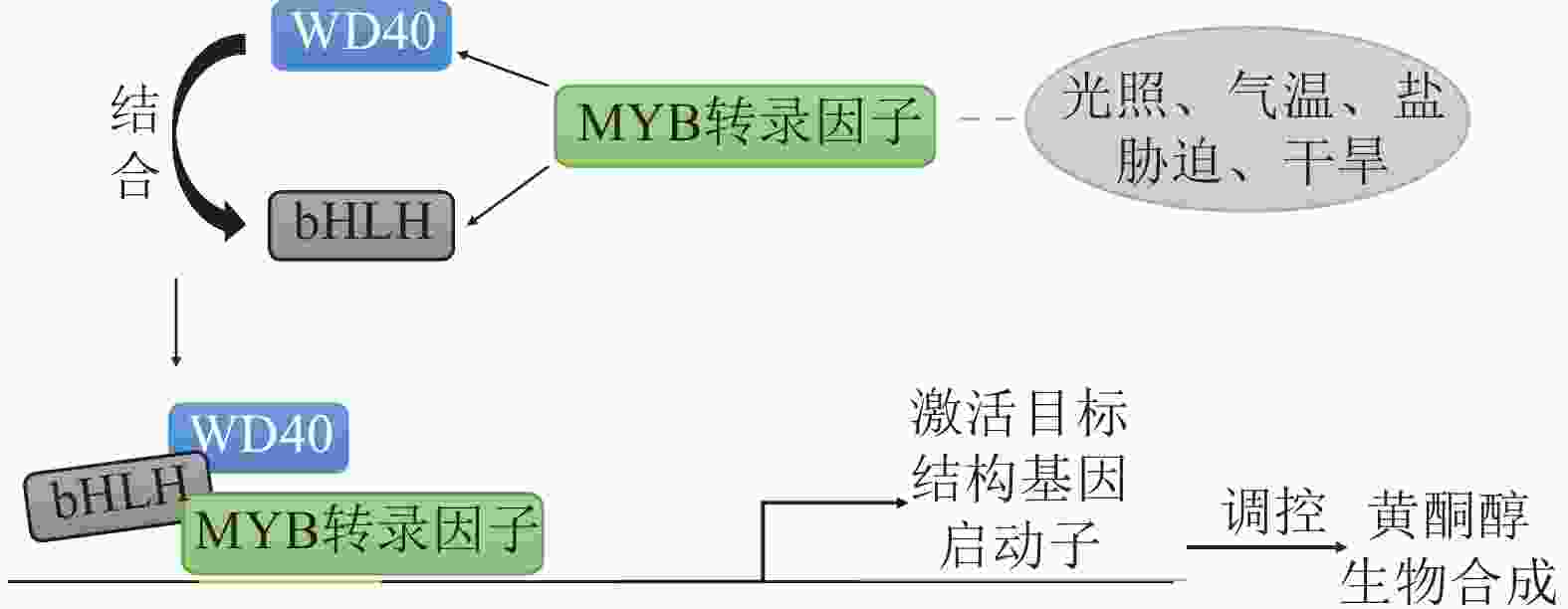
图 4 MYB转录因子调控黄酮醇生物合成机制
Figure 4. Mechanism of MYB transcription factors regulating flavonol biosynthesis
转录因子的调控方式分为正调控和负调控2种,MBW三元复合体的作用方式与之相同。拟南芥中3个密切相关的R2R3-MYB转录因子(AtMYB11、AtMYB12、AtMYB111)是黄酮醇合成的特异性正调节因子。它们与bHLH因子和WD40蛋白形成MBW复合体,直接结合并激活黄酮醇生物合成途径关键基因的启动子[56]。在玉米Zea mays中,bHLH转录因子LC/C1与MYB共同作用调节黄酮醇的生物合成,单独的LC或者MYB却没有这种效果[66]。在番茄果实中,关键转录因子SlMYB12作为SlMYC2-MED25复合物调节类黄酮代谢的直接靶点CHS、CHI、F3H和FLS1,直接影响番茄果实中的黄酮醇含量[67]。MYB转录因子也可以通过破坏激活型MBW三元复合体,抑制黄酮醇的合成。葡萄中VvMYBC2-L1/L2/L3为抑制型MYB,通过竞争性结合MBW三元复合体抑制FLS等基因表达[68]。
其他转录因子家族如WRKY、NAC、bZIP等部分成员也被证实能够与MYB转录因子相互作用,共同调控黄酮醇生物合成中关键基因的表达,进而影响植物黄酮醇合成。苹果Malus domestica中的NtWRKY11b可以与NtMYB12启动子区域相结合,调节烟草黄酮醇的生物合成[69]。银杏Ginkgo biloba中的LncNAT11可以通过抑制GbMYB11的表达来降低黄酮醇的生物合成[70]。茶树Camellia sinensis中的CsbZIP1可以正向激活CsMYB12,增加茶树中黄酮醇的合成[71]。
-
MYB转录因子在整合多种环境信号与黄酮醇合成通路中发挥着核心作用。多种非生物胁迫比如光照、气温、干旱、盐胁迫等均可通过MYB转录因子诱导黄酮醇积累,从而显著增强植物的逆境耐受性。一些环境因素(如光照、低温)作为一种强烈的信号,通过激活HY5或激素信号等上游调控因子,进而诱导或激活黄酮醇合成通路中的关键MYB转录因子,调控其功能与表达[72]。被激活的MYB转录因子进而上调黄酮醇合成途径中一系列关键酶基因的转录,最终导致黄酮醇类物质的显著积累。
MYB转录因子中的一些成员可以通过调控黄酮醇生物合成过程中的基因,调节黄酮醇的积累来响应UV-B。在苦荞Fagopyrum tataricum中,MYB4R1通过与FtCHS、FtFLS和FtUFGT启动子中的L-box基序结合,调节苦荞中响应UV-B的黄酮醇和花青素积累[73]。
气温是影响植物生长发育的关键环境因子。低温条件下BCMYB111响应BcCBF2并诱导黄酮醇的生物合成,增强无穗白菜在冷胁迫下的耐受性[54]。拟南芥AtMYB12通过增加黄酮醇的合成,增强植株对寒冷的耐受性[74]。野菊Chrysanthemum indicum CiMYB3通过调控黄酮醇的生物合成,抵御寒冷胁迫[75]。
干旱和盐胁迫通常都会对植物造成渗透胁迫,诱导相似的防御反应。黄芪Astragalus membranaceus的毛状根中,AmMYB35与AmFLS启动子结合促进了黄酮醇的生物合成,黄酮醇通过清除胁迫引起的活性氧(ROS)提高耐旱性[76]。AtMYB111正向调控植株的耐盐性。研究发现:AtMYB111通过直接结合F3H、FLS1、CHS这些类黄酮合成基因的启动子来促进这些基因的转录,从而具有耐盐胁迫的作用[77]。在盐碱胁迫下,紫花苜蓿Medicago sativa通过脱落酸(ABA)信号激活MsMYB12-MsFLS13通路,主动促进黄酮醇的合成与积累,增强自身的抗氧化能力并维持光合作用效率[78]。
-
植物中的黄酮醇合成调控是一个非常复杂的过程,涉及一系列黄酮醇生物合成关键酶基因的表达,并受到MYB、WRKY、bHLH等转录因子的调控。MYB转录因子在黄酮醇生物合成机制中占据重要的地位。MYB转录因子的翻译后修饰、miRNA对其mRNA的稳定性调控,以及黄酮醇代谢物本身对其合成途径的反馈调节仍需要深入探索。随着功能基因组学与多组学技术的快速发展,当前的研究大多通过系统进化分析、顺式元件鉴定及共表达网络构建等生物信息学方法对未知MYB转录因子进行功能预测,进而利用分子生物学实验加以验证。在此推动下,多个物种中调控黄酮醇生物合成的关键MYB转录因子成员(如拟南芥中的AtMYB11/12/111、番茄中的SlMYB12等)被成功鉴定。然而,目前关于MYB转录因子对黄酮醇生物合成的研究大多集中于模式植物、经济作物和小部分非模式植物上,对于大多数非模式植物的黄酮醇合成的相关转录因子功能尚未发现报道。对转录因子之间的互作模式及其对结构基因的调控网络仍需深入研究,以系统揭示它们对黄酮醇生物合成的调控机制。
此外,环境胁迫对黄酮醇积累具有显著影响,这类胁迫通常通过激活MYB转录因子促进黄酮醇合成,增强植物的环境适应性。目前关于环境因子如何影响植物体内黄酮醇合成与积累的研究仍相对匮乏,尤其是MYB转录因子在感知环境信号后调控黄酮醇合成的分子机制。MYB转录因子调控黄酮醇生物合成的调控网络精密而复杂,其中仍有诸多关键问题悬而未决。尤其在复合胁迫条件下,不同调控机制如何协同作用,是当前研究的难点与前沿。
Research progress on the regulation of flavonol biosynthesis in plants by MYB transcription factors
-
摘要: 黄酮醇是植物界分布最广、含量最丰富的类黄酮化合物。黄酮醇在植物的生长发育、抵御逆境及适应胁迫环境等方面均发挥着重要作用,同时它拥有强大的抗氧化功能,这对于人体健康具有重要意义。鉴于黄酮醇的多重价值,解析其生物合成途径的调控机制已成为研究热点。MYB转录因子作为植物最大转录因子家族之一,在黄酮醇的生物合成中发挥着核心调控作用。MYB既能直接调控下游靶基因,也可与bHLH、WD40蛋白形成MBW复合体进行协同调控,并能整合光照、气温、干旱等环境信号,从而实现对黄酮醇合成的精细控制。本研究概述了黄酮醇的生理功能与生物合成途径,重点阐述了MYB转录因子的结构特征及调控黄酮醇合成的分子机制,系统归纳了不同物种中参与黄酮醇调控的关键MYB转录因子成员及其功能,并讨论了当前研究的局限性与未来的研究方向,旨在为作物营养强化与花卉花色创新提供理论支撑。图4表1参78Abstract: Flavonols are the most widely distributed and abundant flavonoids in the plant kingdom. They play crucial roles in plant growth and development, stress resistance, and adaptation to environmental challenges, while also possessing potent antioxidant properties that are of significant importance to human health. Given the multiple values of flavonols, elucidating the regulatory mechanisms of their biosynthetic pathways has become a research hotspot. As one of the largest families of transcription factors in plants, MYB transcription factors play a central regulatory role in the biosynthesis of flavonols. MYB can directly regulate downstream target genes, collaborate with bHLH and WD40 proteins to form MBW complexes for coordinated regulation, and integrate environmental signals such as light, temperature, and drought to achieve precise control of flavonol synthesis. This paper reviewed the physiological functions and biosynthetic pathways of flavonols, with a particular focus on the structural characteristics of MYB transcription factors and their molecular mechanisms in regulating flavonol synthesis, summarized key MYB transcription factor members involved in flavonol regulation across different species and their functions, while addressing the limitations of current research and outlining future directions, aiming to provide a theoretical basis for improving the nutritional value of crops and breeding new cultivars of flowers with novel colors. [Ch, 4 fig. 1 tab. 78 ref.]
-
Key words:
- flavonoids /
- flavonols /
- MYB transcription factors /
- biosynthesis /
- transcriptional regulation /
- review
-
表 1 植物中参与黄酮醇生物合成调控的MYB转录因子及其功能
Table 1. Representative MYB transcription factors involved in flavonol biosynthesis regulation across different plant species and their functions
物种 MYB转录因子 功能 靶基因 调控作用 参考文献 香蕉 Musa acuminata MaMYBFA 正向调节FLS,增加果实中黄酮醇含量 MaFLS2 正调控 [32] 鹰嘴豆 Cicer arietinum CaMYB39 促进黄酮醇的积累,增加黄酮醇含量 FLS 正调控 [33] 非洲菊 Gerbera hybrida GhMYB1a 促进山柰酚型黄酮醇积累,是形成典型黄色和象牙白花色品种的关键因子 GhCHS, GhF3H, GhFLS 正调控 [34] 棉花 Gossypium arboreum GaYP 上调黄酮醇合成,是花瓣产生黄色的必要基因 GaFLS 正调控 [35] 芍药 Paeonia qiui PqMYBF1 激活黄酮醇通路,将代谢通量从花青素引导至黄酮醇,导致花瓣中黄酮醇大量积累而花青素减少 PqCHS, PqF3H PqFLS 正调控 [36] 水仙 Narcissus tazetta NtMYB12 特异性促进副冠(花冠)中黄酮醇的合成,对副冠的黄色形成至关重要 NtFLS 正调控 [37] 水仙 NtMYB8 促进花冠中黄酮醇的合成,对副冠的黄色形成至关重要 NtDFR 正调控 [38] 番茄 Solanum lycopersicum SlMYB12 主要在果实表皮调控黄酮醇积累,影响果实颜色和抗氧化能力 SlCHS1, SlCHS2, SlFLS 正调控 [39] 草莓 Fragaria × ananassa FaMYB5/FaMYB10-like 在果实中部分调控黄酮醇积累,与FaMYB-10 (主控花青素)功能重叠,共同影响果实
颜色FaFLS 正调控 [40] 烟草 Nicotiana tabacum NTMYB12 上调相关合成基因 AtFLS, AtCHS, AtCHI 正调控 [41–42] 葡萄 Vitis vinifera VvMYBF1
VviMYBF1特异性调控果皮和叶片中黄酮醇苷合成。黄酮醇积累不仅影响品质,还在紫外线胁迫下保护光合组织,增强抗逆性 VvFLS4, VvFLS5,
VviFLS1, VviFLS4正调控 [43–44] 菊花 Chrysanthemum × morifolium CmMYB3a
CmMYB3b响应紫外线等环境信号,通过增加黄酮醇合成以保护植物免受紫外伤害 AtCHS, AtCHI,
AtF3H, AtFLS正调控 [45] 茶 Camellia sinensis CsMYBL2 调控黄酮醇的生物合成,影响茶叶品质 CsFLS, CsUGT 正调控 [46] 桃 Prunus persica PpMYB15
PpMYBF1主要在果实中表达,调控黄酮醇合成,影响果实的营养品质和抗氧化能力 PpCHS1, PpCHI1, PpF3H, PpFLS1 正调控 [47] 梨 Pyrus bretschneideri PbMYB9 结合果实中相关基因的启动子诱导黄酮醇的合成,影响果实的营养品质 PbUFGT1 正调控 [48] 海棠 Malus MdMYBB8 激活果实中MdFLS启动子,增加黄酮醇生物合成,影响果实的营养品质 MdFLS 正调控 [49] 拟南芥 Arabidopsis
thalianaMYB21 特异性调控雄蕊(花粉)中黄酮醇的积累。
对于花粉育性和正常花粉管萌发至关重要FLS 正调控 [50] 淫羊藿 Epimedium sagittatum EsMYB9 调控黄酮醇合成,可能与其药用活性成分的积累有关 NtFLS 正调控 [51] 三叶青 Tetrastigma hemsleyanum ThMYBPAR 激活内源黄酮醇生物合成基因的表达,增加黄酮含量 CHS, FLS 正调控 [52] 银杏 Ginkgo biloba GbMYB36 调控黄酮醇合成,银杏叶黄酮醇苷是著名的心脑血管药物活性成分 GbF3′H 正调控 [53] 白菜 Brassica rapa ssp. chinensis BCMYB111 以通过直接与黄酮醇生物合成相关的启动子结合来调节黄酮醇的合成 BcF3H, BcFLS1 正调控 [54] -
[1] XING Mengyun, CAO Yunlin, GRIERSON D, et al. The chemistry, distribution, and metabolic modifications of fruit flavonols [J]. Fruit Research, 2021, 1(1): 1−11. [2] ZHUANG Weibing, LI Yuhang, SHU Xiaochun, et al. The classification, molecular structure and biological biosynthesis of flavonoids, and their roles in biotic and abiotic stresses[J/OL]. Molecules, 2023, 28(8): 3599[2025-08-01]. DOI: 10.3390/molecules28083599. [3] GERVASI T, CALDERARO A, BARRECA D, et al. Biotechnological applications and health-promoting properties of flavonols: an updated view[J/OL]. International Journal of Molecular Sciences, 2022, 23(3): 1710[2025-08-01]. DOI: 10.3390/ijms23031710. [4] WEN Weiwei, ALSEEKH S, FERNIE A R. Conservation and diversification of flavonoid metabolism in the plant Kingdom [J]. Current Opinion in Plant Biology, 2020, 55: 100−108. [5] LIU Weixin, FENG Yi, YU Suhang, et al. The flavonoid biosynthesis network in plants[J/OL]. International Journal of Molecular Sciences, 2021, 22(23): 12824[2025-08-01]. DOI: 10.3390/ijms222312824. [6] NEUGART S, FIOL M, SCHREINER M, et al. Interaction of moderate UV-B exposure and temperature on the formation of structurally different flavonol glycosides and hydroxycinnamic acid derivatives in kale (Brassica oleracea var. sabellica) [J]. Journal of Agricultural and Food Chemistry, 2014, 62(18): 4054−4062. [7] PARMENTER B H, THOMPSON A S, BONDONNO N P, et al. High diversity of dietary flavonoid intake is associated with a lower risk of all-cause mortality and major chronic diseases [J]. Nature Food, 2025, 6(7): 668−680. [8] SHIONO M, MATSUGAKI N, TAKEDA K. Structure of the blue cornflower pigment[J/OL]. Nature, 2005, 436(7052): 791[2025-08-01]. DOI: 10.1038/436791a. [9] LIU Qingqing, ZHANG Dasheng, LIU Fengluan, et al. Quercetin-derivatives painting the yellow petals of American lotus (Nelumbo lutea) and enzymatic basis for their accumulation [J]. Horticultural Plant Journal, 2023, 9(1): 169−182. [10] CAI Caiping, ZHOU Fan, LI Weixi, et al. The R2R3-MYB transcription factor GaPC controls petal coloration in cotton [J]. The Crop Journal, 2023, 11(5): 1319−1330. [11] PARK S, KIM D H, YANG J H, et al. Increased flavonol levels in tobacco expressing AcFLS affect flower color and root growth[J/OL]. International Journal of Molecular Sciences, 2020, 21(3): 1011[2025-08-01]. DOI: 10.3390/ijms21031011. [12] GRUNEWALD W, de SMET I, LEWIS D R, et al. Transcription factor WRKY23 assists auxin distribution patterns during Arabidopsis root development through local control on flavonol biosynthesis [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(5): 1554−1559. [13] KUHN B M, ERRAFI S, BUCHER R, et al. 7-rhamnosylated flavonols modulate homeostasis of the plant hormone auxin and affect plant development [J]. Journal of Biological Chemistry, 2016, 291(10): 5385−5395. [14] STRACKE R, FAVORY J J, GRUBER H, et al. The Arabidopsis bZIP transcription factor HY5 regulates expression of the PFG1/MYB12 gene in response to light and ultraviolet-B radiation [J]. Plant, Cell & Environment, 2010, 33(1): 88−103. [15] MATTILA H, KHOROBRYKH S, TYYSTJÄRVI E. Flavonols do not affect aphid load in green or senescing birch leaves but coincide with a decrease in Photosystem Ⅱ functionality[J/OL]. Biology Open, 2024, 13(7): bio060325[2025-08-01]. DOI: 10.1242/bio.060325. [16] PANCHE A N, DIWAN A D, CHANDRA S R. Flavonoids: an overview[J/OL]. Journal of Nutritional Science, 2016, 5: e47[2025-08-01]. DOI: 10.1017/jns.2016.41. [17] FATIMA N, BAQRI S S R, BHATTACHARYA A, et al. Role of flavonoids as epigenetic modulators in cancer prevention and therapy[J/OL]. Frontiers in Genetics, 2021, 12: 758733[2025-08-01]. DOI: 10.3389/fgene.2021.758733. [18] CHU Tianjiao, WANG Yuman, WANG Shihao, et al. Kaempferol regulating macrophage foaming and atherosclerosis through Piezo1-mediated MAPK/NF-κB and Nrf2/HO-1 signaling pathway [J]. Journal of Advanced Research, 2025, 75: 635−650. [19] CORTÉS-MARTÍN A, SELMA M V, TOMÁS-BARBERÁN F A, et al. Where to look into the puzzle of polyphenols and health? the postbiotics and gut microbiota associated with human metabotypes[J/OL]. Molecular Nutrition & Food Research, 2020, 64(9): 1900952[2025-08-01]. DOI: 10.1002/mnfr.201900952. [20] VOGT T. Phenylpropanoid biosynthesis [J]. Molecular Plant, 2010, 3(1): 2−20. [21] ACHNINE L, BLANCAFLOR E B, RASMUSSEN S, et al. Colocalization of L-phenylalanine ammonia-lyase and cinnamate 4-hydroxylase for metabolic channeling in phenylpropanoid biosynthesis [J]. The Plant Cell, 2004, 16(11): 3098−3109. [22] WINKEL-SHIRLEY B. Flavonoid biosynthesis. a colorful model for genetics, biochemistry, cell biology, and biotechnology [J]. Plant Physiology, 2001, 126(2): 485−493. [23] PETRONI K, TONELLI C. Recent advances on the regulation of anthocyanin synthesis in reproductive organs [J]. Plant Science, 2011, 181(3): 219−229. [24] RIECHMANN J L, HEARD J, MARTIN G, et al. Arabidopsis transcription factors: genome-wide comparative analysis among eukaryotes [J]. Science, 2000, 290(5499): 2105−2110. [25] ROSINSKI J A, ATCHLEY W R. Molecular evolution of the myb family of transcription factors: evidence for polyphyletic origin [J]. Journal of Molecular Evolution, 1998, 46(1): 74−83. [26] CAO Yunlin, MEI Yuyang, ZHANG Ruining, et al. Transcriptional regulation of flavonol biosynthesis in plants[J/OL]. Horticulture Research, 2024, 11(4): uhae043[2025-08-01]. DOI: 10.1093/hr/uhae043. [27] YANG W T, BAE K D, LEE S W, et al. The MYB-CC Transcription Factor PHOSPHATE STARVATION RESPONSE-LIKE 7 (PHL7) functions in phosphate homeostasis and affects salt stress tolerance in rice[J/OL]. Plants, 2024, 13(5): 637[2025-08-01]. DOI: 10.3390/plants13050637. [28] de LANGE T. SHELTERIN: the protein complex that shapes and safeguards human telomeres [J]. Genes & Development, 2005, 19(18): 2100−2110. [29] WU Yun, WEN Jing, XIA Yiping, et al. Evolution and functional diversification of R2R3-MYB transcription factors in plants[J/OL]. Horticulture Research, 2022, 9: uhac058[2025-08-01]. DOI: 10.1093/hr/uhac058. [30] FENG Guanqiao, BURLEIGH J G, BRAUN E L, et al. Evolution of the 3R-MYB gene family in plants [J]. Genome Biology and Evolution, 2017, 9(4): 1013−1029. [31] DUBOS C, STRACKE R, GROTEWOLD E, et al. MYB transcription factors in Arabidopsis [J]. Trends in Plant Science, 2010, 15(10): 573−581. [32] NAIK J, RAJPUT R, SINGH S, et al. Heat-responsive MaHSF11 transcriptional activator positively regulates flavonol biosynthesis and flavonoid B-ring hydroxylation in banana (Musa acuminata)[J/OL]. The Plant Journal, 2025, 121(5): e70084[2025-08-01]. DOI: 10.1111/tpj.70084. [33] SAXENA S, PAL L, NAIK J, et al. The R2R3-MYB-SG7 transcription factor CaMYB39 orchestrates surface phenylpropanoid metabolism and pathogen resistance in chickpea [J]. New Phytologist, 2023, 238(2): 798−816. [34] ZHONG Chunmei, TANG Yi, PANG Bin, et al. The R2R3-MYB transcription factor GhMYB1a regulates flavonol and anthocyanin accumulation in Gerbera hybrida[J/OL]. Horticulture Research, 2020, 7: 78[2025-08-01]. DOI: 10.1038/s41438-020-0296-2. [35] LI Yaohua, RAN Lingfang, MO Tong, et al. Yellow Petal locus GaYP promotes flavonol biosynthesis and yellow coloration in petals of Asiatic cotton (Gossypium arboreum)[J/OL]. Theoretical and Applied Genetics, 2023, 136(5): 98[2025-08-01]. DOI: 10.1007/s00122-023-04329-7. [36] ZHANG Yue, DUAN Jingjing, WANG Qiaoyun, et al. The Paeonia qiui R2R3-MYB transcription factor PqMYBF1 positively regulates flavonol accumulation[J/OL]. Plants, 2023, 12(7): 1427[2025-08-01]. DOI: 10.3390/plants12071427. [37] YANG Jingwen, WU Xi, AUCAPIÑA C B, et al. NtMYB12 requires for competition between flavonol and (pro)anthocyanin biosynthesis in Narcissus tazetta tepals[J/OL]. Molecular Horticulture, 2023, 3(1): 2[2025-08-01]. DOI: 10.1186/s43897-023-00050-7. [38] 吴立璇, 周平, 范雨昕, 等. 中国水仙NtMYB8的分离及功能研究[J]. 园艺学报, 2021, 48(10): 1895−1906. WU Lixuan, ZHOU Ping, FAN Yuxin, et al. Cloning and functional analysis of NtMYB8 in Narcissus tazetta var. chinensis [J]. Acta Horticulturae Sinica, 2021, 48(10): 1895−1906. [39] BALLESTER A R, MOLTHOFF J, de VOS R, et al. Biochemical and molecular analysis of pink tomatoes: deregulated expression of the gene encoding transcription factors SlMYB12 leads to pink tomato fruit color [J]. Plant Physiology, 2010, 152(1): 71−84. [40] YUE Maolan, JIANG Leiyu, ZHANG Nating, et al. Regulation of flavonoids in strawberry fruits by FaMYB5/FaMYB10 dominated MYB-bHLH-WD40 ternary complexes[J/OL]. Frontiers in Plant Science, 2023, 14: 1145670[2025-08-01]. DOI: 10.3389/fpls.2023.1145670. [41] SONG Zhaopeng, LUO Yong, WANG Weifeng, et al. NtMYB12 positively regulates flavonol biosynthesis and enhances tolerance to low Pi stress in Nicotiana tabacum[J/OL]. Frontiers in Plant Science, 2019, 10: 1683[2025-08-01]. DOI: 10.3389/fpls.2019.01683. [42] SONG Zhongbang, ZHAO Lu, MA Wenna, et al. Ethylene inhibits ABA-induced stomatal closure via regulating NtMYB184-mediated flavonol biosynthesis in tobacco [J]. Journal of Experimental Botany, 2023, 74(21): 6735−6748. [43] CZEMMEL S, STRACKE R, WEISSHAAR B, et al. The grapevine R2R3-MYB transcription factor VvMYBF1 regulates flavonol synthesis in developing grape berries [J]. Plant Physiology, 2009, 151(3): 1513−1530. [44] CZEMMEL S, HÖLL J, LOYOLA R, et al. Transcriptome-wide identification of novel UV-B- and light modulated flavonol pathway genes controlled by VviMYBF1[J/OL]. Frontiers in Plant Science, 2017, 8: 1084[2025-08-01]. DOI: 10.3389/fpls.2017.01084. [45] YANG Feng, WANG Tao, GUO Qiaosheng, et al. The CmMYB3 transcription factors isolated from the Chrysanthemum morifolium regulate flavonol biosynthesis in Arabidopsis thaliana [J]. Plant Cell Reports, 2023, 42(4): 791−803. [46] ZHAO Xuecheng, LI Ping, ZUO Hao, et al. CsMYBL2 homologs modulate the light and temperature stress-regulated anthocyanin and catechins biosynthesis in tea plants (Camellia sinensis) [J]. The Plant Journal, 2023, 115(4): 1051−1070. [47] CAO Yunlin, XIE Linfeng, MA Yingyue, et al. PpMYB15 and PpMYBF1 transcription factors are involved in regulating flavonol biosynthesis in peach fruit [J]. Journal of Agricultural and Food Chemistry, 2019, 67(2): 644−652. [48] ZHAI Rui, WANG Zhimin, ZHANG Shiwei, et al. Two MYB transcription factors regulate flavonoid biosynthesis in pear fruit (Pyrus bretschneideri Rehd. ) [J]. Journal of Experimental Botany, 2016, 67(5): 1275−1284. [49] LI Hua, LI Yu, YU Jiaxuan, et al. MdMYB8 is associated with flavonol biosynthesis via the activation of the MdFLS promoter in the fruits of Malus crabapple[J/OL]. Horticulture Research, 2020, 7: 19[2025-08-01]. DOI: 10.1038/s41438-020-0238-z. [50] ZHANG Xueying, HE Yuqing, LI Linying, et al. Involvement of the R2R3-MYB transcription factor MYB21 and its homologs in regulating flavonol accumulation in Arabidopsis stamen [J]. Journal of Experimental Botany, 2021, 72(12): 4319−4332. [51] HUANG Wenjun, LÜ Haiyan, WANG Ying. Functional characterization of a novel R2R3-MYB transcription factor modulating the flavonoid biosynthetic pathway from Epimedium sagittatum[J/OL]. Frontiers in Plant Science, 2017, 8: 1274[2025-08-01]. DOI: 10.3389/fpls.2017.01274. [52] 王子玥, 杨锋, 王红燕, 等. 三叶青ThMYBPAR基因的克隆及组织差异表达分析[J]. 浙江农林大学学报, 2025, 42(2): 273−280. WANG Ziyue, YANG Feng, WANG Hongyan, et al. Cloning and analysis of tissue-specific expression patterns of ThMYBPAR gene in Tetrastigma hemsleyanum [J]. Journal of Zhejiang A&F University, 2025, 42(2): 273−280. [53] LU Jinkai, TONG Peixi, XU Yuan, et al. SA-responsive transcription factor GbMYB36 promotes flavonol accumulation in Ginkgo biloba[J/OL]. Forestry Research, 2023, 3: 19[2025-08-01]. DOI: 10.48130/FR-2023-0019. [54] CHEN Xiaoshan, WU Ying, YU Zhanghong, et al. BcMYB111 responds to BcCBF2 and induces flavonol biosynthesis to enhance tolerance under cold stress in non-heading Chinese cabbage[J]. International Journal of Molecular Sciences, 2023, 24(10): 8670[2025-08-01]. DOI: 10.3390/ijms24108670. [55] CHEN Cheng, ZHANG Kaixuan, KHURSHID M, et al. MYB transcription repressors regulate plant secondary metabolism [J]. Critical Reviews in Plant Sciences, 2019, 38(3): 159−170. [56] STRACKE R, ISHIHARA H, HUEP G, et al. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling [J]. The Plant Journal, 2007, 50(4): 660−677. [57] WANG Feibing, KONG Weili, WONG G, et al. AtMYB12 regulates flavonoids accumulation and abiotic stress tolerance in transgenic Arabidopsis thaliana [J]. Molecular Genetics and Genomics, 2016, 291(4): 1545−1559. [58] PARK N I, LI Xiaohua, THWE A A, et al. Enhancement of rutin in Fagopyrum esculentum hairy root cultures by the Arabidopsis transcription factor AtMYB12 [J]. Biotechnology Letters, 2012, 34(3): 577−583. [59] DELUC L, BARRIEU F, MARCHIVE C, et al. Characterization of a grapevine R2R3-MYB transcription factor that regulates the phenylpropanoid pathway [J]. Plant Physiology, 2006, 140(2): 499−511. [60] BLANCO E, SABETTA W, DANZI D, et al. Isolation and characterization of the flavonol regulator CcMYB12 from the globe artichoke [Cynara cardunculus var. scolymus (L. ) Fiori][J/OL]. Frontiers in Plant Science, 2018, 9: 941[2025-08-01]. DOI: 10.3389/fpls.2018.00941. [61] SHI Yufeng, LU Taoran, LAI Sanyan, et al. Rosa rugosa R2R3-MYB transcription factors RrMYB12 and RrMYB111 regulate the accumulation of flavonols and anthocyanins[J/OL]. Frontiers in Plant Science, 2024, 15: 1477278[2025-08-01]. DOI: 10.3389/fpls.2024.1477278. [62] XU Zibo, LI Jingwen, SONG Xue, et al. Overexpression of the R2R3-MYB transcription factor GmMYB3a enhances isoflavone accumulation in soybean[J/OL]. Physiologia Plantarum, 2025, 177(1): e70120[2025-08-01]. DOI: 10.1111/ppl.70120. [63] LIU Yitian, ZHANG Shengrui, LI Jing, et al. An R2R3-type MYB transcription factor, GmMYB77, negatively regulates isoflavone accumulation in soybean [Glycine max (L. ) Merr.] [J]. Plant Biotechnology Journal, 2025, 23(3): 824−838. [64] YAN Junhui, WANG Biao, ZHONG Yunpeng, et al. The soybean R2R3 MYB transcription factor GmMYB100 negatively regulates plant flavonoid biosynthesis [J]. Plant Molecular Biology, 2015, 89(1): 35−48. [65] RAMSAY N A, GLOVER B J. MYB-Bhlh-WD40 protein complex and the evolution of cellular diversity [J]. Trends in Plant Science, 2005, 10(2): 63−70. [66] BOVY A, de VOS R, KEMPER M, et al. High-flavonol tomatoes resulting from the heterologous expression of the maize transcription factor genes LC and C1 [J]. The Plant Cell, 2002, 14(10): 2509−2526. [67] DENG Heng, WU Mengbo, WU Yi, et al. SlMYC2-SlMYB12 module orchestrates a hierarchical transcriptional cascade that regulates fruit flavonoid metabolism in tomato [J]. Plant Biotechnology Journal, 2025, 23(2): 477−479. [68] HUANG Y F, VIALET S, GUIRAUD J L, et al. A negative MYB regulator of proanthocyanidin accumulation, identified through expression quantitative locus mapping in the grape berry [J]. New Phytologist, 2014, 201(3): 795−809. [69] WANG Zhong, LUO Zhaopeng, LIU Yongjun, et al. Molecular cloning and functional characterization of NtWRKY11b in promoting the biosynthesis of flavonols in Nicotiana tabacum[J/OL]. Plant Science, 2021, 304: 110799[2025-08-01]. DOI: 10.1016/j.plantsci.2020.110799. [70] LIU Sian, ZHANG Hanyue, MENG Zhaolong, et al. The LncNAT11-MYB11-F3′H/FLS module mediates flavonol biosynthesis to regulate salt stress tolerance in Ginkgo biloba [J]. Journal of Experimental Botany, 2025, 76(4): 1179−1201. [71] ZHAO Xuecheng, ZENG Xiangsheng, LIN Ning, et al. CsbZIP1-CsMYB12 mediates the production of bitter-tasting flavonols in tea plants (Camellia sinensis) through a coordinated activator-repressor network[J/OL]. Horticulture Research, 2021, 8: 110[2025-08-01]. DOI: 10.1038/s41438-021-00545-8. [72] 刘佳欣, 刘慧子, 石晶静, 等. 白桦MYB基因响应激素及盐旱处理的表达研究[J]. 植物研究, 2020, 40(5): 743−750. LIU Jiaxin, LIU Huizi, SHI Jingjing, et al. Expression of MYB genes of birch in response to hormones, salt and drought [J]. Bulletin of Botanical Research, 2020, 40(5): 743−750. [73] LIU Moyang, SUN Wenjun, MA Zhaotang, et al. Integrated network analyses identify MYB4R1 neofunctio-nalization in the UV-B adaptation of Tartary buckwheat[J/OL]. Plant Communications, 2022, 3(6): 100414[2025-08-01]. DOI: 10.1016/j.xplc.2022.100414. [74] BHATIA C, PANDEY A, GADDAM S R, et al. Low temperature-enhanced flavonol synthesis requires light-associated regulatory components in Arabidopsis thaliana [J]. Plant and Cell Physiology, 2018, 59(10): 2099−2112. [75] 孙亚辉, 姜天华, 高康, 等. 野菊黄酮醇合成途径调控因子CiMYB3基因启动子的克隆与表达分析[J]. 现代园艺, 2024(11): 64−66, 74, 162. SUN Yahui, JIANG Tianhua, GAO Kang, et al. Cloning and expression analysis of CiMYB3 gene promoter, a regulator of flavonol synthesis pathway in Chrysanthemum indicum L. [J]. Contemporary Horticulture, 2024(11): 64−66, 74, 162. [76] QI Lanting, SUN Huigai, TIAN Chang, et al. The AmMYB35-AmFLS module mediates the accumulation of flavonol induced by drought stress in Astragalus membranaceus[J/OL]. Food Bioscience, 2025, 68: 106541[2025-08-01]. DOI: 10.1016/j.fbio.2025.106541. [77] LI Baozhu, FAN Ruonan, GUO Siyi, et al. The Arabidopsis MYB transcription factor, MYB111 modulates salt responses by regulating flavonoid biosynthesis[J/OL]. Environmental and Experimental Botany, 2019, 166: 103807[2025-08-01]. DOI: 10.1016/j.envexpbot.2019.103807. [78] ZHANG Lishuang, SUN Yugang, JI Jinqiang, et al. Flavonol synthase gene MsFLS13 regulates saline-alkali stress tolerance in alfalfa [J]. The Crop Journal, 2023, 11(4): 1218−1229. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20250472






 下载:
下载: