-
雄性不育在开花植物中普遍存在,主要表现为雄蕊发育不正常,不能产生具有正常功能的花粉[1]。‘无子瓯柑’Citrus suavissima ‘Seedless’是瓯柑Citrus suavissima的芽变品种,保留了瓯柑肉质饱满、香味特异、耐储藏的优良品质,因果实无核被人们青睐。研究认为,雄性不育是‘无子瓯柑’无核的重要原因之一[2]。张迟等[3]发现‘无子瓯柑’花粉败育始于小孢子母细胞时期,推测其雄性不育与能量代谢异常和营养物质缺乏有关。对矮牵牛Petunia hybrid[4],枸杞Lycium barbarum[5],辣椒Capscum annuum[6]和萝卜Raphanus sativus[7]等植物的研究发现,查尔酮合成酶基因(CHS)的异常表达会引起雄性不育;CHS是黄酮类化合物代谢通路中的关键基因,通过影响查尔酮的产生从而影响黄酮类化合物的生物合成[4],而黄酮类化合物的缺乏/过量是植物雄性不育的重要原因之一。有研究将克隆自10个柑橘种质的CHS基因与温州蜜柑‘国庆4号’Citrus unshiu ‘Guoqing 4’,柚‘冯威’Citrus maxima ‘Fengwei’和甜橙‘红宝石’Citrus sinensis ‘Ruby’的CHS比对,发现不同品种柑橘的CHS基因编码区核苷酸序列相似度极高,达98%以上[8]。本研究以前期工作为基础,以克里曼丁橘Citrus clementina数据库为参照,对‘无子瓯柑’和瓯柑小孢子母细胞时期的花药进行转录组和蛋白质组测序,筛选CHS同源差异表达基因进行克隆和表达量分析,并对CHS基因家族进行生物信息学分析,以期为‘无子瓯柑’雄性不育机理的深入研究打下基础。
HTML
-
‘无子瓯柑’和瓯柑小孢子母细胞时期的花药转录组测序数据源自课题组上传的NCBI数据(NCBI登录号PRJNA430695)。
-
‘无子瓯柑’和瓯柑采自浙江省丽水市富岭街道南寨自然村,采样时间参照前期研究[9]。采集‘无子瓯柑’和瓯柑成熟时期的花蕾进行基因克隆。采集两者小孢子母细胞时期(Ⅰ,花蕾直径2.0~2.4 mm)、减数分裂时期(Ⅱ,花蕾直径2.4~2.8 mm)和四分体时期(Ⅲ,花蕾直径2.8~3.1 mm)的花药,用于分析不同发育时期的基因表达量。采集两者成熟花粉粒时期花蕾(花蕾直径6.5~6.9 mm)的4个不同部位(花药、花丝、雌蕊和花瓣),用于分析不同部位的基因表达量。所有材料均保存于-80 ℃。
-
利用RNA提取试剂盒(TaKaRa MiniBEST Plant RNA Extraction Kit,TaKaRa)提取瓯柑、‘无子瓯柑’不同发育时期的花药和成熟花粉粒时期花蕾不同部位的RNA,并对RNA样品质量进行检测。按反转录试剂盒(PrimeScript RT reagent Kit with gDNA Eraser,TaKaRa)说明书,将得到的RNA反转录成cDNA,并于-20 ℃保存。
以克里曼丁橘基因组数据库为参考,对小孢子母细胞时期‘无子瓯柑’和瓯柑的花药进行转录组和蛋白质组分析,鉴定得到CHS基因家族成员;以差异表达倍数(FC)>1.2,错误发现率(FDR)<0.01为标准[10],筛选到CHS同源差异表达基因;以差异表达倍数(FC)>1.2,假设概率(P)<0.05为标准,筛选CHS同源差异表达蛋白。
对得到的差异表达CHS基因进行克隆及定量引物设计。对‘无子瓯柑’和瓯柑的CHS基因的蛋白质编码区(CDS, Coding Sequence)进行克隆。聚合酶链式反应(PCR, Polymerase Chain Reaction)程序为95 ℃,5 min;95 ℃,30 s,56 ℃,40 s,38个循环;72 ℃,10 min。PCR产物在质量分数为1.5%的琼脂糖凝胶上电泳。利用胶回收试剂盒(TaKaRa)纯化胶回收产物并连接到pMD-18质粒(TaKaRa),转化感受态大肠埃希菌Escherichia coli DH5α(TaKaRa)中,37 ℃震荡培养后涂板挑菌。通过菌液PCR及电泳检测后,选出条带大小正确的菌液送生工生物工程(上海)有限公司测序,获得基因的核苷酸序列。
以克里曼丁橘序列为模板,在线设计qRT-PCR特异性引物(https://www.genscript.com),以甜橙Citrus sinensis的Actin基因(GU911361)[9]为内参基因,引物序列由生工生物工程(上海)有限公司合成。利用CFX96 real-time system实时荧光定量PCR仪(Bio-Rad)及荧光染料SYBR® Premix Ex TaqTMⅡ(TaKaRa)说明书进行实时定量PCR,反应程序为95 ℃,30 s,95 ℃,5 s,57 ℃,30 s,39个循环,65~95 ℃升温检测扩增产物的溶解曲线[9]。设置3个重复,根据2-ΔΔCt方法,以瓯柑小孢子母细胞时期花药的表达量为基准计算基因相对表达量[11],比较‘无子瓯柑’和瓯柑在花药不同发育时期和花粉粒成熟期花蕾不同部位的基因表达量。
-
柑橘CHS基因编码区核苷酸序列相似度极高[8],因此以克里曼丁橘为例开展CHS基因及蛋白质的生物信息学分析。登录克里曼丁橘数据库,下载CHS基因全长序列、基因CDS序列、家族蛋白序列等信息。所有蛋白质序列通过在线数据库ExPASY(https://web.expasy.org/protparam)进行蛋白生理生化分析。基于已有报道[12],利用CELLOv2.5进行亚细胞定位。通过在线数据库Prabi(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行蛋白二级结构分析。利用MEGA6.0软件采用邻接法(Neighbor-Joining)构建克里曼丁橘()、甜橙(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Csinensis)、拟南芥Arabidopsis thaliana(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Athaliana)和水稻Oryza satava()的CHS同源蛋白系统进化树,设置Bootstrap值为1 000,去除支持率低于50%的节点,并显示各分支长度。
1.1. 实验材料
1.1.1. 数据源
1.1.2. 材料
1.2. 基因克隆和基因表达量分析
1.3. CHS基因家族生物信息学分析
-
对小孢子母细胞时期的‘无子瓯柑’和瓯柑花药转录组和蛋白质组数据分析,共得到10个CHS同源基因(表 1);转录组测序结果满足差异表达倍数>1.2且错误发现率<0.01的有3个基因(Ciclev10005133m,Ciclev10001405m,Ciclev10030093m),蛋白质组测序结果满足差异表达倍数>1.2且假设概率<0.05标准的有1个基因(Ciclev10015535m)。得到的4个差异表达CHS基因引物序列如表 2。qRT-PCR特异性引物序列如表 3。
基因名称 差异表达倍数 错误发现率/假设概率 Ciclev10015535m 0.76 0.004 6b Ciclev10005133m 0.65 0.007 5a Ciclev10001405m 1.32 0.006 3a Ciclev10001413m 5.50 0.595 4a Ciclev10001395m 0.94 0.717 6a Ciclev10028604m 5.50 0.161 3a Ciclev10030093m 0.71 0.007 9a Ciclev10028605m 0.91 0.144 1a Ciclev10030398m 5.50 0.399 2a Ciclev10001905m 0.90 0.064 2a 说明: a表示错误发现率; b表示假设概率 Table 1. CHS gene family and expression in C. suavissima 'Seedless' at microsporcyte
基因名称 上游引物(5′→3′) 下游引物(5′→3′) Ciclev10005133m ATGGTGACCGTCGATGAAG CAGTGTTGCCGCTGCTTAA Ciclev10001405m ATGGAGAAAGTTAAAGATG TCCCTACTGTTACAACCTAG Ciclev10030093m ATGACGACAGTGAAAAGTAA ACAAATTTCACCAACTACTGA Ciclev10015535m ATGGCAACCGTTCAAGAGAT GTGTCCCCATCAAAGCTTGA Table 2. Sequences of primer for gene cloning
基因ID 上游引物(5′→3′) 下游引物(5′→3′) GU911361 ATCTGCTGGAAGGTGCTGAG CCAAGCAGCATGAAGATCAA Ciclev10015535m AAGAGCGAGCATATGACGGA CAGCTTCTTTCCCGAGCTTC Ciclev10001405m AATTGTGTCGGGTGCACAAA ATTCCGAACGGACTAAACGC Ciclev10005133m AGCCGAGAACAACAAAGG ATGGGCTTCTCGATCTCAGG Ciclev10030093m CAAGGACCAACAGCAACGAT CTTTCAGCTCGGTCTTGTGG Table 3. Sequences of primer for real-time PCR
对克隆得到的‘无子瓯柑’和瓯柑的CHS同源基因序列(CsCHS)进行比对,结果发现:Ciclev10015535m,Ciclev10005133m和Ciclev10030093m在瓯柑和‘无子瓯柑’间存在核苷酸序列变异,但仅Ciclev10030093m在编码氨基酸水平发生了改变(表 4)。
基因名称 瓯柑 ‘无子瓯柑’ 开放阅读框长度/bp 同源基因在瓯柑与‘无子瓯柑’间的相似度/% 差异碱基位/bp 氨基酸的变化 命名 GenBank
登录号命名 GenBank
登录号Ciclev10015535m CsCHS1 MK070533 CsCHS5 MK070537 1 176 99 501 无 Ciclev10001405m CsCHS6 MK070536 CsCHS6 MK070536 1 188 100 无 无 Ciclev10005133m CsCHS3 MK070534 CsCHS7 MK070538 1 173 98 549~954 922 bp处苏氨酸突变为丙氨酸 Ciclev10030093m CsCHS4 MK070535 CsCHS8 MK070539 1 179 98 342,495 无 Table 4. Alignments of CsCHS nucleotide sequences between Citrus suavissima 'Seedless' and C. suavissima
-
图 1表明:小孢子母细胞时期,‘无子瓯柑’与瓯柑相比Ciclev10005133m,Ciclev10030093m和Ciclev10015535m显著下调,Ciclev10001405m显著上调;减数分裂时期,Ciclev10005133m,Ciclev10001405m和Ciclev10015535m显著下调;四分体时期,Ciclev10001405m和Ciclev10030093m显著下调,Ciclev10005133m显著上调。

Figure 1. Expression level of anther in different development stages in C. suavissima 'Seedless' and C. suavissima
由图 2可知:成熟花粉粒时期,‘无子瓯柑’和瓯柑的花丝中CHS基因表达量相仿。花药中Ciclev10001405m和Ciclev10005133m表达量显著高于其他花器官;Ciclev10030093m,Ciclev10005133m和Ciclev10015535m的表达在‘无子瓯柑’和瓯柑的花药中存在显著差异,可能会引起该时期‘无子瓯柑’和瓯柑的花药中黄酮含量的差异。总的来说,花药中Ciclev10001405m表达量远高于花瓣、雌蕊和花丝,推测Ciclev10001405m的表达在花药中具有特异性。
-
克里曼丁橘基因组数据库共鉴定到13个CHS基因家族成员。对CHS蛋白质理化性质的分析表明:编码的氨基酸数量最少的是Ciclev10003127m(309个),最多的是Ciclev10001395m(396个);平均等电点为6.12,所有蛋白质均为酸性蛋白。相比之下,‘无子瓯柑’和瓯柑中,Ciclev10005133m(390个)编码的氨基酸数量最少,Ciclev10001405m(395个)编码的氨基酸数量最多;平均等电点为6.27(表 5)。亚细胞定位结果表明:这些CHS蛋白都分布在细胞质中,说明CHS蛋白在细胞中的分布具有特异性。蛋白质二级结构分析表明:α-螺旋比例和数量均最高,β-转角则相对较少;表现为α-螺旋数量>无规则卷曲数量>延伸链数量>β-转角数量。‘无子瓯柑’与瓯柑中筛选的4个CHS蛋白也表现出相似趋势(表 6),推测α-螺旋在蛋白质二级结构中起主导作用。
基因名称 氨基酸数量/个 分子量/Da 等电点 Ciclev10015535m 391 42 592.17 6.47 Ciclev10005133m 390 42 676.30 5.84 Ciclev10015900m 328 35 477.11 6.11 Ciclev10001405m 395 42 918.26 6.95 Ciclev10001413m 394 43 364.92 6.56 Ciclev10001395m 396 43 377.16 6.77 Ciclev10028604m 395 43 383.97 5.48 Ciclev10030093m 392 43 406.82 5.82 Ciclev10028605m 395 43 436.08 6.17 Ciclev10030398m 390 429 51.35 6.10 Ciclev10001905m 312 33 958.16 5.44 Ciclev10001806m 330 36 069.43 6.33 Ciclev10003127m 309 33 988.20 5.59 Table 5. Basic information of CHS genes and proteins
蛋白名称 α螺旋 延伸链 β转角 无规则卷曲 数量/个 比例/% 数量/个 比例/% 数量/个 比例/% 数量/个 比例/% Ciclev10015535m 161 41.18 65 16.62 32 8.18 133 34.02 Ciclev10005133m 170 43.59 60 15.38 31 7.95 129 33.08 Ciclev10015900m 142 43.29 59 17.99 29 8.84 98 29.88 Ciclev10001405m 160 40.51 63 15.95 28 7.09 144 36.46 Ciclev10001413m 170 43.15 63 15.99 27 6.85 134 34.01 Ciclev10001395m 175 44.19 62 15.66 26 6.57 133 33.59 Ciclev10028604m 169 42.78 65 16.46 25 6.33 136 34.43 Ciclev10030093m 156 39.80 64 16.33 31 7.91 141 35.97 Ciclev10028605m 161 40.76 66 16.71 32 8.10 136 34.43 Ciclev10030398m 165 42.31 61 15.64 28 7.18 136 34.87 Ciclev10001905m 131 41.99 56 17.95 26 8.33 99 31.73 Ciclev10001806m 133 40.30 53 16.06 22 6.67 122 36.97 Ciclev10003127m 119 38.51 57 18.45 25 8.09 108 34.95 Table 6. Secondary structure analysis of CHS proteins
-
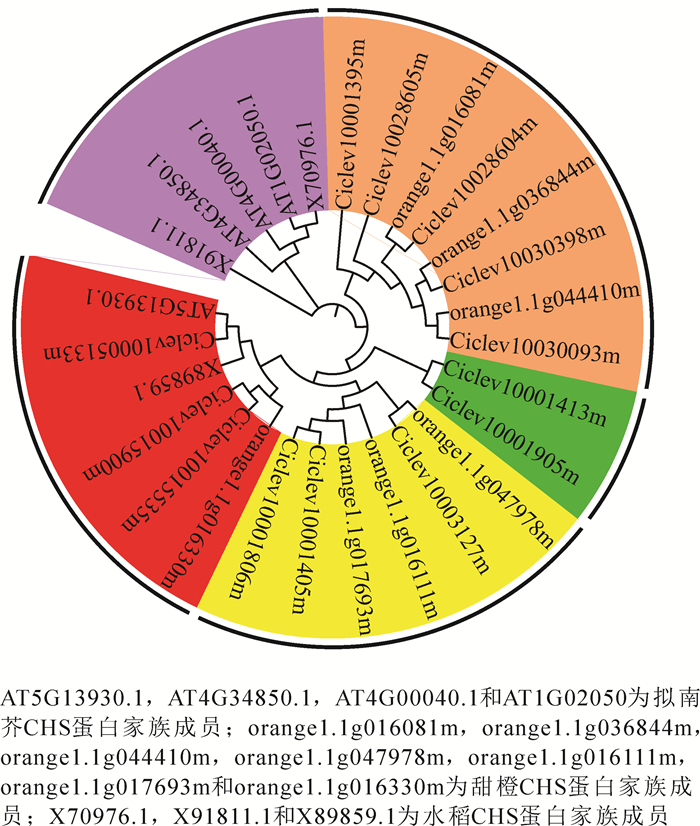
现有研究筛选到克里曼丁橘13个CHS蛋白、甜橙7个CHS蛋白、拟南芥4个CHS蛋白和水稻3个CHS蛋白。按照遗传距离可以将CHS蛋白聚类为5个亚家族(图 3)。Group Ⅰ包含3个拟南芥CHS蛋白和2个水稻CHS蛋白。Group Ⅱ包含5个克里曼丁橘CHS蛋白和3个甜橙CHS蛋白。Group Ⅲ包含2个克里曼丁橘CHS蛋白。Group Ⅳ包含3个克里曼丁橘CHS蛋白和3个甜橙CHS蛋白。Group Ⅴ包含上述4个物种的CHS蛋白成员。其中Ciclev10005133m蛋白和Ciclev10015535m蛋白均聚类于Group Ⅴ,Ciclev10030093m蛋白和Ciclev10001405m蛋白则分别聚类于Group Ⅱ和Group Ⅳ。
2.1. CHS基因家族成员的鉴定和克隆
2.2. CHS基因家族成员的表达分析
2.3. CHS基因家族的生物信息学分析
2.3.1. CHS基因家族成员鉴定
2.3.2. CHS家族同源蛋白系统进化树构建
-
查尔酮合成酶(CHS)催化丙二酰-CoA和香豆酰-CoA结合形成查尔酮,是黄酮类化合物代谢通路(ko00941)中的第1个限速酶;查尔酮是各种黄酮类化合物的基本骨架,其数量异动会影响黄酮类化合物的形成,进而造成花药颜色、形态异常[4-5]。一般认为黄酮类化合物是柑橘具有高抗氧化性和良好药用功效的原因[8];矮牵牛CHS突变后花药颜色由黄色变成白色,花药功能异常,引起雄性不育[4];枸杞CHS的下调表达会影响花药功能,造成枸杞雄性不育[5];水稻中CHS的过量表达会引起花药表面色素大量沉淀,造成水稻雄性不育[13]。
CHS基因家族中成员不多,不同植物中数量也不同。如:拟南芥基因组数据库中仅鉴定到4个同源基因,大豆Glycine max[14]中略多(8个)。对已筛选的CHS同源基因的研究发现[13]:X89859.1基因与水稻绒毡层的发育极其紧密,而后者发育正常与否直接关系着花粉的育性[15]。本研究在CHS蛋白系统进化树分析中发现,柑橘CHS同源蛋白Ciclev10005133m和水稻CHS同源蛋白X89895.1聚类于Group Ⅴ,推测两者具有相似的功能。在‘无子瓯柑’中,Ciclev10005133m同源基因在小孢子母细胞至减数分裂时期的表达显著低于瓯柑,因此,Ciclev10005133m同源基因的异常表达可能对‘无子瓯柑’小孢子母细胞发育过程产生不利影响。此外,Ciclev10015535m和Ciclev10030093m同源基因的表达也在小孢子母细胞时期出现显著下调,因此,CHS同源基因起始于小孢子母细胞时期的下调表达可能成为后期‘无子瓯柑’小孢子败育的重要原因。
本研究以克里曼丁橘数据库为基础,从‘无子瓯柑’和瓯柑的转录组和蛋白质组数据中筛选出4个CHS同源差异表达基因,其核苷酸序列在‘无子瓯柑’和瓯柑中的相似度达到98%,并且3个同源基因在‘无子瓯柑’中表达的显著下调起始于小孢子母细胞时期。CHS的异常表达可能是‘无子瓯柑’小孢子发育异常从而造成雄性不育的原因。












 DownLoad:
DownLoad: