-
朱顶红Hippeastrum rutilum又名红花莲、孤挺花等,系石蒜科Amaryllidaceae朱顶红属Hippeastrum
所有种类的总称,多年生草本植物,性喜温暖、湿润气候,稍耐寒[1]。现有约70个种[2]在全球呈离散分布,野生种主要集中分布在巴西和玻利维亚一带。在中国,朱顶红属外来引入花卉[3]。朱顶红花色艳丽且复杂多变,花瓣形状丰富,极具观赏价值,除可用于一般的盆景植物外,还可用于户外景观植物栽培,是元旦、春节和国庆的重要装饰。目前,国际上流行的园艺杂交种朱顶红有100多个品种,其中在中国栽培的品种有60多个[4]。作为高档观赏花卉,国内外开展的对朱顶红相关研究,主要集中在栽培技术(包括组培)、育种、扩繁技术和病虫害防治等方面。近年来,育种学家们培育出了很多花型、花色变化丰富,花瓣厚和花期长的新品种[3],但在国内推广应用的朱顶红品种主要引自荷兰,缺少自主培育的新品种,而且朱顶红的遗传背景较复杂,无法确定大部分栽培品种的遗传背景和品种间的亲缘关系,采用传统的分类方法很难区分其种下类群和品种[5]以及朱顶红种质资源的遗传多样性分析数据[4],因此,了解朱顶红的遗传多样性对于朱顶红遗传育种具有重要意义。已有研究表明:简单序列重复技术(ISSR)[6−7]已经成功应用于朱顶红种质资源遗传关系分析和品种鉴定,但这并不能完全反映朱顶红遗传多样性。2009年COLLARD等[8]新开发的基于植物基因组起始密码子ATG侧翼序列的保守性,设计单引物对目的基因进行扩增的新型分子标记技术——目标起始密码子多态性分子标记(start codon targeted polymorphism, SCoT)。ISSR引物扩增介于反向重复序列位点间的序列[9],部分序列存在无法扩增的现象。与ISSR标记相比,SCoT标记单引物与ATG结合后扩增目的基因或其附近DNA序列[10],有更为广阔的DNA扩增范围,其实验结果更具有可靠性。目前,SCoT标记已经成功应用于花生Arachis[11]、茶树Camellia sinensis[12−13]、栀子Gardenia jasminoides[14]、油菜Brassica rapa[15]、石蒜属Lycoris[16]、猕猴桃Actinidia[17]、铁皮石斛Dendrobium officinale[18]等植物遗传多样性、亲缘关系分析和变异鉴定以及基因变异表达等方面。为了更进一步分析更多朱顶红品种间的遗传多样性和亲缘关系,本研究利用建立的SCoT标记体系对朱顶红品种进行遗传多样性和亲缘关系分析,旨在了解朱顶红在分子水平上的遗传变异,为朱顶红杂交育种、种质资源保护和利用提供理论依据和技术基础。 -
植物材料朱顶红采自浙江农林大学遗传学科朱顶红植物种质资源圃,共计41个朱顶红品种(表1)。于2019年4月采集朱顶红花瓣,液氮冷冻并于−80 ℃冰箱保存备用。本研究所用引物是根据COLLARD等[8]设计的SCoT引物。
表 1 41个朱顶红品种性状描述
Table 1. Character description of 41 H. rutilum cultivars used in this stduy
品种编号 品种名 性状描述 花色 品种编号 品种名 性状描述 花色 1 ‘阿弗雷’‘Alfresco’ 重瓣 白NN155-D 22 ‘奇迹’‘A Miracle 单瓣 红46-A 2 ‘爱神’‘Aphrodite’ 重瓣 白NN155-D 23 ‘奇妙仙子’‘Tinker Bell’ 单瓣 红41-D,白NN155-D 3 ‘冰后’‘Ice Queen’ 重瓣 白NN155-D 24 ‘瑞贝卡’‘Rebecca’ 单瓣 红紫73-C,白NN155-D 4 ‘焦点’‘Spotlight’ 单瓣 白NN155-D,红53-B 25 ‘世外桃源’‘Paradise’ 单瓣 红紫73-A,白NN155-D 5 ‘粉色惊奇’‘Pink Surprise’ 单瓣 红54-A 26 ‘欲望’‘Desire’ 单瓣 红41-C 6 ‘鬼魅’‘Joker’ 重瓣 橙红N30-A 27 ‘珍妮小姐’‘Lady Jane’ 单瓣 红47-A,红56-A 7 ‘黑天鹅’‘Royal Velvet’ 重瓣 红53-A 28 ‘婚礼舞曲’‘Wedding Dance’ 单瓣 白NN155-A 8 ‘快车’‘The Express’ 单瓣 橙红N34-A 29 ‘托斯卡’‘Tosca’ 单瓣 红紫58-B 9 ‘哈库’‘Haku’ 单瓣 红54-B 30 ‘双梦’‘Double Dream’ 重瓣 红54-A 10 ‘露天’‘Alfresco’ 重瓣 白NN155-D 31 ‘红狮’‘Red Lion’ 单瓣 红41-A 11 ‘红宝石之星’‘Star of Ruby’ 单瓣 橙红N34-A,黄绿145-D 32 ‘千禧蛋’‘Millennium Egg’ 单瓣 白155-A 12 ‘红唇’‘Tres Chic’ 单瓣 红42-A,黄2-D 33 ‘焰火’‘Fireworks’ 单瓣 橙红34-A 13 ‘红娘’‘Matdhmaker’ 重瓣 红42-A,黄2-D 34 ‘马格’‘Magné’ 单瓣 红47-A 14 ‘侯爵’‘Marquis’ 重瓣 白NN155-D 35 ‘世界和平’‘World Peace’ 单瓣 红50-A 15 ‘花瓶’‘Gervase’ 单瓣 红50-B 36 ‘北极女神’‘Arctic Nymph’ 重瓣 白NN155-B 16 ‘滑稽演员’‘Harlequin’ 重瓣 白NN155-D 37 ‘首映’‘Premiere’ 单瓣 灰红182-A 17 ‘黄金岁月’‘Golden Years’ 重瓣 红46-A 38 ‘圣诞快乐’‘Merry Christmans’ 重瓣 白NN155-C,红45-A 18 ‘甜蜜妮芙’‘Sweet Nymph’ 重瓣 红45-C 39 ‘清晨阳光’‘Morning Light’ 单瓣 白NN155-D,红紫58-B 19 ‘玫瑰花样’‘Rose Petal’ 重瓣 白NN155-D 40 ‘超级黛丝’‘Giantama Deus’ 重瓣 白NN155-D,红54-C 20 ‘绣球’‘Hydrangea’ 单瓣 白NN155-D 41 ‘迷雾’‘Misty’ 单瓣 黄4-C,红46-A 21 ‘迎春’‘Jasminum Nudiflorum’ 重瓣 橙红N34-A,白NN155-C 说明:表中大写字母表示不同的色系代码 -
采用改良 CTAB法[19]提取朱顶红花瓣基因组DNA,采用微量核酸测定仪(Nanodrop 2000)测定DNA的浓度和质量,并通过质量分数为1.0%的琼脂糖凝胶电泳检测,检测合格的DNA样品于−20 ℃冰箱保存备用。
-
为获得朱顶红SCoT-PCR反应的最佳体系,参考姜小凤[20]和潘媛等[14]报道的SCoT扩增反应条件,通过对PCR反应五要素(模板、引物、MgCl2、dNTPs、rTaqDNA聚合酶)进行5因素4水平正交实验(
$L^4_5 $ ,表2~3)。PCR扩增反应体系为20 μL,PCR扩增反应程序为:94 ℃预变性5 min,94 ℃变性35 s,退火35 s,72 ℃复性90 s,35个循环;72 ℃延伸10 min,16 ℃保存。PCR扩增产物进行质量分数为1.0%琼脂糖凝胶电泳。表 2
${{L}}^4_5 $ 正交实验因素及水平Table 2.
$L^4_5 $ factors and levers of orthogonal design水平因素 引物/(μmol·L−1) MgCl2/(mmol·L−1) DNA/ng dNTPs/(mmol·L−1) rTaq/(×16.67 nkat) 1 0.1 1.5 30 0.1 0.50 2 0.2 2.0 40 0.2 0.75 3 0.3 2.5 50 0.3 1.00 4 0.4 3.0 60 0.4 1.25 表 3 朱顶红SCoT-PCR正交试验体系筛选
Table 3. Screening of orthogonal systems
处理
编号模板
DNA/ng引物/
(μmol·L−1)Mg2+/
(mmol·L−1)dNTPs/
(mmol·L−1)rTaq/
(×16.67 nkat)处理
编号模板
DNA/ng引物/
(μmol·L−1)Mg2+/
(mmol·L−1)dNTPs/
(mmol·L−1)rTaq/
(×16.67 nkat)1 30 0.1 1.5 0.1 0.50 9 50 0.4 1.5 0.3 0.75 2 30 0.2 2.0 0.2 0.75 10 50 0.3 2.0 0.1 0.50 3 30 0.3 2.5 0.3 1.00 11 50 0.2 2.5 0.4 1.25 4 30 0.4 3.0 0.4 1.25 12 50 0.1 3.0 0.2 1.00 5 40 0.3 1.5 0.2 1.25 13 60 0.2 1.5 0.4 1.00 6 40 0.4 2.0 0.1 1.00 14 60 0.1 2.0 0.3 1.25 7 40 0.1 2.5 0.4 0.75 15 60 0.4 2.5 0.2 0.50 8 40 0.2 3.0 0.3 0.50 16 60 0.3 3.0 0.1 0.75 -
参照COLLARD等[8]设计SCoT引物,并由南京金斯瑞生物技术有限公司合成。引物筛选先利用1个样品对55条SCoT引物进行初步筛选,选择条带清晰的引物,再利用性状差异较大的3个朱顶红样品进行复筛,选择条带清晰、具多态性条带的引物进行后续41份朱顶红样品的SCoT-PCR扩增。SCoT-PCR扩增反应程序为:94 ℃预变性5 min,94 ℃变性35 s,退火35 s,72 ℃复性90 s,36个循环,72 ℃延伸10 min,16 ℃保存。PCR扩增产物用质量分数为1.0%琼脂糖凝胶电泳检测。
-
由于引物与DNA结合点可由琼脂糖凝胶电泳图谱的条带表示,因此,PCR电泳图中的每个条带就是1个位点。在数据统计时,记录重复的DNA电泳条带峰同一引物的扩增产物,迁移率相同的条带记为“1”,没有条带记为“0”,并建立矩阵,只记录易于辨认的条带,排除模糊不清的条带。利用Excel计算多态性条带百分比(percentage of bands,PPB)。根据“1,0”矩阵并使用NTSYspc软件计算遗传距离和遗传相似系数,依据SHAN程序按照非加权配对算术平均法(UPGMA)法构建朱顶红样品间的聚类图。
-
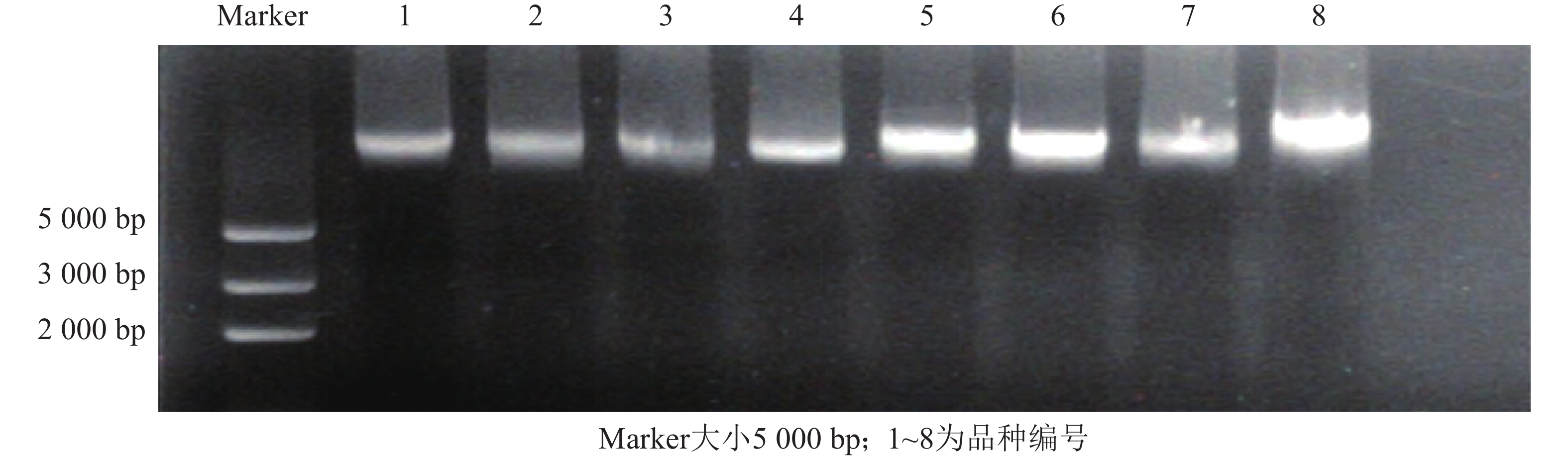
采用改良CTAB法提取朱顶红基因组DNA,经质量分数为1.0%琼脂糖凝胶电泳检测,各泳道朱顶红基因组DNA条带清晰、亮度好(图1),点样孔附近无杂质残留,表明提取DNA完好,降解少;使用微量核酸测定仪检测DNA质量浓度与纯度,吸光度值D(260)/D(280)为1.88~2.00,表明提取的朱顶红基因组DNA纯度较高,杂质较少;经测定朱顶红DNA质量浓度为80.0~896.2 mg·L−1,可满足后续实验要求。
-
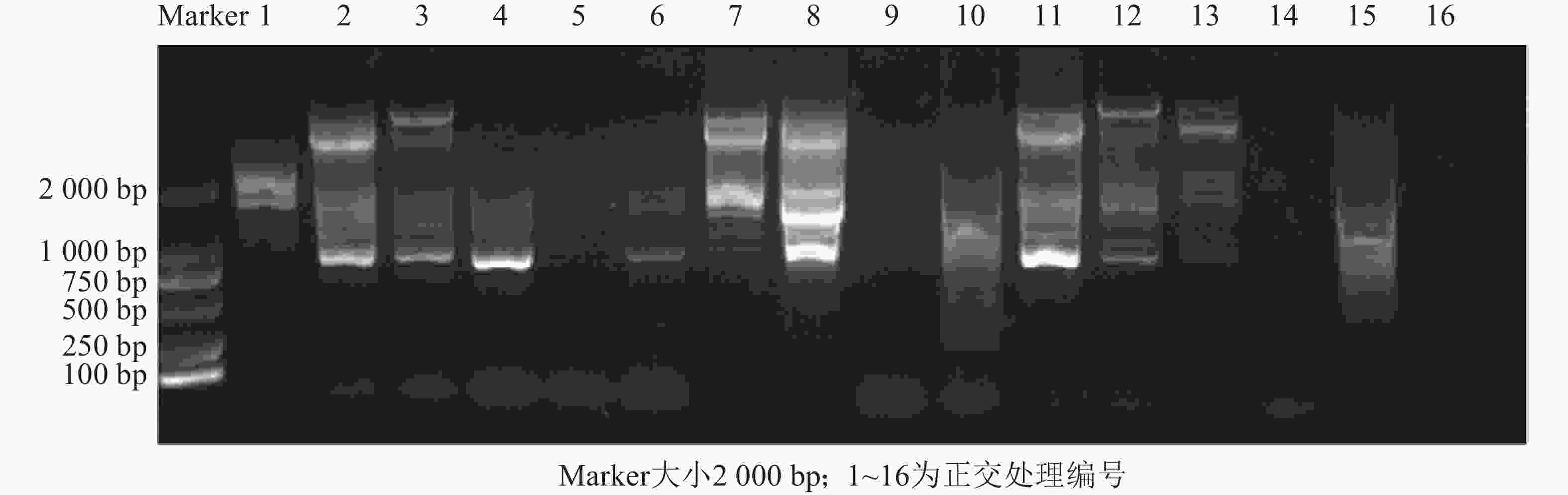
通过5因素4水平(
$L^4_5 $ )正交试验设计(表3),采用SCoT引物P55对朱顶红样品‘黑天鹅’DNA进行PCR扩增,结果发现:5号、9号、10号、14号、15号和16号处理,无扩增条带或条带模糊,可能是由于dNTPs用量过少而导致的。1号处理条带清晰,但条带数量少;12号处理条带清晰、数目多但有非特异性条带发生;7号处理由于条带清晰、数量多且没有非特异性条带的扩增(图2),因此,综合扩增条带与正交体系筛选分析,朱顶红SCoT-PCR扩增条带清晰且多态性良好的最佳反应体系为7号处理(20 μL):DNA 40 ng,引物 0.1 μmol·L−1;MgCl2 2.0 mmol·L−1,dNTPs 0.4 mmol·L−1和rTaq DNA聚合酶0.75 U (1 U =16.67 nkat)。 -
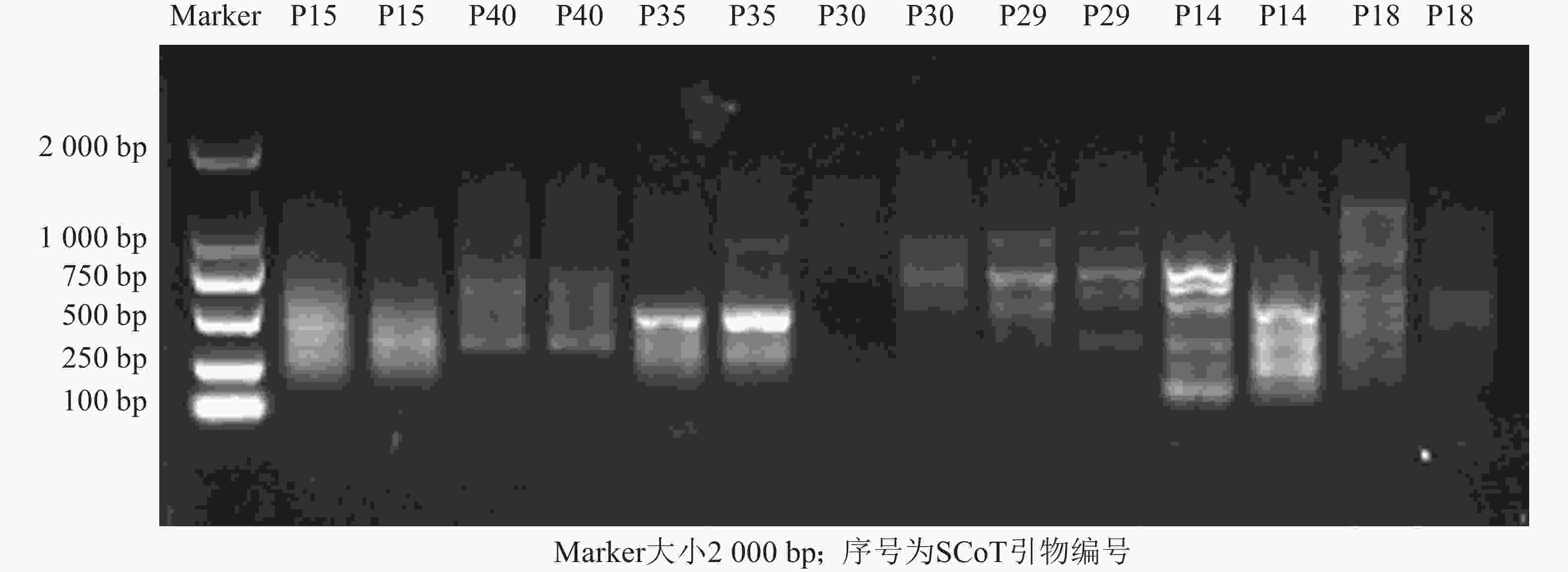
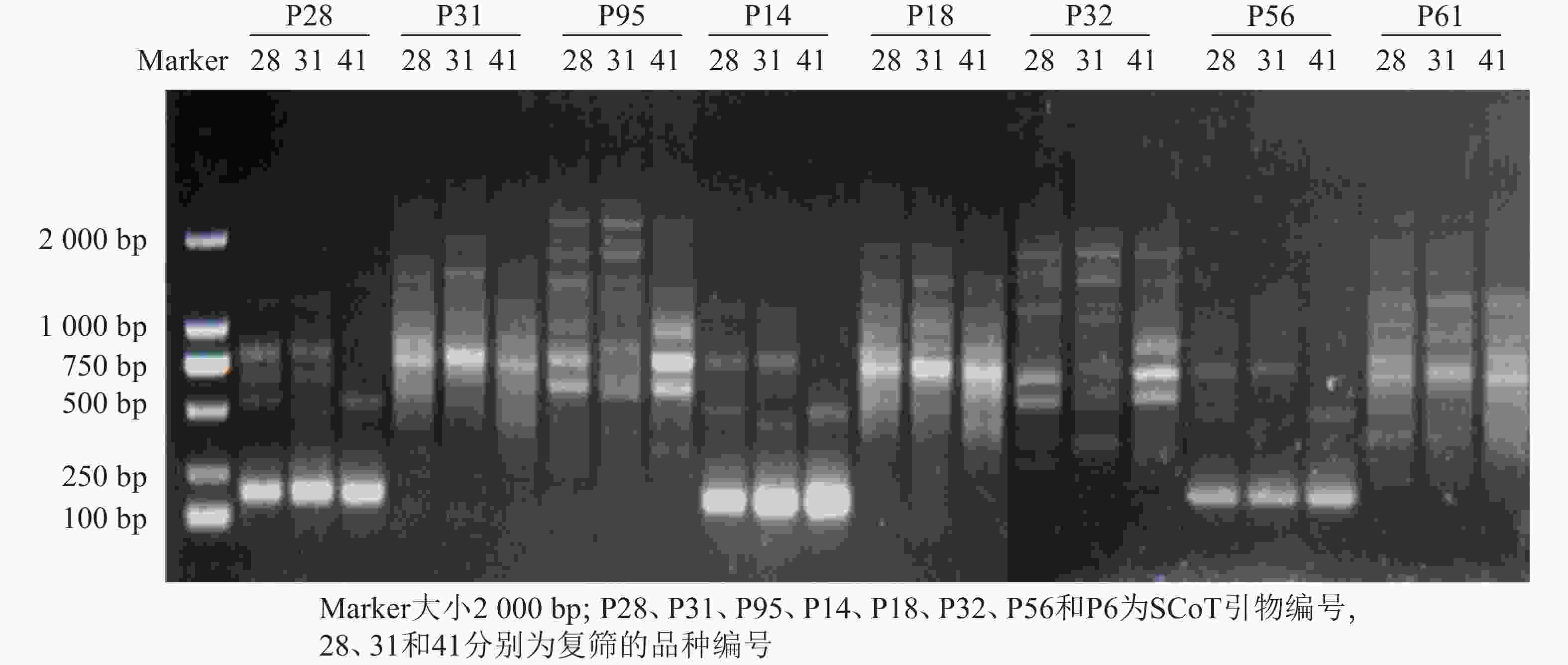
利用建立的SCoT体系从55条SCoT引物初筛出32条条带多且清晰的引物(图3),再用3个花色差异较大的朱顶红样品(‘婚礼舞曲’‘红狮’‘迷雾’)对32条SCoT引物进行复筛(图4),最终筛选到12条条带清晰且多态性良好的SCoT引物,用于后续41份朱顶红样品SCoT- PCR扩增。
-
利用12条引物对41个朱顶红品种的DNA进行PCR扩增,产物分布在 200~3 000 bp,共扩增出89条清晰的条带,每条引物扩增的条带数为5~11条,其中,P56扩增条带数最多,可达11条,多态性条带11条;P5扩增条带数最少为5条,多态性条带4条,平均每条引物扩增条带为7.42条。多态性条带合计77条,平均每条引物可扩增出的多态性位点为6.42条,多态性比率为86.52%。多态性最高的引物是P32、P40和P56均达100%;而引物P12的多态性最低,仅有60%(表4)。以上结果表明:SCoT分子标记适用于朱顶红的多态性位点的检测,便于朱顶红品种间的遗传多样性的分析。
表 4 SCoT标记引物扩增结果
Table 4. Amplification results of SCoT marker primers
引物编号 引物序列 扩增条带数 多态性条带数 多态性比率/% P5 CAACAATGGCTACCACGA 5 4 80.00 P12 ACGACATGGCGACCAACG 5 3 60.00 P13 ACGACATGGCGACCATCG 9 7 77.78 P32 CCATGGCTACCACCGCAC 10 10 100.00 P40 CCATGGCTACCACCGCCG 5 5 100.00 P41 AACCATGGCTACCACCGA 6 5 83.33 P43 AACCATGGCTACCACCGG 7 7 100.00 P54 ACAATGGCTACCACCAGC 5 4 80.00 P55 ACAATGGCTACCACCAGG 8 6 75.00 P56 ACAATGGCTACCACCAGA 11 11 100.00 P57 ACAATGGCTACCACCAGT 9 7 77.78 P61 ACCATGGCTACCACCGAG 9 8 88.89 平均 7.42 6.42 86.52 合计 89 77 86.52 -
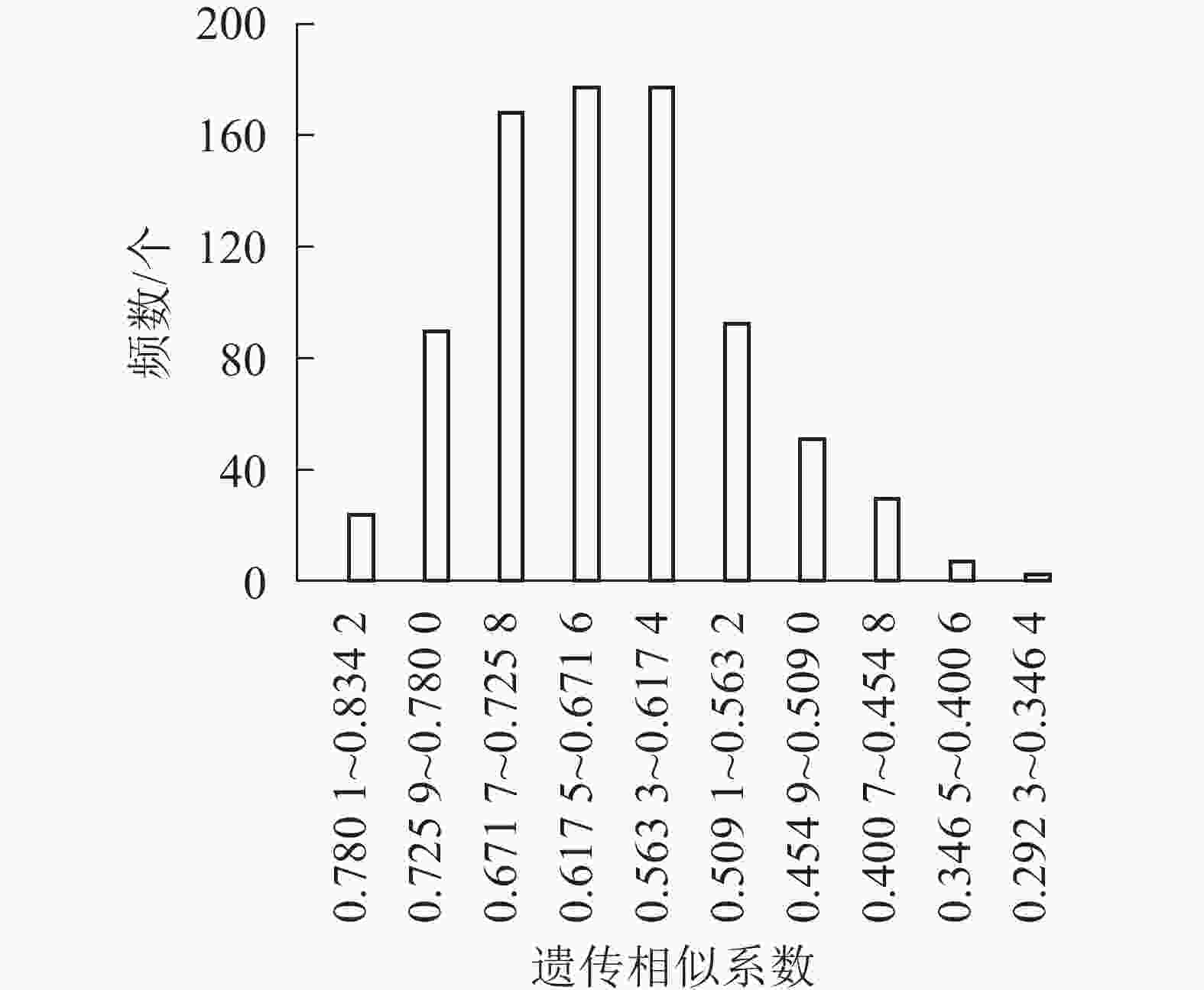
根据12条多态性良好的SCoT引物对41份朱顶红样品的扩增结果,使用NTSYspc软件计算品种遗传相似系数。41份朱顶红品种间共获得861个相似系数组成相似性矩阵,41份朱顶红样材料两两之间的相似系数为0.292 3~0.833 3,平均为0.498 4。其中,2号‘爱神’和3号‘冰后’相似系数最大(0.833 3),表明两者之间亲缘关系较近;23号‘奇妙仙子’和41号‘迷雾’相似系数最小(0.292 3),表明两者之间亲缘关系较远,相差较大。
同时,对41份朱顶红品种的遗传相似系数进行统计分析,以0.054 1为间距,分成10组,分析基因相似系数的频数条形图(图5)。大部分朱顶红品种种间相似度集中在0.400 7~0.563 2,数目为524个,所占比例为63.91%;剩下的基因相似系数在两侧呈不对称分布。表明朱顶红不同品种之间存在明显差异,具有丰富的遗传多样性。对相似性最高的10对品种的瓣型花色进行比对可以发现:相似性高的品种间瓣型具有高度的一致性(100%),而花色的一致性较低(40%),说明朱顶红在花色方面的遗传多样性更为丰富。
-
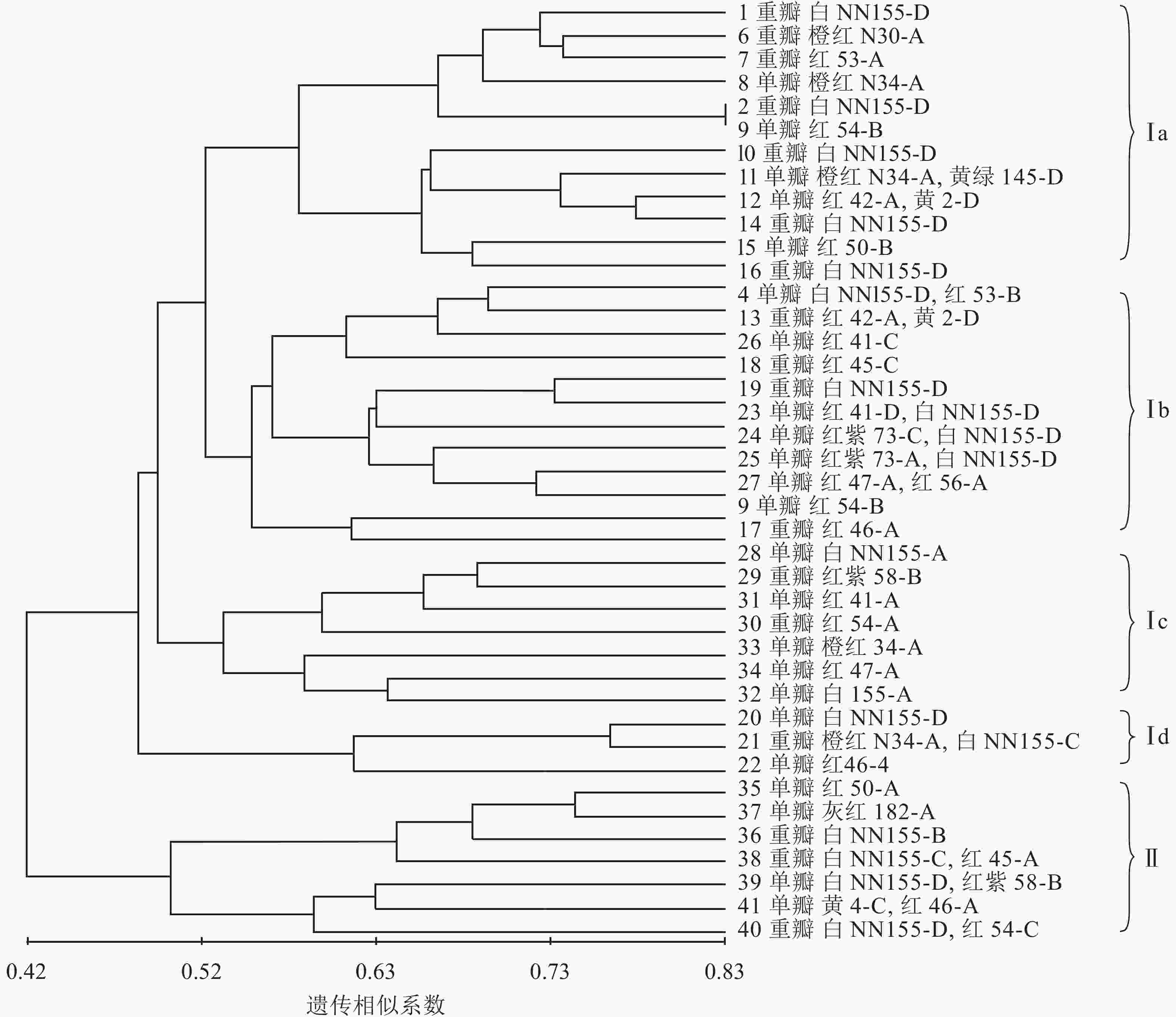
利用NTSYS 2.1软件中的非加权配对算术平均法(UPGMA)对41份朱顶红品种进行聚类分析,构建分子系统树(图6)。由图6可知:在遗传相似系数的0.42处可以将41份朱顶红品种划分为2个大类,说明这2个类群之间有较为明显的差异。第Ⅰ大类有34个品种,既有重瓣品种又有单瓣品种,第Ⅰ大类又分为4个小类,其中Ⅰa类中,白色的2号‘爱神’和3号‘冰后’聚在一起,橙红色6号‘鬼魅’、8号‘快车’和红色的7号‘黑天鹅’聚在一起;Ⅰb小类多为红色系列,而且还有多个由白色和红色形成的复色花,如4号‘焦点’、24号‘瑞贝卡’、25号‘世外桃源’和13号‘红唇’;Ⅰc小类多为红色繁殖类,其中白色的可能是品种变异的结果;Ⅰd小类中单瓣、白色的20号‘绣球’和红色的22号‘奇迹’是重瓣、橙红和白色组成的复色花(21号‘迎春’)的可能亲本;第Ⅱ大类品种较为简单,包括35号‘世界和平’、36号‘北极女神’、37号‘首映’、38号‘圣诞快乐’、39号‘清晨阳光’、40号‘超级黛丝’、41号‘迷雾’在内的7个品种(系),除36号‘北极女神’和37号‘首映’为重瓣外,均为单瓣,且多为复色花。
-
朱顶红是国际市场上常用的观赏植物,其种间杂交育种技术极大地丰富了朱顶红品种(系),具有较高的经济价值。但目前,朱顶红品种改良和选育主要以传统的杂交育种方式为主,从自然杂交、人工杂交的后代中分离优良性状单株,通过2~3 α培育后鉴定花瓣性状、花色等形态学特征,性状不同于亲本且具有稳定遗传性状的被认定为新品种。此类育种方法工作年限长且进展缓慢,无法区分真假杂交种,有极大可能面临育种失败的风险。
近年发展起来的SCoT分子标记技术兼具操作简单、多态性高、遗传信息丰富、成本低和引物通用性强等特点[21],是一种分析物种遗传多样性和亲缘关系有效的分子标记辅助育种技术,为植物种质鉴定和指纹图谱构建等研究提供了新的技术手段;它可以排除外界环境带来的表观遗传变化,从基因组水平真实反映材料的遗传基础与特征,从而快速判断是否为新品种,极大地缩短育种年限,降低育种成本。SCoT标记技术已应用于园林植物育种中,如玫瑰Rosa rugosa杂交种后代早期选择、遗传多样性和亲缘关系分析等方面[21]。本研究应用12条SCoT引物对41份朱顶红品种进行PCR扩增后均检测到清晰的条带,平均多态性条带比率高达86.52%,品种间遗传相似系数为0.292 3~0.834 3,与利用ISSR分子标记检测61份朱顶红品种间(遗传相似系数为0.371 4~0.842 9)[6−7]的结果相比,本实验的朱顶红品种间的遗传范围更广泛,遗传多样性更高,可为朱顶红的新品种选育提供基础。聚类结果表明:本研究的41个朱顶红品种没有完全按照花的瓣型,而是根据遗传相似系数混合聚类,多数花色性状相似品种被聚在一起,如第Ⅱ大类多为红色和白色组成的复色花。这与张林等[6−7]利用ISSR标记对61个朱顶红品种的聚类不太一致,可能是由于SCoT标记技术是一种能跟踪性状,并获得与性状相关目的基因的新型分子标记技术;利用SCoT标记技术还可以初步判断可能的杂交亲本,如Ⅰd小类中单瓣、白色的‘绣球’(20号)和红色的‘奇迹’(22号)是重瓣,橙红和白色组成的复色花(21号‘迎春’)的可能亲本。此假设可以在后期分子水平实验中进一步验证。
本研究成功有效地利用SCoT标记技术研究了朱顶红品种间的遗传多样性和对可能亲本的鉴定。今后可从朱顶红种质资源圃中选择亲缘关系较远的可育品种作为亲本,进行品种间杂交,培育花色丰富和花型多样的朱顶红新品种,进一步改良和选育朱顶红的新品种。
Genetic diversity analysis of Hippeastrum rutilum cultivars based on SCoT markers
-
摘要:
目的 进一步从分子水平分析朱顶红Hippeastrum rutilum品种间的遗传多样性、亲缘关系和亲本鉴定。 方法 利用正交试验设计方法筛选朱顶红目标起始密码子多态性分子标记(SCoT)体系,并以41份朱顶红品种为材料对遗传多样性和亲缘关系进行分析。 结果 ①朱顶红SCoT标记最佳反应体系(20 μL):包括DNA 40 ng,引物 0.1 μmol·L−1;MgCl2 2.0 mmol·L−1,dNTPs 0.4 mmol·L−1和rTaq DNA聚合酶0.75 U (1 U=16.67 nkat)。② 12条SCoT引物从41份朱顶红品种获得77条多态性条带,平均多态性比率高达86.52%。朱顶红品种间遗传相似系数为0.292 3~0.834 3,表明41份朱顶红品种间遗传多样性较高,遗传范围广。③非加权配对算术平均法(UPGMA)聚类表明:朱顶红SCoT标记聚类与花色的相关性较大,但与瓣型相关性不大。在遗传相似系数0.420 0处将41份朱顶红品种分为两大类:第Ⅰ大类既有重瓣品种又有单瓣品种,第Ⅰ大类又分为4个小类,其中相似花色的聚为一类,Ⅰd小类中单瓣、白色的‘绣球’‘Hydrangea’(20号)和红色的‘奇迹’‘Miracle’(22号)可能是重瓣、橙红和白色组成的复色花(21号‘迎春’‘Yingchun’)的亲本;第Ⅱ大类品种多为复色花。 结论 SCoT标记技术可有效地应用于朱顶红品种间遗传多样性分析和可能亲本的鉴定。图6表4参21 Abstract:Objective The objective of this study is to further analyze the genetic diversity, genetic relationship and parental identification of Hippeastrum rutilum cultivars at molecular level. Method The SCoT marker system of H. rutilum was screened by orthogonal design method, and genetic diversity and genetic relationship of 41 cultivars were analyzed. Result (1) The optimum reaction system of SCoT markers for H. rutilum (20 μL) included DNA 40 ng, primer 0.1 μmol·L−1, MgCl2 2.0 mmol·L−1, dNTPs 0.4 mmol·L−1 and rTaq DNA polymerase 0.75 U (1 U=16.67 nkat). (2) 77 polymorphic bands were obtained from 41 H. rutilum cultivars by 12 SCoT primers, and the average polymorphic band ratio was up to 86.52%. The genetic similarity coefficient between H. rutilum cultivars was 0.292 3−0.834 3, indicating that the 41 cultivars had high genetic diversity and wide genetic range. (3)UPGMA(unweighted pair-group method with arithmetic means) cluster analysis showed that the SCoT marker clustering of H. rutilum had significant correlation with flower color, but not with the petal type. At genetic similarity coefficient of 0.420 0, the 41 cultivars were divided into two groups. The first group had both double and single petal cultivars. The first group was divided into four subgroups, among which those with similar flower colors were clustered into one group. In subgroupⅠd, the single-petal, white ‘Hydrangea’ (No. 20) and red ‘Miracle’ (No. 22) were the possible parents of double-petal, orange-red and white multicolor flowers (No. 21 ‘Yingchun’). The second group was mostly multicolor flowers. Conclusion SCoT marker technique can be effectively applied to genetic diversity analysis and identification of possible parents of H. rutilum cultivars. [Ch, 6 fig. 4 tab. 21 ref.] -
Key words:
- plant breeding /
- Hippeastrum rutilum /
- SCoT marker /
- genetic diversity /
- parental identification
-
表 1 41个朱顶红品种性状描述
Table 1. Character description of 41 H. rutilum cultivars used in this stduy
品种编号 品种名 性状描述 花色 品种编号 品种名 性状描述 花色 1 ‘阿弗雷’‘Alfresco’ 重瓣 白NN155-D 22 ‘奇迹’‘A Miracle 单瓣 红46-A 2 ‘爱神’‘Aphrodite’ 重瓣 白NN155-D 23 ‘奇妙仙子’‘Tinker Bell’ 单瓣 红41-D,白NN155-D 3 ‘冰后’‘Ice Queen’ 重瓣 白NN155-D 24 ‘瑞贝卡’‘Rebecca’ 单瓣 红紫73-C,白NN155-D 4 ‘焦点’‘Spotlight’ 单瓣 白NN155-D,红53-B 25 ‘世外桃源’‘Paradise’ 单瓣 红紫73-A,白NN155-D 5 ‘粉色惊奇’‘Pink Surprise’ 单瓣 红54-A 26 ‘欲望’‘Desire’ 单瓣 红41-C 6 ‘鬼魅’‘Joker’ 重瓣 橙红N30-A 27 ‘珍妮小姐’‘Lady Jane’ 单瓣 红47-A,红56-A 7 ‘黑天鹅’‘Royal Velvet’ 重瓣 红53-A 28 ‘婚礼舞曲’‘Wedding Dance’ 单瓣 白NN155-A 8 ‘快车’‘The Express’ 单瓣 橙红N34-A 29 ‘托斯卡’‘Tosca’ 单瓣 红紫58-B 9 ‘哈库’‘Haku’ 单瓣 红54-B 30 ‘双梦’‘Double Dream’ 重瓣 红54-A 10 ‘露天’‘Alfresco’ 重瓣 白NN155-D 31 ‘红狮’‘Red Lion’ 单瓣 红41-A 11 ‘红宝石之星’‘Star of Ruby’ 单瓣 橙红N34-A,黄绿145-D 32 ‘千禧蛋’‘Millennium Egg’ 单瓣 白155-A 12 ‘红唇’‘Tres Chic’ 单瓣 红42-A,黄2-D 33 ‘焰火’‘Fireworks’ 单瓣 橙红34-A 13 ‘红娘’‘Matdhmaker’ 重瓣 红42-A,黄2-D 34 ‘马格’‘Magné’ 单瓣 红47-A 14 ‘侯爵’‘Marquis’ 重瓣 白NN155-D 35 ‘世界和平’‘World Peace’ 单瓣 红50-A 15 ‘花瓶’‘Gervase’ 单瓣 红50-B 36 ‘北极女神’‘Arctic Nymph’ 重瓣 白NN155-B 16 ‘滑稽演员’‘Harlequin’ 重瓣 白NN155-D 37 ‘首映’‘Premiere’ 单瓣 灰红182-A 17 ‘黄金岁月’‘Golden Years’ 重瓣 红46-A 38 ‘圣诞快乐’‘Merry Christmans’ 重瓣 白NN155-C,红45-A 18 ‘甜蜜妮芙’‘Sweet Nymph’ 重瓣 红45-C 39 ‘清晨阳光’‘Morning Light’ 单瓣 白NN155-D,红紫58-B 19 ‘玫瑰花样’‘Rose Petal’ 重瓣 白NN155-D 40 ‘超级黛丝’‘Giantama Deus’ 重瓣 白NN155-D,红54-C 20 ‘绣球’‘Hydrangea’ 单瓣 白NN155-D 41 ‘迷雾’‘Misty’ 单瓣 黄4-C,红46-A 21 ‘迎春’‘Jasminum Nudiflorum’ 重瓣 橙红N34-A,白NN155-C 说明:表中大写字母表示不同的色系代码 表 2
${{L}}^4_5 $ 正交实验因素及水平Table 2.
$L^4_5 $ factors and levers of orthogonal design水平因素 引物/(μmol·L−1) MgCl2/(mmol·L−1) DNA/ng dNTPs/(mmol·L−1) rTaq/(×16.67 nkat) 1 0.1 1.5 30 0.1 0.50 2 0.2 2.0 40 0.2 0.75 3 0.3 2.5 50 0.3 1.00 4 0.4 3.0 60 0.4 1.25 表 3 朱顶红SCoT-PCR正交试验体系筛选
Table 3. Screening of orthogonal systems
处理
编号模板
DNA/ng引物/
(μmol·L−1)Mg2+/
(mmol·L−1)dNTPs/
(mmol·L−1)rTaq/
(×16.67 nkat)处理
编号模板
DNA/ng引物/
(μmol·L−1)Mg2+/
(mmol·L−1)dNTPs/
(mmol·L−1)rTaq/
(×16.67 nkat)1 30 0.1 1.5 0.1 0.50 9 50 0.4 1.5 0.3 0.75 2 30 0.2 2.0 0.2 0.75 10 50 0.3 2.0 0.1 0.50 3 30 0.3 2.5 0.3 1.00 11 50 0.2 2.5 0.4 1.25 4 30 0.4 3.0 0.4 1.25 12 50 0.1 3.0 0.2 1.00 5 40 0.3 1.5 0.2 1.25 13 60 0.2 1.5 0.4 1.00 6 40 0.4 2.0 0.1 1.00 14 60 0.1 2.0 0.3 1.25 7 40 0.1 2.5 0.4 0.75 15 60 0.4 2.5 0.2 0.50 8 40 0.2 3.0 0.3 0.50 16 60 0.3 3.0 0.1 0.75 表 4 SCoT标记引物扩增结果
Table 4. Amplification results of SCoT marker primers
引物编号 引物序列 扩增条带数 多态性条带数 多态性比率/% P5 CAACAATGGCTACCACGA 5 4 80.00 P12 ACGACATGGCGACCAACG 5 3 60.00 P13 ACGACATGGCGACCATCG 9 7 77.78 P32 CCATGGCTACCACCGCAC 10 10 100.00 P40 CCATGGCTACCACCGCCG 5 5 100.00 P41 AACCATGGCTACCACCGA 6 5 83.33 P43 AACCATGGCTACCACCGG 7 7 100.00 P54 ACAATGGCTACCACCAGC 5 4 80.00 P55 ACAATGGCTACCACCAGG 8 6 75.00 P56 ACAATGGCTACCACCAGA 11 11 100.00 P57 ACAATGGCTACCACCAGT 9 7 77.78 P61 ACCATGGCTACCACCGAG 9 8 88.89 平均 7.42 6.42 86.52 合计 89 77 86.52 -
[1] 北京林业大学园林学院花卉教研室. 花卉学[M]. 北京: 中国林业出版社, 2009. [2] 王凤祥. 朱顶红[M]. 北京: 中国林业出版社, 2002. [3] 马慧, 王琪, 袁燕波, 等. 朱顶红属植物种质资源及园林应用[J]. 世界林业研究, 2012, 25(4): 28 − 33. MA Hui, WANG Qi, YUAN Yanbo, et al. Germplasm resources of Hippeastrum spp. and their application to landscaping [J]. World For Res, 2012, 25(4): 28 − 33. [4] 原雅玲, 张延龙. 我国朱顶红生产现状及发展策略[C]//中国园艺学会. 中国园艺学会球根花卉分会2008年年会暨球根花卉产业发展研讨会论文集. 北京: 中国园艺学会, 2008: 193 − 196. [5] MEEROW A W, BROSCHAT T K, KANE M E. Breeding of new Hippeastrum cultivars using diploid species [J]. Acta Hortic, 2015, 325(9): 583 − 589. [6] 张林, 徐迎春, 成海钟, 等. 基于ISSR标记的62个朱顶红品种的遗传关系分析及指纹图谱构建[J]. 植物资源与环境学报, 2012, 21(4): 48 − 54. ZHANG Lin, XU Yingchun, CHENG Haizhong, et al. Genetic relationship analysis and fingerprint construction of 62 cultivars of Hippeastrum spp. based on ISSR marker [J]. J Plant Res Environ, 2012, 21(4): 48 − 54. [7] 张林. 基于形态性状和ISSR标记的朱顶红品种遗传多样性分析及ISSR指纹图谱构建[D]. 南京: 南京农业大学, 2012. ZHANG Lin. Genetic Diversity Analysis and ISSR Fingerprint Construction of Hippeastrum Based on Morphological Characters and ISSR Markers[D]. Nanjing: Nanjing Agricultural University, 2012. [8] COLLARD B C Y, MACKILL D J. Start codon targeted (SCoT) polymorphism: a simple, novel DNA marker technique for generating gene-targeted markers in plants [J]. Plant Mol Biol Rep, 2009, 27: 86 − 93. [9] 龙治坚, 范理璋, 徐刚, 等. SCoT分子标记在植物研究中的应用进展[J]. 植物遗传资源学报, 2015, 16(2): 336 − 343. LONG Zhijiang, FAN Lizhang, XU Gang, et al. Application advance of SCoT molecular markers in plants [J]. J Plant Gen Resour, 2015, 16(2): 336 − 343. [10] 张俊丽. 扇脉杓兰遗传多样性的SCoT分析及扇脉组的亲缘地理学研究[D]. 上海: 华东师范大学, 2016. ZHANG Junli. Genetic Diversity of Cypripedium japonicum Thunb. with SCoT Analysis and the Phylogeography Study of Set. Flabellinervia[D]. Shanghai: East China Normal University, 2016. [11] 熊发前, 蒋菁, 钟瑞春, 等. 目标起始密码子多态性(SCoT)分子标记技术在花生属中的应用[J]. 作物学报, 2010, 36(12): 2055 − 2061. XIONG Faqiang, JIANG Jing, ZHONG Ruichun, et al. Application of SCoT molecular marker in genus Arachis [J]. Acta Agron Sin, 2010, 36(12): 2055 − 2061. [12] 林伟东, 陈志丹, 孙威江, 等. 基于SCoT标记的福建茶树品种(系)遗传多样性分析[J]. 茶叶科学, 2018, 38(1): 43 − 57. LIN Weidong, CHEN Zhidan, SUN Weijiang, et al. Analysis of genetic diversity of fujian tea varieties by SCoT markers [J]. J Tea Sci, 2018, 38(1): 43 − 57. [13] 王令, 李佼, 席彦军, 等. 基于SCoT标记的陕西茶树种质资源遗传多样性分析[J]. 西北农业学报, 2018, 27(2): 244 − 252. WANG Ling, LI Jiao, XI Yanjun, et al. Analysis on genetic diversity of tea germplasm in Shaanxi based on SCoT markers [J]. Acta Agric Boreali-occident Sin, 2018, 27(2): 244 − 252. [14] 潘媛, 陈大霞, 宋旭红, 等. 基于SCoT标记的栽培栀子种质资源遗传多样性研究[J]. 中草药, 2018, 49(14): 3376 − 3381. PAN Yuan, CHEN Daxia, SONG Xuhong, et al. Genetic diversity of cultivated Gardenia jasminoides germplasms detected by SCoT markers [J]. Chin Tradit Herbal Drug, 2018, 49(14): 3376 − 3381. [15] 卓么草, 郭文文, 杨广环, 等. 基于SCoT标记分析西藏白菜型黄籽油菜遗传多样性[J]. 中国油料作物学报, 2018, 40(4): 486 − 491. ZHUO Mecao, GUO Wenwen, YANG Guanghuan, et al. Genetic diversity of Tibet yellow seed rape based on SCoT markers [J]. Chin J Oil Crop Sci, 2018, 40(4): 486 − 491. [16] GAO Yanhui, ZHU YuQiu, TONG Zaikang, et al. Analysis of genetic diversity and relationships among genus Lycoris based on start codon targeted (SCoT) marker [J]. Biochem Syst Ecol, 2014, 57: 221 − 226. [17] 陈伯伦, 张晋, 黄继魁, 等. SCoT分子标记在猕猴桃遗传多样性分析与变异鉴定上的应用[J]. 农业生物技术学报, 2018, 26(1): 77 − 86. CHEN Bolun, ZHANG Jin, HUANG Jikui, et al. Application of SCoT markers on genetic diversity analysis and variation identification of Actinidia [J]. J Agric Biotechnol, 2018, 26(1): 77 − 86. [18] 李东宾, 高燕会, 斯金平. 冷胁迫下铁皮石斛抗寒相关基因的SCoT差异表达分析[J]. 中国中药杂志, 2013, 38(4): 511 − 515. LI Dongbin, GAO Yanhui, SI Jinping. SCoT differential expression of cold resistance related genes in Dendrobium officinale under low temperature stress [J]. China J Chin Mater Med, 2013, 38(4): 511 − 515. [19] 王关林, 方宏筠. 植物基因工程[M]. 2版. 北京: 科学出版社, 2002. [20] 姜小凤. 石蒜属植物杂交育种技术的研究[D]. 杭州: 浙江农林大学, 2013. JIANFG Xiaofeng. Research of Cross-breeding of Lycoris[D]. Hangzhou: Zhejiang A&F University, 2013. [21] 王玉, 于晓艳, 丰震, 等. 玫瑰自然杂交后代数量分类及SCoT标记研究[J]. 园艺学报, 2015, 42(2): 1195 − 1204. WANG Yu, YU Xiaoyan, FENG Zhen, et al. Studies on numerical taxonomy and SCoT marker of rose natural hybrids [J]. Acta Hortic Sin, 2015, 42(2): 1195 − 1204. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20190614







 下载:
下载: