-
GRF(general regulatory factor)蛋白质最先由MOORE等[1]在牛脑中发现,并根据淀粉凝胶电泳上的迁移特性命名。GRF蛋白质是一类高度保守的同源或异源的二聚体蛋白质,具有多种功能,广泛存在于真核生物中,如酵母Pichia guilliermondii、拟南芥Arabidopsis thaliana、水稻Oryza sativa、花生Arachis hypogaea等。已有研究[2]表明:GRF蛋白质家族通过与磷酸化的靶蛋白质相互作用参与植物信号传导、细胞定位、转录调控和应激反应等多种重要生命活动过程,在植物代谢调控和生物合成反应中发挥着重要作用,如拟南芥GRF蛋白质可以与感光系统中的蛋白质相互作用调节根系生长发育[3];葡萄Vitis vinifera GRF蛋白质参与冷热应激反应[4];木薯Manihot esculenta GRF蛋白质主要分布在细胞质中,作用于淀粉合成酶Ⅲ靶蛋白质,对淀粉的合成起到负调控作用[2];菊花Dendranthema morifolium GRF蛋白质参与开花和周期调控,盐、冷等胁迫响应过程[5];动物细胞中GRF蛋白质还可通过调节细胞周期,影响细胞凋亡,参与多种信号通路等方式来调控肿瘤进程[6]。GRF活化后可以使G2/M期阻滞从而起到负调控细胞周期,发挥抑制癌基因的作用[7]。在动物中GRF蛋白质的过表达可能转化为一种致癌因子,促进肿瘤的发生[8],还可能与肿瘤细胞耐药性有关[9]。毛竹Phyllostachys edulis用途广泛,笋和叶具有食用、药用价值;竹材多用于建筑制造、工艺品制作。毛竹林是一种重要的经济林,具有重要生态价值,其固碳作用机制在不同的生长阶段有所差异[10]。毛竹基因组草图已公布,且大量转录组数据也可以从公共数据库中获取[11]。目前根据毛竹全基因组数据进行基因家族分析已取得了一定的成果,如ZF-HD基因家族[12]、B3基因家族[13]、APX基因家族[14]等,也分析了毛竹快速生长期的基因表达[15-16]。但对于毛竹GRF基因家族的全基因组数据分析尚未有相关报道。本研究通过毛竹公开的相关测序结果,利用生物信息学的方法,从基因组及转录组数据入手,对毛竹GRF基因进行全基因组的鉴定与表达分析,拟为进一步明确GRF基因家族在毛竹重要生长发育过程中的功能解析提供依据。
-
毛竹基因组序列、编码序列(CDS)、蛋白质序列和基因组GFF注释文件均从以下站点ftp://parrot.genomics.cn/gigadb/pub/10.5524/100001_101000/100498/[12]下载。从Pfam数据库[17]中下载隐马可夫模型(HMM) PF00244.17的结构域数据,并以此结构域数据为种子模型,用HMMER[18]检索本地毛竹蛋白质数据库。在Excel 2018中,将E-value设置为≤1E−20,对检索结果排序整理,去除重复,获得候选基因。进一步从毛竹全基因组数据库中提取得到GRF家族成员的基因、CDS、蛋白质序列以及基因结构和位置信息;利用在线工具ProtParam(https://web.expasy.org/protparam/)、ProtScale(https://web.expasy.org/protscale/)[19]以及SignalP 4.1[20]在线分析GRF家族各成员理化性质等。
-
依据毛竹、拟南芥、水稻GRF家族成员蛋白质序列,分别通过ClustalW多重比对,用MEGA 7.0软件邻位连接(neighbor-Joining, NJ)法构建种内和种间系统进化树,自检值取1 000次抽样[21]。
-
根据毛竹全基因组的GFF注释文件基因位置信息,分析毛竹GRF家族的基因结构并绘制基因结构图;利用在线网站NCBI Conserve Domain(https://www.ncbi.nlm.nih.gov/cdd/)和MEME(https://www.ncbi.nlm.nih.gov/cdd/)对GRF家族成员的保守结构域(domain)和基序(motif)进行预测[22],并通过TBtools[23]将结果可视化。
-
提取毛竹GRF基因上游1 500 bp序列作为启动子序列信息,通过在线预测软件PlantCare[24]预测毛竹GRF基因的顺式作用元件,并整理预测结果,富集顺式作用元件,利用TBtools上的Simple Biosequence viewer功能进行可视化分析。
-
利用MCScanX[25]获取GRF家族种内、种间共线性关系,并用TBtools软件Amazing Super Circos[26]和Multipe Synteny Plot分别对种内和种间的结果可视化。
-
选取NCBI SRA数据库中毛竹不同组织器官:根(登录号为ERR105075、ERR105076),花序(登录号为ERR105069、ERR105070、ERR105071),叶(登录号为ERR105067、ERR105068、ERR105075),鞭(登录号为ERR105073、ERR105074)和笋不同生长高度:0.2 m(登录号为SRR6131114、SRR131113、SRR6131115),0.5 m(登录号为SRR131117、SRR6131118、SRR5710699)和1.0 m(登录号为SRR5710701、SRR5710702、SRR5710697)的转录组数据,分别计算毛竹GRF基因的TPM(transcripts per million reads)值表示基因的表达丰度。为方便统计,对每个表达数值取以2为底的对数(log2),使用TBtools Amazing Heatmap绘制基因表达热图,用对数转换预处理数据,再用正态标准化的方法处理数据。
-
利用SWISSMODEL(https://www.swissmodel.expasy.org/)在线软件[27]预测GRF蛋白质的3D结构。模建结果使用SAVES v5.0[19]进行评估。
-
根据植物GRF隐马可夫模型(PF00244.17)搜索毛竹相关基因组数据,获得相关GRF家族成员,然后通过E-value(≤1E−20)筛选、保守结构域、基序特征分析,去除相同转录本重复,最终筛选得到13个GRF家族成员(表1)。将获得13个GRF家族成员按照其在scaffold的分布先后顺序命名为PeGRF01~PeGRF13。进一步对PeGRF作蛋白质特性分析,13个GRF蛋白质中长度最短的为PeGRF10(256个氨基酸),最长的为PeGRF09(293个氨基酸),平均长度266.8个氨基酸;各GRF蛋白质等电点最小的为4.70(PeGRF02),最大的为5.29(PeGRF01),平均等电点为4.82;各GRF蛋白质分子量最小的为PeGRF04(28.65 kD),最大的为PeGRF09(32.41 kD),平均分子量为29.79 kD。
表 1 毛竹GRF基因及其蛋白质理化特性
Table 1. Characteristics of PeGRF family genes and their deduced proteins
基因登录号 基因名称 等电点(pI) 平均分子量/kD 内含子数量/个 氨基酸数量/个 PH02Gene26029.t1 PeGRF01 5.29 32.20 5 286 PH02Gene21972.t1 PeGRF02 4.70 29.68 4 262 PH02Gene06378.t1 PeGRF03 4.79 29.14 4 263 PH02Gene19868.t1 PeGRF04 4.82 28.65 3 261 PH02Gene15394.t1 PeGRF05 4.76 31.08 4 274 PH02Gene31988.t1 PeGRF06 4.73 29.94 6 270 PH02Gene44376.t1 PeGRF07 4.79 29.15 4 263 PH02Gene09923.t1 PeGRF08 4.86 29.28 4 261 PH02Gene25395.t2 PeGRF09 4.72 32.41 5 293 PH02Gene13806.t1 PeGRF10 4.76 29.02 4 256 PH02Gene13908.t1 PeGRF11 4.82 29.15 4 260 PH02Gene26176.t1 PeGRF12 4.84 28.76 3 263 PH02Gene15240.t1 PeGRF13 4.75 28.77 4 256 -
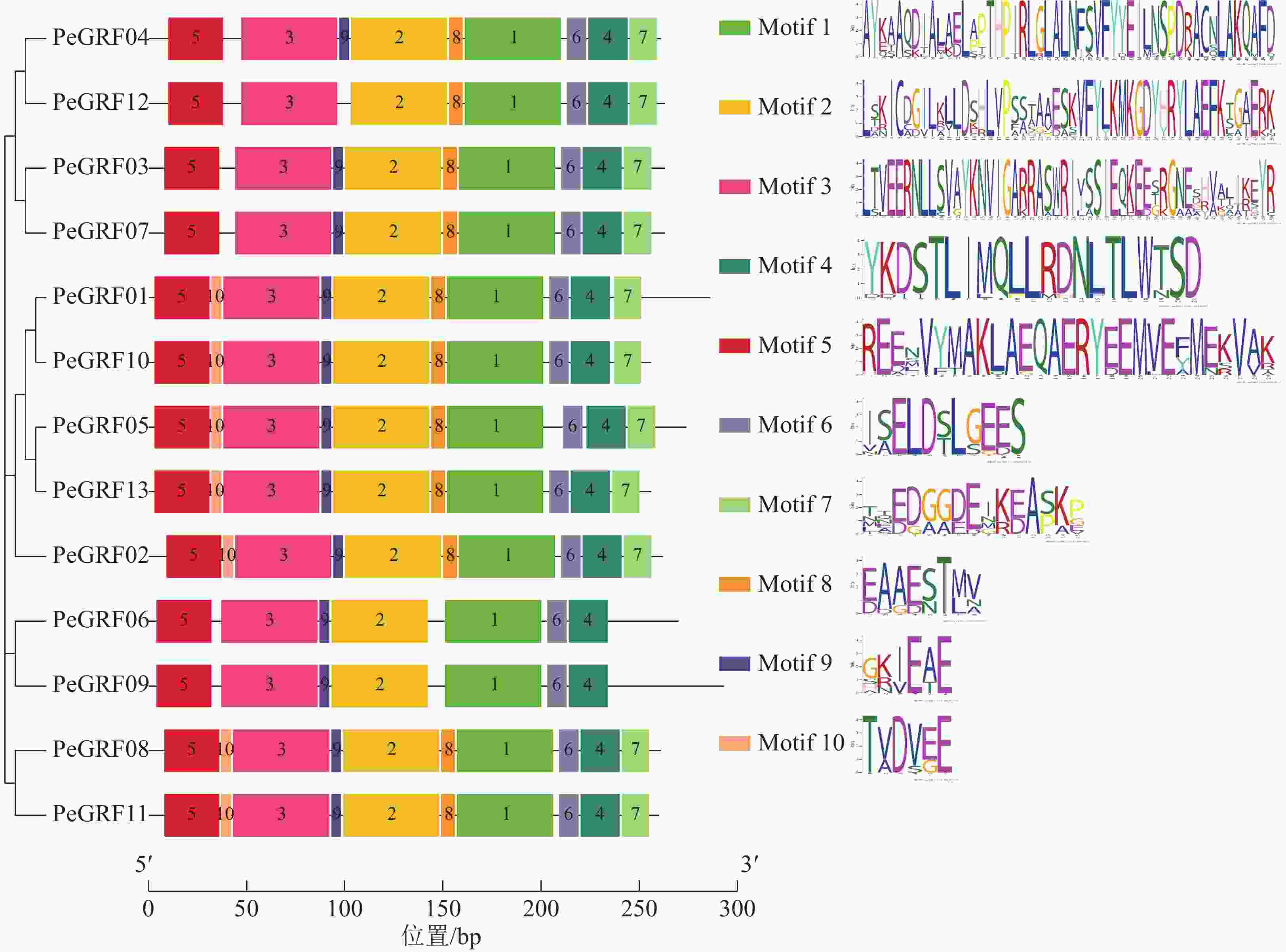
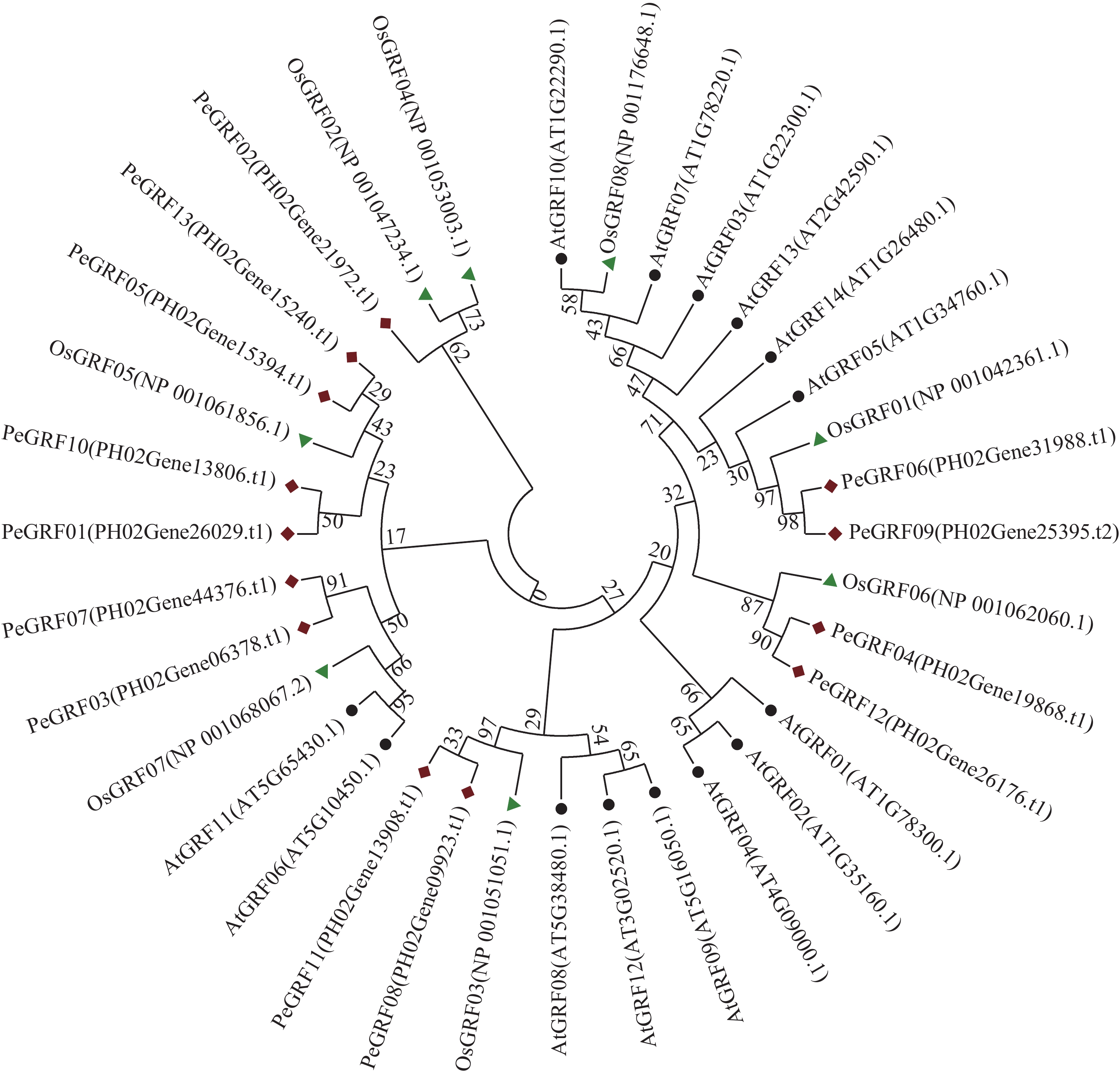
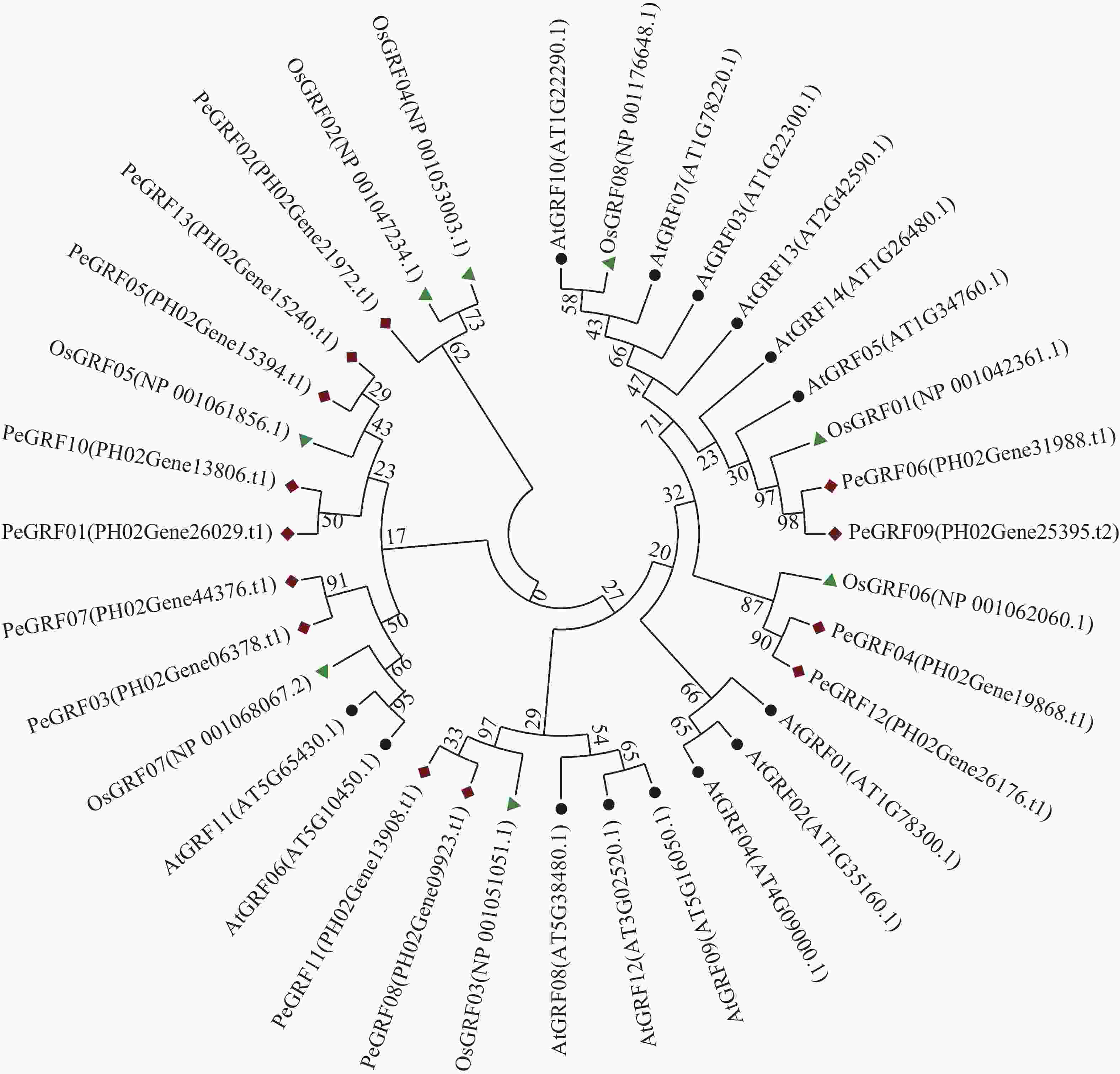
利用MEGA 7.0对13个毛竹GRF、14个拟南芥GRF和8个水稻GRF的氨基酸序列比对后,采用NJ法进行系统聚类分析(图1),绝大部分毛竹基因家族成员和水稻处于同一分支,表明毛竹与水稻的进化关系较近。
-
对毛竹GRF基因结构分析发现:内含子数量存在差异,非ε组成员都包含4个外显子和3个内含子,它们在位置上高度保守。ε组成员都具有不同于非ε组的内含子-外显子结构,具有2个额外的N-末端内含子[21]。利用NCBI-CDD对毛竹GRF基因进行保守结构域分析,PeGRF蛋白质均包含14/3/3结构域,毛竹GRF基因家族14/3/3结构域存在一定的保守性,但该结构域的分布位置有一定分化。利用MEME在线工具对该基因家族的保守基序预测,基数设置为10,结果显示(图2):Motif1~6在每个家族成员中均出现,属于高度保守结构,其余基序在家族成员中出现的频率及所在位置均存在一定的差异。
-
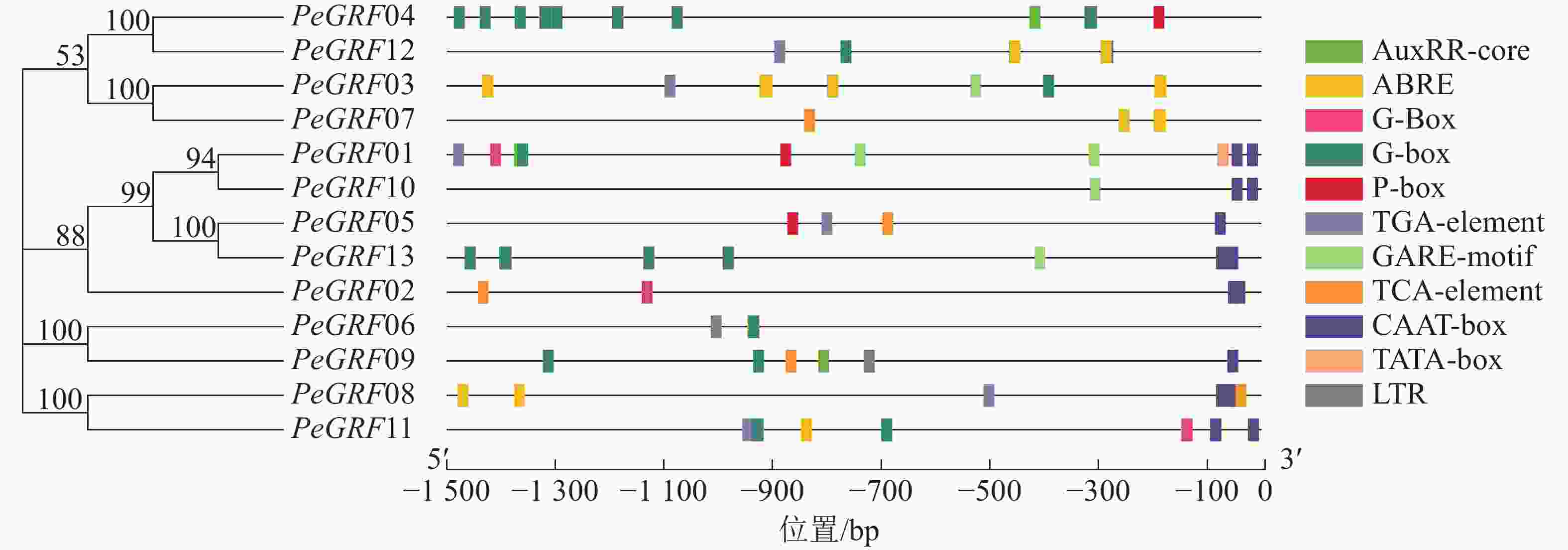
如图3所示:筛选出的部分典型的顺式调控元件,除核心启动子TATA-box(5个)和CAAT-box(16个)外,还有与激素相关的顺式调控元件,包括与赤霉素相关的GARE-motif(5个)、P-box(3个),与生长素有关的AuxRR-core(3个)、TGA-element(6个),与脱落酸有关的ABRE(42个),与水杨酸有关的TCA-element(5个);与外部条件有关的顺式调控元件,包括参与低温响应的LTR(2个)和光响应的G-box(48个)。推测毛竹GRF蛋白质家族可能参与激素和非生物胁迫响应,家族基因表达模式可能有所不同。
-
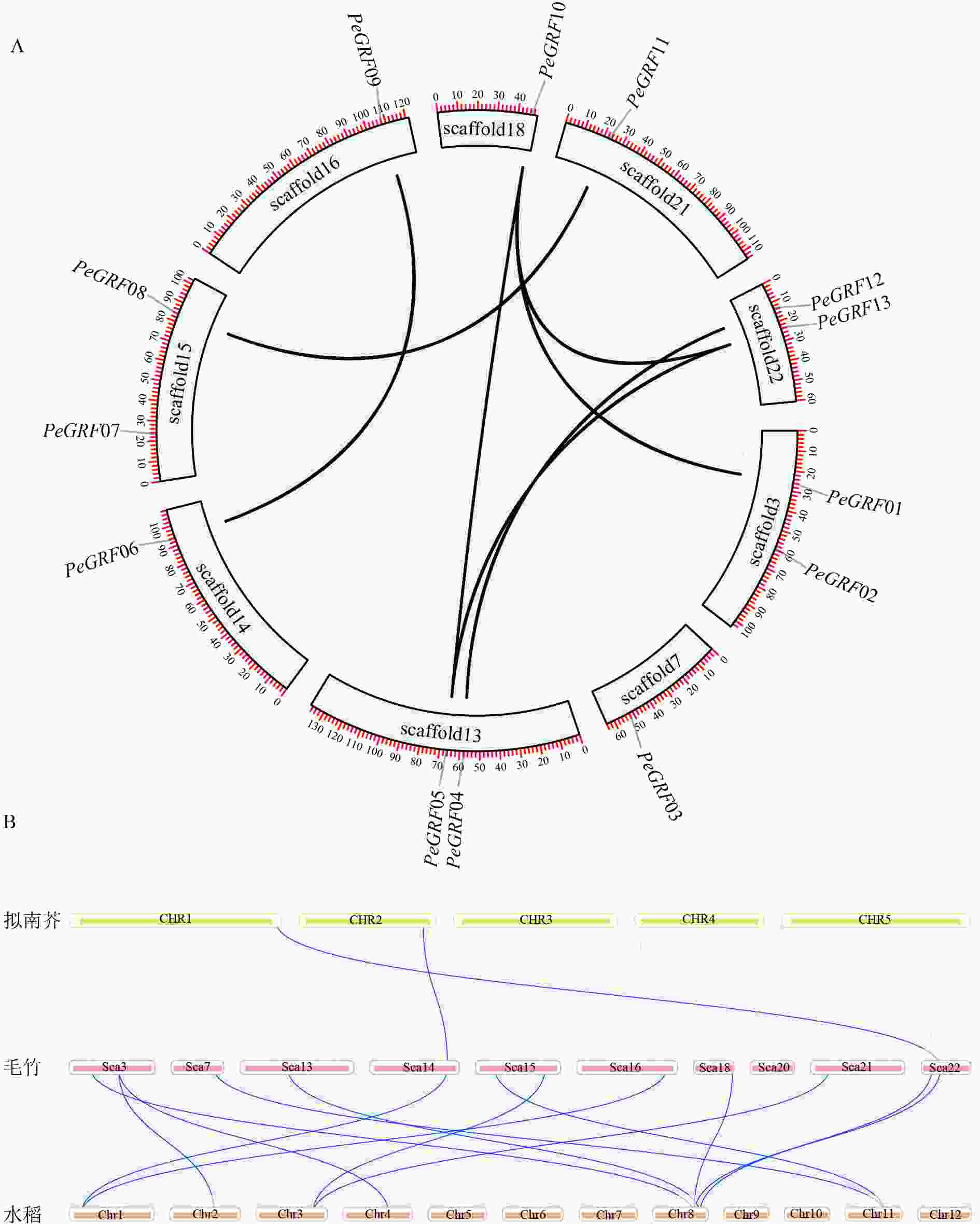
利用毛竹基因组GFF注释文件提取PeGRF在scaffold上的分布特征,结果显示:毛竹GRF基因在scaffold上分布不均匀,不同的scaffold基因分布密度不同,scaffold7、14、16、18和21仅包含1个PeGRF,scaffold3、13、15和22上分别包含2个。
利用TBtools工具,将毛竹GRF基因种内和种间的共线性关系进行了可视化分析。从图4A中可以看出:除PeGRF02、PeGRF03和PeGRF07不存在种内共线性关系外,其余家族基因成员间均有显著的共线性关系,说明GRF基因家族存在基因复制现象,推测在进化过程中GFR基因可能通过复制进行家族成员数量的扩张。但PeGRF不存在串联重复基因。物种间的共线性关系是反映不同物种来源于同一个祖先的现象。从图4B可以看出:毛竹与水稻的共线性关系要明显多于拟南芥,这可能与水稻和毛竹同属于禾本科Gramineae,进化关系较近有关。
-
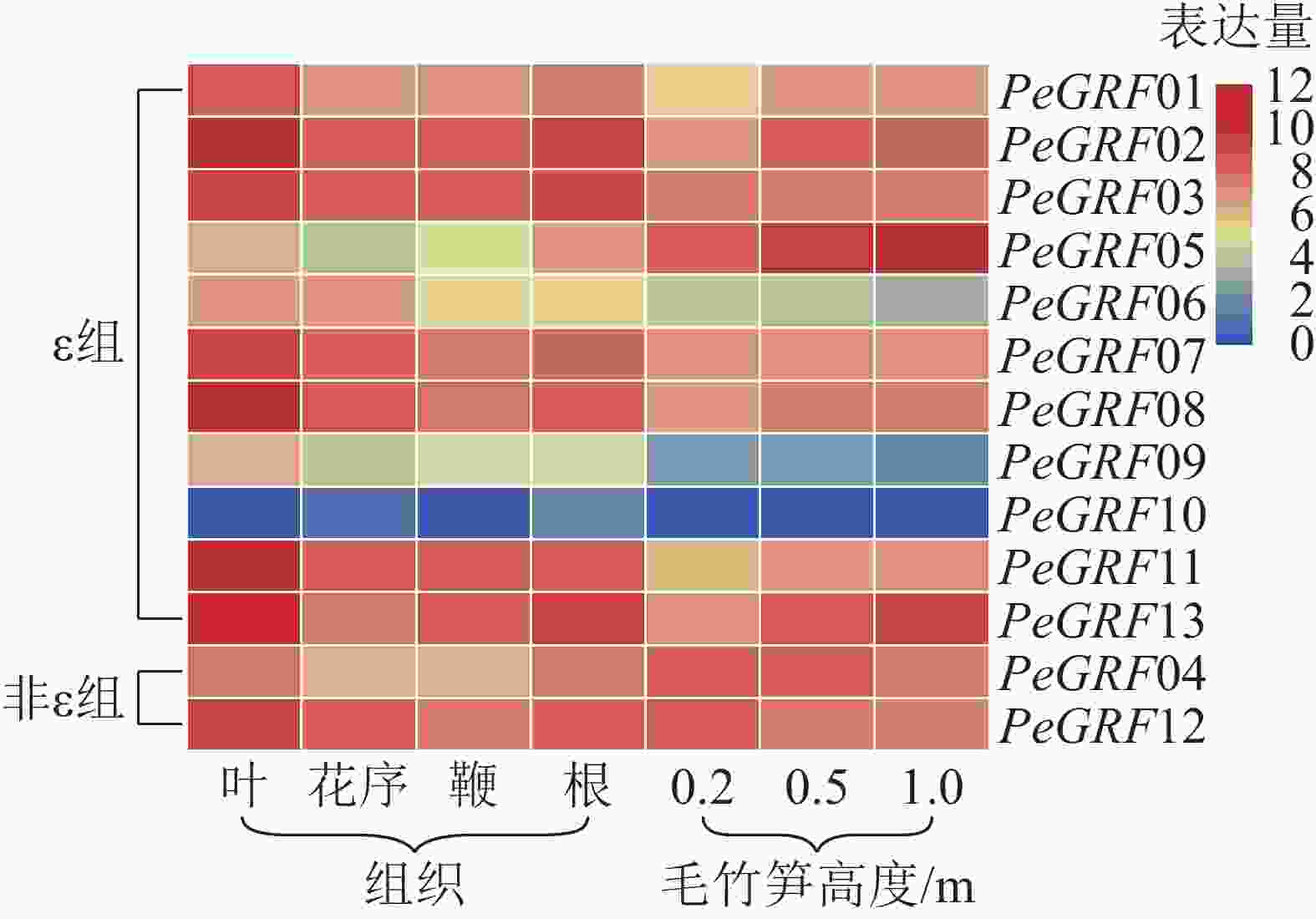
本研究基于毛竹RNA-Seq转录组数据,对毛竹不同组织(叶、花序、鞭及根)以及不同生长高度(0.2、0.5、1.0 m)的毛竹笋中的GRF表达量绘制热图。由图5可以看出:除PeGRF10,PeGRF09在不同组织和生长高度保持较低的表达量外,其他成员均有较高的表达量。在毛竹不同组织中,根和花序的表达量相对于叶和鞭要稍高;非ε组的GRF基因均有较高的表达。在竹笋的不同生长阶段,非ε组的GRF基因保持较高的表达水平;ε组不同的基因表达量有增有减,如PeGRF05在竹笋生长各个阶段均有较高的表达量,且随生长进程表达量不断增高;PeGRF06表达量随生长进程呈下降趋势。推测不同家族成员在参与组织器官发育的过程中发挥不同的作用,但其中的内在分子机制还值得进一步研究。
-
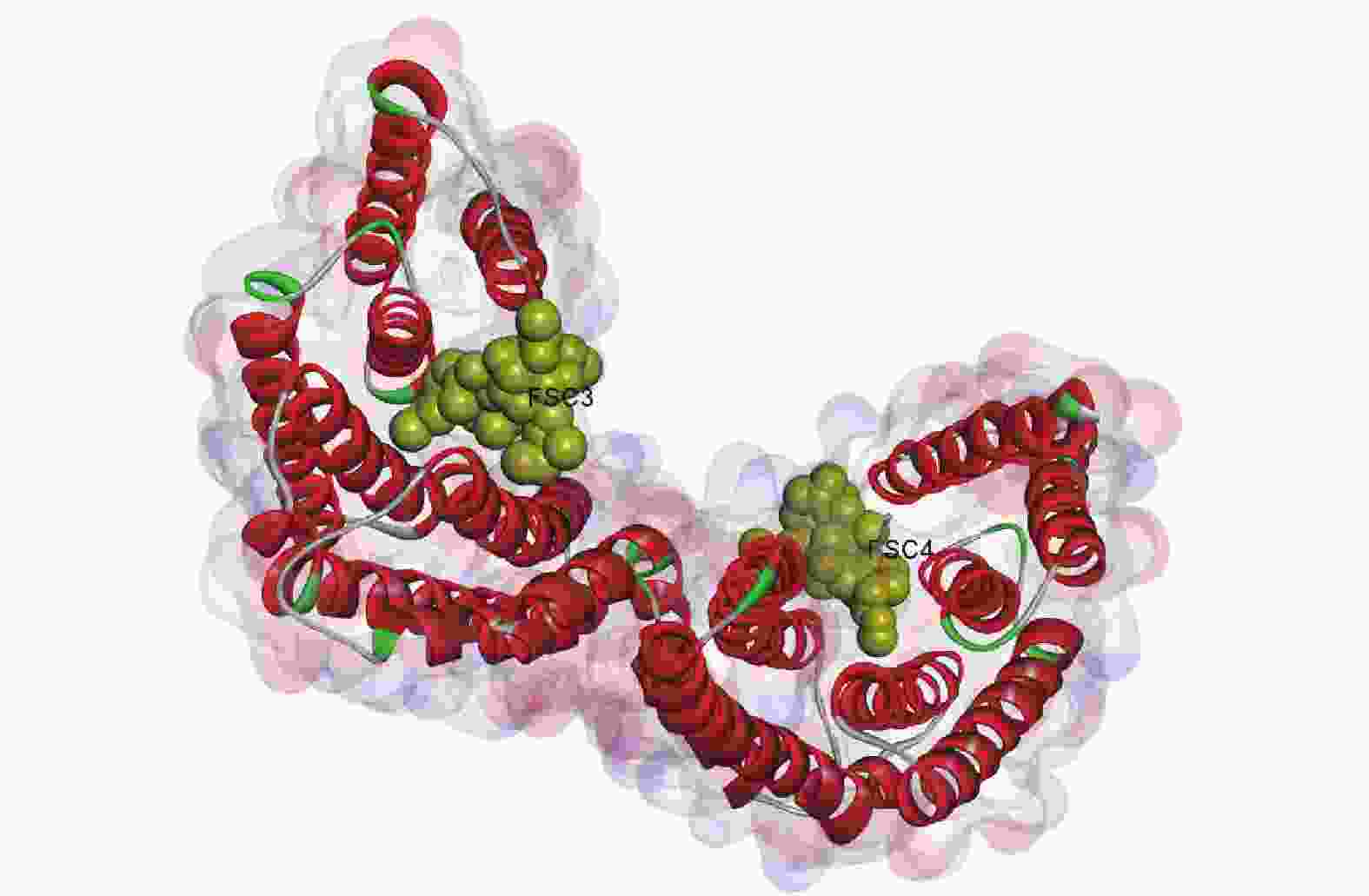
由图6所示:毛竹GRF蛋白质由2个单体连接而成,每个单体由反向平行的9个α螺旋组成,每个单体都存在与配体(FSC3、FEC4)相互作用的结合位点,2个FSC配体均与壳梭孢素有关,单体间构成同源或异源二聚体,总体呈“W”型[28-29]。
-
物种基因组全序列的测定推动了生物信息学的迅速发展,在海量数据的基础上,利用生物信息学手段,对物种基因家族进行高效的统计分类和分析,预测基因家族的结构、功能及作用机制,将极大地推动相关功能基因的挖掘和农艺性状遗传的改良进程[30]。随着2018年第2版毛竹基因组数据的公布以及大量毛竹转录组数据的共享,毛竹GRF基因家族的生物信息学分析成为可能[11]。本研究通过全基因组数据分析发现:毛竹GRF家族成员共13个,数量多于水稻,可能的原因是毛竹染色体经过加倍,基因组数据远大于水稻;另外,共线性分析进一步证实:正是通过基因复制扩增,毛竹GRF在数量上有优势。毛竹GRF基因家族各成员间的理化性质存在一定的差异,但均含有14/3/3蛋白质结构域,其中有6种基序在每个成员中均出现。根据基因结构将PeGRF分为ε组和非ε组,其中ε组可能保留了祖先的蛋白质功能,这与PIOTROWSKI等[31]和WANG等[32]的研究结果相似。
大量研究表明GRF蛋白质参与激素信号的转导。如在拟南芥的研究中发现:GRF参与油菜素类激素(BR)调控细胞核发育的途径[33];在烟草Nicotiana tabacum中,GRF参与赤霉素(GA)生物合成调控[34];在水稻中,GRF表达同脱落酸(ABA)密切相关[35]。本研究发现:毛竹GRF顺式作用元件存在许多激素相关元件。由此可以推测毛竹GRF蛋白质可能介导激素信号的转导过程。但毛竹GRF同其他激素的相互关系还需进一步验证。
GRF蛋白质参与了植物的生长发育,特别是在花器官的发育中具有重要作用。PERTL等[36]证实随着百合Lilium brownii var. viridulum花粉管的生长,GRF蛋白质的表达量也明显增加。李兵娟[37]也证实雷竹Phyllostachys violascens GRF基因参与开花调控机制。本研究通过转录组数据分析发现:GRF蛋白质在花序组织中高表达,且表达量明显高于竹叶和竹鞭,这表明毛竹GRF基因可能参与花序的发育和调控。除此之外,在研究毛竹GRF顺式作用元件时还发现其启动子区域存在许多光响应元件,结合光周期对植物开花的作用机制以及在模式植物水稻上的研究[38],GRF基因可能是通过光响应元件接受外界环境信号从而触发其高表达,最终影响毛竹花的发育。由于受毛竹花发育相关材料的限制,该假设将在后续实验验证。
毛竹GRF蛋白质是以一个螺旋结构为主的同源二聚体,二聚体界面内包着多个疏水残基和多个极性残基,外周则由盐桥连接,三级结构呈“W”型,每个单体分别含有2个凹槽,可能用于结合配体靶蛋白质。毛竹GRF蛋白质序列在进化谱系中高度保守,并且与配体结合的氨基酸残基极端保守,这同SEHNKE等[28]发现的结果相似。另外,虽然毛竹GRF蛋白质的N端和C端同源性较低,但可能通过碱性簇维持空间构象的稳定[28]。PAUL等[39]在研究拟南芥GRF蛋白质时发现,GRF蛋白质还可以通过结合磷酸化的蛋白质,参与重力反应等生理过程。GRF蛋白质在进化上高度保守,毛竹PeGRF可能也具有相似的分子作用机制。但毛竹GRF蛋白质生物学功能与上述空间结构之间的关系还需进一步的探索。
Genome identification and expression analysis of GRF gene family in Phyllostachys edulis
-
摘要:
目的 探究毛竹Phyllostachys edulis GRF基因家族的性质、结构特点以及在不同组织中的表达水平,为进一步研究GRF在毛竹生长发育中的分子作用机制奠定基础。 方法 采用生物信息学方法,对毛竹全基因组和转录组信息进行分析,筛选出13个毛竹GRF基因家族成员,并对GRF基因家族的理化性质、进化、基因结构、保守结构域、启动子、基因家族表达模式及三级结构进行分析。 结果 毛竹GRF基因家族成员按照其在scaffold上分布的位置,分别被命名为PeGRF01~PeGRF13;将含有3个内含子的PeGRF04和PeGRF12归为非ε组,其余为ε组。毛竹各GRF基因家族成员理化性质存在一定的差异,但是其结构域相对保守,均含有14/3/3结构域。毛竹GRF家族启动子区含有大量与光响应、低温响应、激素调控等相关的顺式作用元件。毛竹GRF存在基因复制扩增的现象,与水稻Oryza sativa的共线性关系明显高于拟南芥Arabidopsis thaliana。毛竹GRF在不同组织器官中均有表达,各家族成员的表达量存在差异;在毛竹花和根组织中的表达量略高于叶和鞭,同时家族成员间的表达量也存在一定差异。GRF蛋白质由2个单体构成,每个单体由9个α-螺旋构成,整体结构呈“W”型。 结论 毛竹GRF基因家族具有典型的14/3/3结构域,可能参与根、鞭、叶、花序及笋芽的生长发育过程。图6表1参39 Abstract:Objective This study aims to explore the nature and structural features of general regulatory factor(GRF) gene family in moso bamboo(Phyllostachys edulis) and its expression levels in different tissues, so as to lay the foundation for further study on the molecular mechanism of GRF in growth and development of Ph.edulis. Method Bioinformatics approach was used to analyze the whole genome and transcriptome information of Ph. edulis, and 13 members of GRF gene family were selected and analyzed for their physicochemical properties, evolution, gene structure, conserved domains, promoters, gene family expression patterns, and tertiary structure. Result Members of the GRF gene family in Ph. edulis were named PeGRF01~PeGRF13 according to their distribution on the scaffold. PeGRF04 and PeGRF12 with 3 introns were classified as non-ε group, and the rest were ε group. The physicochemical properties of the members of each GRF gene family differed, but their domains were relatively conservative, all containing 14/3/3 domains. The promoter region of GRF family in Ph. edulis contained a large number of cis-acting elements related to light response, low temperature response, and hormone regulation. The gene duplication and amplification in PeGRF was significantly higher in collinearity with Oryza sativa than with Arabidopsis thaliana. PeGRFs were expressed in different tissues and organs, and the expression levels of each family member were different. The expression levels in panicle and root tissues of Ph. edulis were slightly higher than those in leaf and rhizome, and there were some differences among family members. The GRF protein was composed of 2 monomers, each of which was composed of 9 α-helices, and the overall structure was W-shaped. Conclusion The GRF gene family of Ph. edulis has a typical 14/3/3 domain, which may be involved in the development of roots, rhizomes, leaves, panicles and shoots. [Ch, 6 fig. 1 tab. 39 ref.] -
Key words:
- Phyllostachys edulis /
- GRF gene family /
- phylogenetics /
- collinearity /
- gene expression level
-
表 1 毛竹GRF基因及其蛋白质理化特性
Table 1. Characteristics of PeGRF family genes and their deduced proteins
基因登录号 基因名称 等电点(pI) 平均分子量/kD 内含子数量/个 氨基酸数量/个 PH02Gene26029.t1 PeGRF01 5.29 32.20 5 286 PH02Gene21972.t1 PeGRF02 4.70 29.68 4 262 PH02Gene06378.t1 PeGRF03 4.79 29.14 4 263 PH02Gene19868.t1 PeGRF04 4.82 28.65 3 261 PH02Gene15394.t1 PeGRF05 4.76 31.08 4 274 PH02Gene31988.t1 PeGRF06 4.73 29.94 6 270 PH02Gene44376.t1 PeGRF07 4.79 29.15 4 263 PH02Gene09923.t1 PeGRF08 4.86 29.28 4 261 PH02Gene25395.t2 PeGRF09 4.72 32.41 5 293 PH02Gene13806.t1 PeGRF10 4.76 29.02 4 256 PH02Gene13908.t1 PeGRF11 4.82 29.15 4 260 PH02Gene26176.t1 PeGRF12 4.84 28.76 3 263 PH02Gene15240.t1 PeGRF13 4.75 28.77 4 256 -
[1] MOORE B W, PEREZ V J. Specific acidic proteins of the nervous system[M]//CARLSON F D. Physiological and Biochemical Aspects of Nervous Integration. Englewood Cliffs: Prentice-Hall, 1968: 343−359. [2] 潘冉冉, 秦于玲, 乔景娟, 等. 14-3-3蛋白结构与功能预测及其在木薯成熟期的表达分析[J]. 中国农学通报, 2018, 34(36): 49 − 57. PAN Ranran, QIN Yuling, QIAO Jingjuan, et al. The structure, function prediction of 14-3-3 protein and its expression at maturity stage of cassava [J]. Chin Agric Sci Bull, 2018, 34(36): 49 − 57. [3] MAYFIELD J D, PAUL A L, FERL R J, et al. The 14-3-3 proteins of Arabidopsis regulate root growth and chloroplast development as components of the photosensory system [J]. J Exp Bot, 2012, 63(8): 3061 − 3070. [4] CHENG Cheng, WANG Yi, CHAI Fengmei, et al. Genome-wide identification and characterization of the 14-3-3 family in Vitis vinifera L. during berry development and cold- and heat-stress response[J]. BMC Genomics, 2018, 19(1): 579. doi: 10.1186/s12864-018-4955-8. [5] 曹沛沛, 毛雅超, 刘涛, 等. 菊花Cm14-3-3υ基因的克隆及表达分析[J]. 南京农业大学学报, 2017, 40(5): 820 − 826. CAO Peipei, MAO Yachao, LIU Tao, et al. Cloning and expression analysis of Cm14-3-3υ gene in chrysanthemum [J]. J Nanjing Agric Univ, 2017, 40(5): 820 − 826. [6] 李易桐, 付琰, 张雅婷, 等. 14-3-3蛋白在胃癌中的作用及机制研究进展[J]. 解放军医学杂志, 2020, 45(1): 97 − 101. LI Yitong, FU Yan, ZHANG Yating, et al. Progress of effects of 14-3-3 protein on gastric cancer and its mechanism [J]. Med J Chin People ’s Lib Army, 2020, 45(1): 97 − 101. [7] HYNES N E, SMIRNOVA T. The 14-3-3σ tumor suppressor has multiple functions in ErbB2-induced breast cancer [J]. Cancer Discov, 2012, 2(1): 19 − 22. [8] YOO J O, KWAK S Y, AN H J, et al. miR-181b-3p promotes epithelial–mesenchymal transition in breast cancer cells through Snail stabilization by directly targeting YWHAG [J]. Biochim Biophys Acta Mol Cell Res, 2016, 1863(7): 1601 − 1611. [9] TSENG C W, YANG J C, CHEN C N, et al. Identification of 14-3-3β in human gastric cancer cells and its potency as a diagnostic and prognostic biomarker [J]. Proteomics, 2011, 11(12): 2423 − 2439. [10] 周国模, 姜培坤. 毛竹林的碳密度和碳贮量及其空间分布[J]. 林业科学, 2004, 40(6): 20 − 24. ZHOU Guomo, JIANG Peikun. Density, storage and spatial distribution of carbon in Phyllostachy pubescens forest [J]. Sci Silv Sin, 2004, 40(6): 20 − 24. [11] 黎帮勇, 胡尚连, 曹颖, 等. 毛竹NAC转录因子家族生物信息学分析[J]. 基因组学与应用生物学, 2015, 34(8): 1769 − 1777. LI Bangyong, HU Shanglian, CAO Ying, et al. Bioinformatics analysis of NAC gene family in moso bamboo [J]. Genomics Appl Biol, 2015, 34(8): 1769 − 1777. [12] 马瑞芳, 陈家璐, 刘笑雨, 等. 毛竹锌指同源结构域基因家族全基因组鉴定及表达分析[J]. 农业生物技术学报, 2020, 28(4): 645 − 657. MA Ruifang, CHEN Jialu, LIU Xiaoyu, et al. Genome-wide identification and expression analysis of zinc finger homologous domain gene family in Phyllostachys edulis [J]. J Agric Biotechnol, 2020, 28(4): 645 − 657. [13] 吴佳军, 俞率成, 刘志刚, 等. 毛竹B3家族全基因组鉴定及表达模式分析[J]. 农业生物技术学报, 2019, 27(1): 43 − 54. WU Jiajun, YU Shuaicheng, LIU Zhigang, et al. Genome identification and expression pattern analysis of Phyllostachys edulis B3 family [J]. J Agric Biotechnol, 2019, 27(1): 43 − 54. [14] 宋笑龙, 孔波, 高志民, 等. 毛竹APX家族基因鉴定和表达分析[J]. 热带亚热带植物学报, 2020, 28(3): 255 − 264. SONG Xiaolong, KONG Bo, GAO Zhimin, et al. Identification and expression analysis of the APX gene family in Phyllostachys edulis [J]. J Trop Subtrop Bot, 2020, 28(3): 255 − 264. [15] 卜柯丽, 傅卢成, 王灵杰, 等. 毛竹茎秆快速生长期PeATG1/PeATG4基因表达分析[J]. 浙江农林大学学报, 2020, 37(1): 43 − 50. BU Keli, FU Lucheng, WANG Lingjie, et al. Analysis of PeATG1/PeATG4 gene expression in Phyllostachys edulis during rapid growth [J]. J Zhejiang A&F Univ, 2020, 37(1): 43 − 50. [16] 栗青丽, 王灵杰, 高培军, 等. 竹茎秆快速生长期淀粉分解相关酶基因表达的分析[J]. 浙江农林大学学报, 2020, 37(6): 1128 − 1135. LI Qingli, WANG Lingjie, GAO Peijun, et al. Gene expression of starch decomposing enzymes in Phyllostachys edulis stems during the rapid growth period [J]. J Zhejiang A&F Univ, 2020, 37(6): 1128 − 1135. [17] FINN R D, COGGILL P, EBERHARDT R Y, et al. The Pfam protein families database: towards a more sustainable future [J]. Nucleic Acids Res, 2016, 44(D1): 279 − 285. [18] FINN R D, CLEMENTS J, EDDY S R. HMMER web server: interactive sequence similarity searching [J]. Nucleic Acids Res, 2011, 39(S1): W29 − W37. [19] 陈家璐, 张智俊, 刘笑雨, 等. 毛竹Dirigent基因家族的全基因组鉴定与分析[J]. 植物生理学报, 2019, 55(9): 1406 − 1417. CHEN Jialu, ZHANG Zhijun, LIU Xiaoyu, et al. Genome-wide identification and analysis of Dirigent gene family in moso bamboo (Phyllostachys edulis) [J]. Plant Physiol J, 2019, 55(9): 1406 − 1417. [20] PETERSEN T N, BRUNAK S, VON HEIJNE G, et al. SignalP 4.0: discriminating signal peptides from transmembrane regions [J]. Nat Methods, 2011, 8(10): 785 − 786. [21] KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets [J]. Mol Biol Evol, 2016, 33(7): 1870 − 1874. [22] BAILEY T L, BODEN M, BUSKE F A, et al. MEME SUITE: tools for motif discovery and searching [J]. Nucleic Acids Res, 2009, 37(S2): W202 − W208. [23] CHEN Chengjie, CHEN Hao, ZHANG Yi, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data [J]. Mol Plant, 2020, 13(8): 1194 − 1202. [24] LESCOT M, DÉHAIS P, THIJS G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences [J]. Nucleic Acids Res, 2002, 30(1): 325 − 327. [25] WANG Yupeng, TANG Haibao, DEBARRY J D, et al. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Res, 2012, 40(7): e49. doi: 10.1093/nar/gkr1293. [26] KRZYWINSKI M, SCHEIN J, BIROL I, et al. Circos: an information aesthetic for comparative genomics [J]. Genome Res, 2009, 19(9): 1639 − 1645. [27] WATERHOUSE A, BERTONI M, BIENERT S, et al. SWISS-MODEL: homology modelling of protein structures and complexes [J]. Nucleic Acids Res, 2018, 46(W1): W296 − W303. [28] SEHNKE P C, DELILLE J M, FERL R J. Consummating signal transduction: the role of 14-3-3 proteins in the completion of signal-induced transitions in protein activity [J]. Plant Cell, 2002, 14(suppl 1): S339 − S354. [29] MANAK M S, FERL R J. Divalent cation effects on interactions between multiple Arabidopsis 14-3-3 isoforms and phosphopeptide targets [J]. Biochemistry, 2007, 46(4): 1055 − 1063. [30] 张长青, 王进, 李广平, 等. 园艺植物分子育种相关生物信息资源及其应用[J]. 植物学通报, 2005, 22(4): 494 − 501. ZHANG Changqing, WANG Jin, LI Guangping, et al. Bioinformatics resources for molecular breeding of horticultural plants [J]. Chin Bull Bot, 2005, 22(4): 494 − 501. [31] PIOTROWSKI M, OECKING C. Five new 14-3-3 isoforms from Nicotiana tabacum L.: implications for the phylogeny of plant 14-3-3 proteins [J]. Planta, 1998, 204(1): 127 − 130. [32] WANG Wenfu, SHAKES D C. Molecular evolution of the 14-3-3 protein family [J]. J Mol Evol, 1996, 43(4): 384 − 398. [33] CHANG I F, CURRAN A, WOOLSEY R, et al. Proteomic profiling of tandem affinity purified 14-3-3 protein complexes in Arabidopsis thaliana [J]. Proteomics, 2009, 9(11): 2967 − 2985. [34] ISHIDA S, FUKAZAWA J, YUASA T, et al. Involvement of 14-3-3 signaling protein binding in the functional regulation of the transcriptional activator REPRESSION OF SHOOT GROWTH by gibberellins [J]. Plant Cell, 2004, 16(10): 2641 − 2651. [35] CHEN Yixing, ZHOU Xiaojin, CHANG Shu, et al. Calcium-dependent protein kinase 21 phosphorylates 14-3-3 proteins in response to ABA signaling and salt stress in rice [J]. Biochem Biophys Res Commun, 2017, 493(4): 1450 − 1456. [36] PERTL H, HIMLY M, GEHWOLF R, et al. Molecular and physiological characterisation of a 14-3-3 protein from lily pollen grains regulating the activity of the plasma membrane H+ ATPase during pollen grain germination and tube growth [J]. Planta, 2001, 213(1): 132 − 141. [37] 李兵娟. 雷竹14-3-3基因家族克隆和功能分析[D]. 杭州: 浙江农林大学, 2014. LI Bingjuan. Molecular Cloning and Functional Analysis of 14-3-3 Genes in Phyllostachys violascens[D]. Hangzhou: Zhejiang A&F University, 2014. [38] 袁玺垒, 王振山, 贾小平, 等. 光周期调控植物开花分子机制以及CCT基因家族研究进展[J]. 浙江农业学报, 2020, 32(6): 1133 − 1140. YUAN Xilei, WANG Zhenshan, JIA Xiaoping, et al. Research advances on molecular mechanisms of photoperiod-regulation plant flowering and CCT gene family [J]. Acta Agric Zhejiang, 2020, 32(6): 1133 − 1140. [39] PAUL A L, DENISON F C, SCHULTZ E R, et al. 14-3-3 phosphoprotein interaction networks-does isoform diversity present functional interaction specification?[J]. Front Plant Sci, 2012, 3: 190. doi: 10.3389/fpls.2012.00190. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20200544






 下载:
下载: