-
高等植物成花受到自身遗传因素和外界环境因素的共同影响[1-2]。当植物完成成花诱导,花器官原基开始分化,逐渐形成花萼、花瓣、雄蕊和心皮等组织。在这期间,植物的茎尖分生组织形态发生了很大的变化,同时成花素FT(FLOWERING LOCUS T)的表达量显著增加并参与调控花芽分化[3]。在模式植物拟南芥Arabidopsis thaliana以及许多物种中已经证实,FT可以通过激活花芽分化组织特异性基因AP1(APETALA1)的表达促使植物完成开花进程[4]。过表达AP1基因,不论在长、短日照下拟南芥都会出现早花表型[5],其突变体ap1则出现明显的晚花表型[6]。在茎尖分生组织,AP1基因不仅在植物成花诱导的调控网络中起核心作用,而且也决定花器官的形成。AP1基因是植物ABCDE分化模型中的A类基因,控制第一轮花萼和第二轮花被片组织的形成[7],同时AP1也调控SEP3(SEPALLATA3)与LFY(LEAFY)基因的表达,促进花萼和花瓣的分化[8-9]。另外在拟南芥ap1突变体研究中发现,AP1突变会引起花器官的异常发育,如花萼发育成叶片、花瓣缺失等[10-11]。人们已从多种观赏植物中分离得到了AP1基因,并且对多个物种的AP1基因进行了功能验证和分析。超表达AP1及其同源基因都能促进开花,在拟南芥中异源表达百合Lilium longiflorum,蝴蝶兰Phalaenopsis aphrodite以及山茶Camellia japonica的AP1同源基因都能引起早花现象[12-14]。同时,在拟南芥AP1突变体中异源表达百合,枇杷Eriobotrya japonica以及麻疯树Jatropha curcas中的AP1基因,都可以回补其突变体花器官缺陷的性状[12, 15-16]。桂花Osmanthus fragrans是中国十大传统名花之一,也是常见的园林绿化树种。按照开花习性不同,可分为秋桂和四季桂,秋桂仅在每年秋季开花,花序常为无总梗的聚伞花序[17];关于其成花过程以及花芽分化和发育报道较少。木本植物桂花成花的机制与草本植物拟南芥等有一定差异。为深入了解桂花的成花机制,有必要分离成花相关基因并展开相关研究。本研究以秋桂品种‘堰虹桂’Osmanthus fragrans ‘Yanhonggui’为材料,克隆桂花AP1基因(OfAP1),并对其序列进行生物信息学分析,同时运用荧光定量对其组织和时空表达进行分析,以明确OfAP1基因的基本特征和表达模式,为今后桂花的开花分子机理研究提供科学依据。
-
以盆栽‘堰虹桂’为材料,选取相同株龄且生长一致的植株。取桂花不同组织的样品,包括根、茎、叶、叶芽、花芽、盛开期的花朵;同时根据桂花花芽分化进程取叶芽、花序分化期、花萼花瓣分化期、雌雄蕊分化期以及花开放不同阶段圆珠期、铃梗期、初花期、盛花期和盛花末期的样品[18],用于基因克隆、组织特异性和时空表达分析。所有材料使用液氮冷冻并于-80 ℃保存备用。
RNA提取试剂盒(RNAprep pure Plant Kit)购自天根公司(北京)。反转录试剂盒、荧光定量试剂盒、胶回收试剂盒、Premix Taq酶、pMD-18T载体和大肠埃希菌Escherichia coli DH5α均购自Takara公司(大连),运用实时荧光定量PCR(Applied Biosystems,Foster City,美国)分析OfAP1基因的组织特异性及时空表达变化。
-
按照RNAprep pure Plant Kit试剂盒说明书提取各样品的总RNA,并参照Reverse Transcriptase M-MLV反转录酶说明书合成cDNA的第一链后,储存于-20 ℃备用。
-
利用前期转录组测序获得的AP1 Unigene序列,设计特异性引物AP1-F:5′-TAGAGTGAGAAAATGGGGAGA-3′;AP1-R:5′-AATACAATCCCTGGCTACTT-3′。以花芽分化期茎尖RNA反转录的cDNA为模板进行PCR扩增,PCR反应体系为:上下游引物各1 μL,cDNA 1 μL,Premix Taq DNA 10 μL和去离子水7 μL。PCR反应条件:95 ℃预变性5 min;然后进行35个循环,每个循环包括95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min;最后72 ℃保温10 min,4 ℃保存。PCR反应产物经质量分数为1%的琼脂糖凝胶电泳检测后回收、纯化,与pMD18-T载体连接,转化大肠埃希菌DH5α感受态细胞,蓝白斑筛选阳性克隆,经PCR鉴定后送生工生物工程(上海)股份有限公司测序。
-
对获得的基因序列进行生物信息学分析,其中编码区分析采用美国国家生物技术信息中心(NCBI)在线网站ORFFinder(),编码蛋白质的保守区段分析采用NCBI Conserved Domain Search软件(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),编码区蛋白质特征分析采用在线软件ProtParam(http://web.expasy.org/protparam/)和TMHMM Server v2.0()。多序列比对采用软件DNAman,系统进化树构建采用Clustal X+MEGA 7.0软件并选择邻位连接法(neighbor joining method)用Bootstrap法检验1 000次。
-
按照SYBR® Premix Ex Taq TM Ⅱ(Tli RNaseH Plus)试剂盒的说明书,检测桂花OfAP1基因不同组织以及不同时期的表达情况。OfAP1定量引物为qAP1-F:5′-GCAGAAGTGGCTTTGATTTG-3′;qAP1-R:5′-GTTTGCTGGTGACTGAGGTT-3′。同时以OfACT为内参qACT-F:5′-CCCAAGGCAAACAGAGAAAAAAT-3′;qACT-R:5′-ACCCCATCACCAGAATCAAGAA-3′[19]。qRT-PCR反应体系:2×SYBR Premix Ex TaqⅡ(Tli RNaseH Plus)10 μL,上下游定量引物(10 μmol·L-1)各0.8 μL,cDNA 2 μL,50×ROX Reference Dye 0.4 μL,双蒸水补齐至20 μL。qRT-PCR程序如下:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min。3次生物学重复。反应后根据熔解曲线分析qRT-PCR产物的特异性,并采用2-△△CT法计算相对表达量。
-
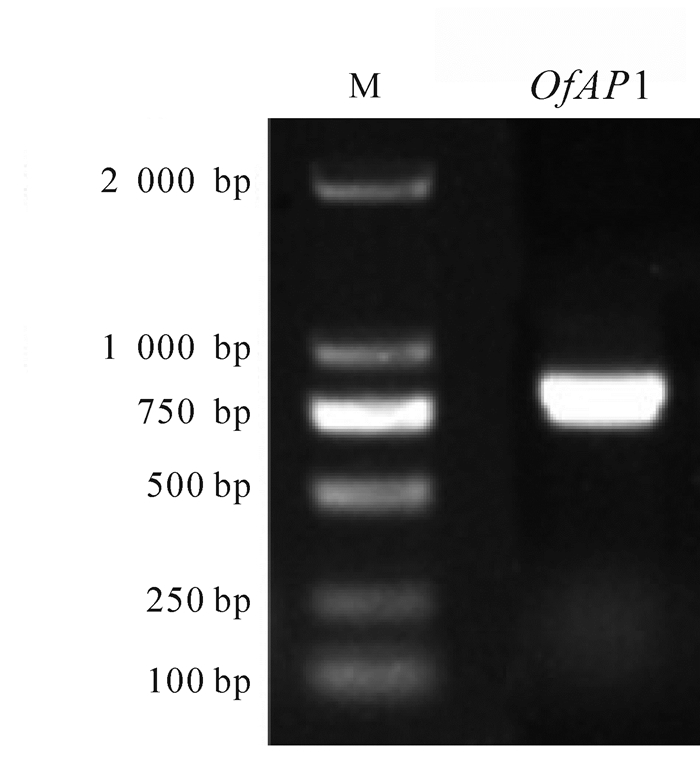
以‘堰虹桂’花芽分化期cDNA为模板,扩增得到约750 bp片段(图 1),其开放阅读框长为720 bp,编码239个氨基酸(图 2)。该基因片段具有MADS-box基因的保守区,在NCBI上进行BLASTX在线分析,发现该序列与芝麻Sesamum indicum(AIS82596.1),葡萄Vitis vinifera(ACZ26528.1)和油橄榄Olea europaea(XP_022878350.1)等中AP1基因的同源性最高,命名为OfAP1,并在GenBank注册,登录号为MH593222。
-
OfAP1蛋白氨基酸序列运用NCBI在线CDD(Conserved Domain Datebase)分析发现,OfAP1具有典型的MEF2_like MADS结构域和K-box结构域,MADS结构域位于N端第2~74位氨基酸,K-box结构域位于第90~168位氨基酸(图 2)。OfAP1蛋白的分子式为C1199H1941N341O368S16,其相对分子质量为27 534.62,理论等电点为8.4。负电荷残基总数(天冬氨酸Asp+谷氨酸Glu)为32,正电荷残基总数(精氨酸Arg+赖氨酸Lys)为35。在组成OfAP1蛋白的20种氨基酸中,亮氨酸(Leu)所占比例最高,为11.7%,其次为谷氨酸(Glu),占10.0%,色氨酸(Trp)所占比例最低,为0.8%。预测亲水性指数(GRAVY)为-0.665,表明OfAP1具有较好的亲水性。
采用GOR4软件对OfAP1蛋白二级结构预测发现,OfAP1蛋白二级结构由α螺旋、伸展链和无规则卷曲组成,其中,α螺旋(Hh)占56.49%,β折叠(Ee)占10.46%,无规则卷曲(Cc)占33.05%。同时采用Phyre 2在线工具对OfAP1蛋白三级结构进行预测,OfAP1有3个典型的α螺旋和2个β折叠(图 3)。NetPhos 2.0 Server预测结果表明:OfAP1蛋白序列中存在11个潜在的磷酸化位点,包括6个Ser位点(22Ser,61Ser,74Ser,87Ser,110Ser,121Ser,135Ser,150Ser,216Ser)和1个Thr位点(220Thr)。NetNGlyc 1.0 Server预测结果表明:OfAP1蛋白存在1个潜在的N-糖基化位点,90Asn。
-
为了分析OfAP1基因与其他物种中AP1基因的系统进化关系,采用MEGA 7.0软件构建系统进化树(图 4)。单子叶植物(如短柄草Brachypodium distachyon,水稻Oryza sativa,玉米Zea mays)和双子叶植物可以明显地分为2个大类。桂花OfAP1与芝麻,豇豆Vigna unguiculata,枇杷,印加果Plukenetia volubilis,麻疯树,葡萄,芍药Paeonia lactiflora,柑橘Citrus sinensis以及拟南芥中的AP1基因聚在一起,说明这些基因亲缘关系较近且功能具有一定的相似性。
-
为明确OfAP1基因在桂花不同组织以及花芽分化不同时期的表达情况,对不同组织和器官(根、茎、叶、叶芽、花芽以及花)及不同发育时期的样品进行荧光定量RT-PCR分析。结果表明:OfAP1基因在花芽中的表达量最高,其次为叶,在茎中的表达比较弱,在根中几乎不表达(图 5)。同时,与叶芽时期相比,OfAP1基因在分化初期(即花原基的形成以及花萼花瓣分化期)表达量最高,随后呈下降趋势(图 6),到花开放阶段表达量均较低。
-
AP1及其同源基因已在多个物种中克隆得到,但桂花中的AP1至今没有相关报道。本研究克隆得到OfAP1基因,发现其与芝麻、葡萄以及油橄榄等AP1基因高度相似,OfAP1氨基酸序列含有高度保守的MEF2_like MADS结构域和次级保守的K-box结构域以及可变C,MADS结构域具有结合DNA、蛋白质二聚化以及与其他因子结合的功能;K-box结构域的蛋白二级结构为3个α螺旋,具有介导蛋白-蛋白之间的相互作用[20]。AP1基因C末端的变化较大,但是不同类的MADS-box基因常含有一些保守的基序(motif),这些基序在蛋白复合体的形成和转录激活中起重要作用[21]。
开花是植物重要的发育阶段,是多条基因网络共同调控的复杂过程[2]。AP1基因是植物特有的MIKCC-Type MADS-box基因,既参与花分生组织的形成,又是花器官形成的重要基因,在植物成花中起关键调控作用[3]。根据在模式植物拟南芥中的研究,AP1是花分生组织形成的标志物,成花整合基因FT和SOC1能够激活其表达并使其参与对成花抑制基因TFL1基因的拮抗,促进花分生组织的形成。在花器官发育中,AP1一方面通过促进AP3和PI的表达诱导花瓣和雄蕊的发育,另一方面受到AG基因的抑制作用控制萼片和花瓣的发育[22]。在百合中AP1的同源基因LMADS5/6转基因拟南芥后发现其能够造成早花,并且花器官中萼片变成心皮状,花瓣变成雄蕊状结构[12];同样地,在洋桔梗Eustoma grandiflorum中超表达AP1发现:转基因植株花期提前,部分花瓣出现雄蕊类似的结构,进一步突变体互补实验表明:AP1基因对花瓣形成,尤其是第2轮花被片的发育有重要作用[23]。在建兰Cymbidium ensifolium,芍药以及菊花Chrysanthemum morifolium等中,AP1基因在花芽中的表达量显著高于其他器官组织,尤其在花芽分化的初期阶段[24-26]。在桂花中,OfAP1在花芽中有较高的表达,其在花芽分化初期表达量显著上调,尤其是成花转变以及花瓣、花萼分化时期,而当完成花萼花瓣分化后,表达量则显著下调。这充分说明桂花OfAP1基因参与了桂花成花转变和花瓣、花萼等器官分化,但其具体作用机制还有待进一步通过转化实验加以验证。
cDNA cloning and expression analysis of OfAP1 in Osmanthus fragrans
-
摘要: AP1(APETALA1)基因在植物花分生组织及花器官形成过程中发挥着重要作用。以桂花品种‘堰虹桂’Osmanthus fragrans‘Yanhonggui’为材料,根据前期获得的转录组数据中AP1的序列设计引物,利用PCR技术,克隆得到约750 bp桂花AP1 cDNA序列,即OfAP1基因,其中开放阅读框长为720 bp(注册号为MH593222)。氨基酸序列比对发现,与其他物种的AP1基因的同源性高达69%~88%。荧光定量PCR结果表明:在不同组织中,桂花的OfAP1基因在花芽中的表达量显著高于其他组织,根中几乎不表达;在花芽分化过程中,OfAP1在成花转变及花芽分化初期(花瓣、花萼分化期)表达量较高,随后呈下降趋势。这说明OfAP1具有组织表达特异性,同时在桂花成花转变、花芽分化和发育中有重要作用。Abstract: Osmanthus fragrans is one of the top ten traditional flowers and a common landscaping tree in China. To better understand the function of AP1 in regulation of Osmanthus flowering, the OfAP1 gene was cloned from the Osmanthus cultivator 'Yanhonggui', and its function was analyzed through bioinformatics method and real time PCR. Our results showed that the cDNA length of OfAP1 was 750 bp (GenBank accession No. MH593222), in which the Open Reading Frame length was 720 bp, the amino acid was 239. The OfAP1 sequence alignment revealed high homology with other species ranging from 69% to 88%. Additionally, expression profiles showed that the expression of OfAP1 in flower buds was much higher than that in other tissues (root, stem, leaf, leaf bud and flower), and the expression was barely expressed in roots. During the development of flower bud, the OfAP1 showed higher expression in S1 (calyx and petal differentiation) stage than other stages. Thus, these results suggested that OfAP1 had a tissue specific expression and played an important role in flowering transformation, flower bud differentiation, and development of O. fragrans. And the results of OfAP1 can lay a foundation for further study of the molecular mechanism of O. fragrans.
-
Key words:
- forest tree breeding /
- Osmanthus fragrans /
- AP1 /
- gene cloning /
- expression analysis /
- flower bud development
-
[1] 周玉萍, 陈琼华, 黄小玲, 等.拟南芥开花时间调控的分子基础[J].植物学报, 2014, 49(4):469-482. ZHOU Yuping, CHEN Qionghua, HUANG Xiaoling, et al. Molecular basis of flowering time regulation in Arabidopsis[J]. Chin Bull Bot, 2014, 49(4):469-482. [2] FORNARA F, de MONTAIGU A, COUPLAND G. SnapShot:control of flowering in Arabidopsis[J]. Cell, 2010, 141(3):550-550. [3] HUANG T, BÖHLENIUS H, ERIKSSON S, et al. The mRNA of the Arabidopsis gene FT moves from leaf to shoot apex and induces flowering[J]. Science, 2005, 309(5741):1694-1696. [4] LIU Lu, ZHU Yang, SHEN Lisha, et al. Emerging insights into florigen transport[J]. Curr Opin Plant Biol, 2013, 16(5):607-613. [5] MANDEL M A, YANOFSKY M F. A gene triggering flower formation in Arabidopsis[J]. Nature, 1995, 377(6549):522. [6] PUTTERILL J, LAURIE R, MACKNIGHT R. It's time to flower:the genetic control of flowering time[J]. Bioessays, 2004, 26(4):363-373. [7] MANDEL M A, GUSTAFSON-BROWN C, SAVIDGE B, et al. Molecular characterization of the Arabidopsis floral homeotic gene APETALA1[J]. Nature, 1992, 360(6401):273. [8] 戚晓利, 卢孟柱.拟南芥APETALA1基因在花发育中的网络调控及其生物学功能[J].中国农学通报, 2011, 27(8):103-107. QI Xiaoli, LU Mengzhu. Regulation network and biological roles of APETALA1 of Arabidopsis thaliana in flower development[J]. Chin Agric Sci Bull, 2011, 27(8):103-107. [9] KAUFMANN K, WELLMER F, MUINO J M, et al. Orchestration of floral initiation by APETALA1[J]. Science, 2010, 328(5974):85-89. [10] IRISH V F, SUSSEX I M. Function of the APETALA1 gene during Arabidopsis floral development[J]. Plant Cell, 1990, 2(8):741-753. [11] BOWMAN J L, ALVAREZ J, WEIGEL D, et al. Control of flower development in Arabidopsis thaliana by APETALA1 and interacting genes[J]. Development, 1993, 119(3):721-743. [12] CHEN Mingkun, LIN Ichun, YANG Changhsien. Functional analysis of three lily (Lilium longiflorum) APETALA1-like MADS box genes in regulating floral transition and formation[J]. Plant Cell Physiol, 2008, 49(5):704-717. [13] LIU Yuexue, KONG Jin, LI Tianzhong, et al. Isolation and characterization of an APETALA1-like gene from pear (Pyrus pyrifolia)[J]. Plant Mol Biol Rep, 2013, 31(4):1031-1039. [14] SUN Yingkun, FAN Zhengqi, LI Xinlei, et al. The APETALA1 and FRUITFUL homologs in Camellia japonica and their roles in double flower domestication[J]. Mol Breed, 2014, 33(4):821-834. [15] LIU Yuexue, SONG Huwei, LIU Zongli, et al. Molecular characterization of loquat EjAP1 gene in relation to flowering[J]. Plant Growth Regulation, 2013, 70(3):287-296. [16] TANG Mingyong, TAO Yanbin, XU Zengfu. Ectopic expression of Jatropha curcas APETALA1(JcAP1) caused early flowering in Arabidopsis, but not in Jatropha[J]. Peer J, 2016, 4:e1969. doi:10.7717/peerj.1969. [17] 向其柏, 刘玉莲.中国桂花品种图志[M].浙江:浙江科学技术出版社, 2008:56. [18] 王英, 张超, 付建新, 等.桂花花芽分化和花开放研究进展[J].浙江农林大学学报, 2016, 33(2):340-347. WANG Ying, ZHANG Chao, FU Jianxin, et al. Progress on flower bud differentiation and flower opening in Osmanthus fragrans[J]. J Zhejiang A & F Univ, 2016, 33(2):340-347. [19] 付建新, 张超, 王艺光, 等.桂花组织基因表达中荧光定量PCR内参基因的筛选[J].浙江农林大学学报, 2016, 33(5):727-733. FU Jianxin, ZHANG Chao, WANG Yiguang, et al. Reference gene selection for quantitative real-time polymerase chain reaction (qRT-PCR) normalization in the gene expression of sweet osmanthus tissues[J]. J Zhejiang A & F Univ, 2016, 33(5):727-733. [20] THEISSEN G, BECKER A, DI ROSA A, et al. A short history of MADS-box genes in plants[J]. Plant Mol Biol, 2000, 42(1):115-149. [21] NG M, YANOFSKY M F. Function and evolution of the plant MADS-box gene family[J]. Nat Rev Genet, 2001, 2(3):186-195. [22] LIU Chang, XI Wanyan, SHEN Lisha, et al. Regulation of floral patterning by flowering time genes[J]. Dev Cell, 2009, 16(5):711-722. [23] CHUANG Tienhsin, LI Kunhung, LI Peifang, et al. Functional analysis of an APETALA1-like MADS box gene from Eustoma grandiflorum in regulating floral transition and formation[J]. Plant Biotechnol Rep, 2018, 12(2):1-11. [24] 吴菁华, 吴少华, 杨超, 等.建兰AP1基因的克隆、表达及其与MADS-box转录因子相互作用的分析[J].园艺学报, 2013, 40(10):1935-1942. WU Jinghua, WU Shaohua, YANG Chao, et al. Cloning and expression analysis on AP1 homologous gene from Cymbidium ensifolium and interaction analysis between AP1 and MADS-box transcription factors[J]. Acta Hortic Sin, 2013, 40(10):1935-1942. [25] 吴彦庆, 葛金涛, 陶俊.芍药花分生组织决定基因(AP1)克隆、序列分析及其在不同发育时期花瓣中的表达变化规律[J].农业生物技术学报, 2015, 23(12):1559-1567. WU Yanqing, GE Jintao, TAO Jun. cDNA cloning, sequence analysis and tissue expression detection of APETALA1 gene (AP1) in Paeonia lactiflora Pall. petals of different development stages[J]. Chin J Agric Biotechol, 2015, 23(12):1559-1567. [26] 梁芳, 黄萍, 袁秀云, 等.菊花CmAP1基因克隆及其植物表达载体构建[J].南方农业学报, 2017, 48(6):952-959. LIANG Fang, HUANG Ping, YUAN Xiuyun, et al. Cloning of CmAP1 gene from Chrysanthemum morifolium and establishment of its plant expression vector[J]. J South Agric, 2017, 48(6):952-959. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.2019.04.005







 下载:
下载: