-
光作为重要的环境因子控制着植物一系列生长发育过程,其调控作用是通过光受体介导的信号传导的途径来完成的[1]。目前,在拟南芥Arabidopsis thaliana中共发现了3种CRY(CRYPTOCHROME):CRY1,CRY2和CRY3,其中CRY1和CRY2通过负调控光形态建成的负调控因子COP1(COnstitutive Photomorphogenic 1)来实现其调控光形态建成的功能[2-3]。COP1作为一种重要的调控因子,广泛存在于动植物体内[4]。在植物中,COP1作为一种E3泛素连接酶,具有E3泛素连接酶活性使转录调控因子LAF1,HY5,HFR1等泛素化,并调控其降解,从而起到抑制光信号调节的作用,进而在光信号转导及植物光形态建成中起重要作用[5-7]。COP1在细胞核内充当一个特异转录因子的总的调节因子[8],通过与其互作因子相互结合而使这些光形态建成发育的正向调节子失活[9-11]。CO是光周期开花转换途径中一个最重要的基因[12],通过激活下游基因FT,SOC1等表达,使植物由营养生长向生殖发育转变[13-14]。COP1与CO存在直接的蛋白相互作用,并在体外作为泛素E3连接酶使CO蛋白发生泛素化,在植物体内促进CO蛋白的降解,而CRY介导的信号可能通过负调控COP1对CO蛋白的降解作用,使CO蛋白稳定并发生积累,从而激活下游基因FT的表达,促进开花[15-17]。山核桃Carya cathayensis是中国著名的特色干果,因其种仁中的不饱和脂肪酸成分对人体具有滋补、健脑等保健效果,得到了广大消费者的青睐[18]。成花是决定山核桃产量的关键因素,而木本植物需要较长时间的营养生长(童期)才转向生殖生长的[19]。在广泛的研究中,已通过利用分子生物学、遗传学等技术研究花的发育,极大地促进了成花机制的研究[20-21]。如何运用分子机制促进木本植物山核桃提早开花,为将来进一步阐释山核桃成花的分子机制提供了重要的基因平台,又为促进山核桃早花、早实、丰产等的实际生产应用推广提供必需的理论支持。山核桃全基因组测序工作的完成(数据未发表)得到CcCOP1 E3连接酶片段,利用cDNA末端快速扩增技术(RACE)技术克隆获得了CcCOP1 E3连接酶的全长;利用生物信息学手段分析了它的蛋白特性和不同物种的同源基因之间的亲缘关系;利用实时荧光定量聚合酶链式反应(real-time PCR)技术进一步分析了在CcCOP1 E3连接酶雌雄花发育过程中的表达变化。该研究旨在为研究该基因的生物学功能,为进一步阐明山核桃成花调控的分子机制打下良好的基础。
HTML
-
受试植物材料采自浙江省临安市太湖源镇的1株山核桃树,树龄约为30 a。于2013年2月24日、2月28日、3月3日、3月8日、3月12日、3月14日、3月16日、3月18日、3月20日、3月23日、3月26日、3月31日依次采取短果枝顶芽(前期为营养芽状态,后期为雌花芽状态),共采样12次。于2013年4月29日、5月6日、5月11日、5月14日、5月17日、5月21日、5月26日、5月29日、6月1日、6月4日、6月16日、6月23日依次采集短果枝的侧芽(前期为营养芽状态,后期为雄花芽状态),共采样16次。样品采集后立即放入液氮,带回实验室-70 ℃冰箱保存,用于核糖核酸(RNA)提取。反转录试剂盒、胶回收试剂盒、氨苄青霉素(amp)、限制性内切酶、rTaq DNA聚合酶及PMD18-T载体分别购自生工生物工程(上海)股份有限公司和宝生物工程大连有限公司。
-
采用十六烷基三甲基溴化铵(CTAB)法提取山核桃样品的总RNA。以总RNA为模板,使用TaKaRa的PrimeScriptTM RT reagent Kit perfect Real Time试剂盒反转录获得cDNA用于反转录-聚合酶链式反应(RT-PCR)。
-
CcCOP1 E3连接酶片段取自山核桃成花转录组454测序拼接获得的contig数据。分别设计3′RACE和5′RACE引物(3′RACE引物1:5′-AGCGTGCGTGGAGTGTTGATTTT-3′,3′RACE引物2:5′-CCTGGATCTAGCAATTACATTGCGG-3′,5′RACE引物1:5′-CGCACGCTTTTCATGCTCTTCATAC-3′,5′RACE引物2:5′-ATAGCCGCAGTGTGCTGTCTGTAGA-3′按照Clontech公司RACE试剂盒的说明书,将提好的RNA进行已获得的cDNA为模板分别进行3′RACE和5′RACE反应,RACE反应体系为50.0 μL,内含1×PCR缓冲液(buffer),氯化镁(MgCl2)1.5 mmol·L-1,三磷酸脱氧核糖核苷酸(dNTPs)0.2 mmol·L-1,10×通用引物组合(UPM)1.0 μmol·L-1,基因特异引物0.2 μmol·L-1,Taq酶(33.34 nkat),模板约100 ng。RACE扩增的条件为:94 ℃ 5 min预变性后,94 ℃ 1 min,57 ℃ 1 min 15 s,72 ℃ 2 min 45 s,共40个循环,最后72 ℃延伸10 min。用DNA回收试剂盒回收目标片段,将目标片段连接到pMD-18T simple载体中进行T/A克隆,最后将带有目标片段的质粒回收后交付生工生物工程(上海)股份有限公司进行测序。测得的序列通过美国生物技术信息中心(NCBI)的BLAST进行同源搜索和功能域推导;利用ExPASy数据库中的ProParam在线工具分析其编码蛋白的理化性质和亲水性分析;用Clustalx 1.83软件进行蛋白序列比对,用MEGA 3.1软件的邻接法(neighbor-joining,NJ)构建系统进化树。
-
根据基因的保守序列设计real-time RT PCR引物(正向引物:ATGAAGAGCATGAAAAGCGTGCGTG;反向引物GGGTAAACGCTTTGTTCTCC)。Actin作为内参(Actin正向引物:TAATGTGTTGGACTCTGGTGATGG;反向引物:ACATATGAAACAGCTTTCCTGTGCC),在Bio-RAD荧光定量PCR仪上扩增。用Excel 2003分析试验数据。
1.1. 植物材料
1.2. 研究方法
1.2.1. 山核桃总RNA的提取、反转录合成cDNA
1.2.2. CcCOP1 E3连接酶的克隆与序列分析
1.2.3. 实时荧光定量反转录聚合酶链式反应(real-time RT PCR)
-
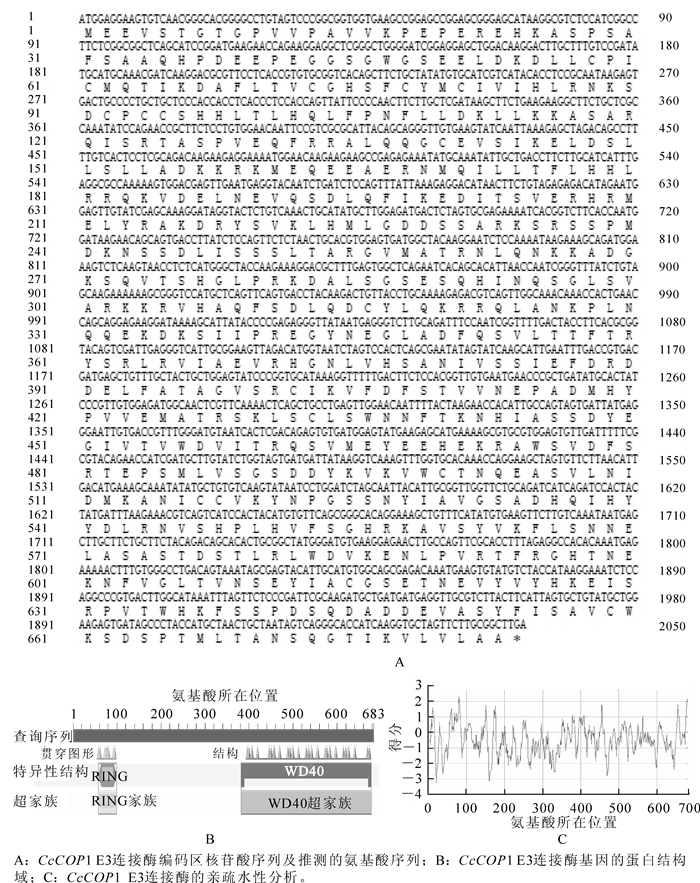
通过5′RACE和3′RACE的方法分别获得长度约为1 300 bp的5′末端序列和700 bp的3′末端序列。将最终扩增片段进行测序分析,获得了2 331 bp的全长cDNA序列(图 1A),命名为Carya cathayensis E3 ubiquitin protein ligase COP1-like,以下统称为CcCOP1 E3连接酶,Genbank登录号为KF983530。编码683个氨基酸残基(图 1B),分子量为77.113 kD,PI为7.0。它由2个特殊的结构域组成即环形锌指结合域和WD-40重复序列。预测这个基因编码的蛋白质都有较强的亲水性,而且亲水性氨基酸主要集中在氨基端;可能都没有跨膜结构域(图 1C)。
-
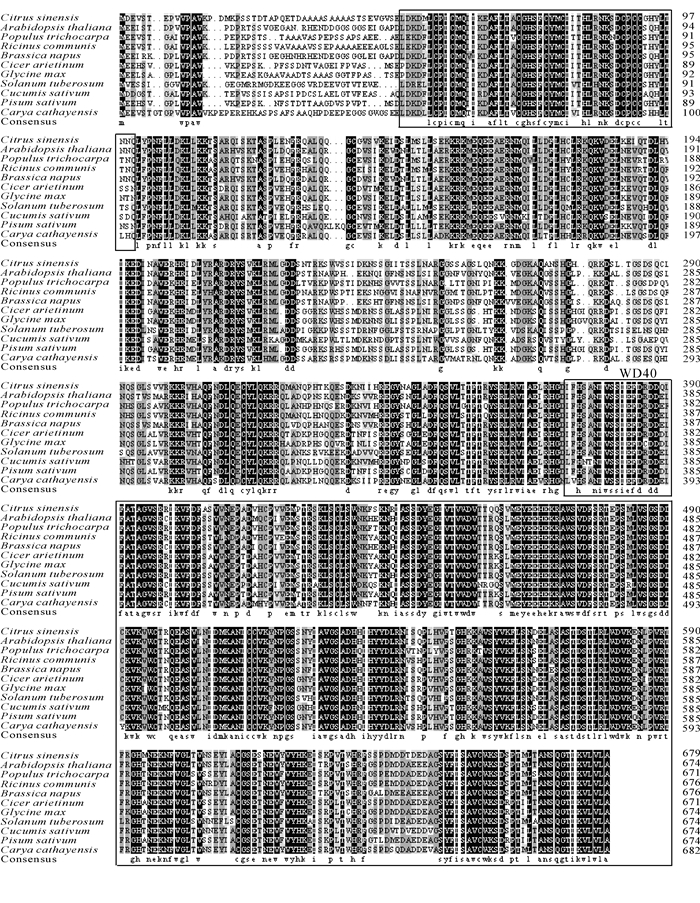
从NCBI网站上搜寻并下载其他物种的CcCOP1 E3连接酶的序列。序列比对发现,山核桃CcCOP1 E3连接酶与毛果杨Populus trichocarpa等的COP1 E3连接酶同源基因相似度较高,总体高达77.80%,其二级结构由2个特殊的结构域组成,即N-端的环形锌指结合域(ring finger zinc-binding domain,RING),C-端的WD-40重复序列(WD-40 repeats,图 2)。
为了了解CcCOP1 E3连接酶与其他植物COP1 E3连接酶的亲缘关系,利用MEGA3.1软件,将不同植物的COP1 E3连接酶构建系统进化树(图 3)。根据进化树聚类分析发现,在已公布的同源基因中,CcCOP1 E3连接酶与蓖麻Ricinus communis等的亲缘关系最近,而蓖麻属于双子叶植物,与乌拉尔图小麦Triticum urartu和二穗短柄草Brachypodium distachyon等的亲缘关系相对较远,乌拉尔图小麦和二穗短柄草属于单子叶植物。表明该基因的进化过程存在种属差异性,与双子叶植物亲缘关系更为接近。
-
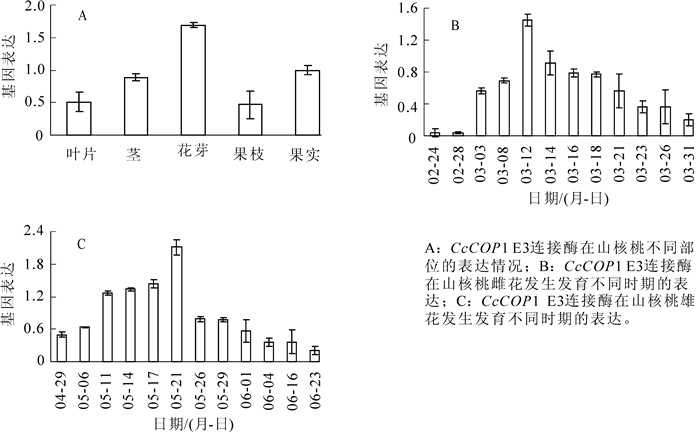
总体上,CcCOP1 E3基因在山核桃的叶片、茎、花芽、果枝和果实中均有表达,如(图 4A),尤其在花芽中的表达量为最高。CcCOP1 E3连接酶在山核桃雌花发育过程中3月12日达到高峰,之后表达量逐渐下降(图 4B);在雄花芽中的表达量从5月11明显升高,到5月21日到达高峰,之后的表达量显著下降(图 4C)。总体上,CcCOP1 E3连接酶在山核桃雌花和雄花中的表达均呈现“上升—高峰—下降”的趋势。
2.1. CcCOP1 E3连接酶全长的克隆、测序和结构分析
2.2. CcCOP1 E3连接酶序列的同源性分析
2.3. 山核桃雌雄花发生发育过程CcCOP1 E3连接酶的转录表达
-
对CcCOP1 E3连接酶功能域分析结果表明:由2个特殊的结构域组成即环形锌指结合域和WD-40重复序列。预测这个基因编码的蛋白质都有较强的亲水性,而且亲水性氨基酸主要集中在氨基端;可能都没有跨膜结构域。在系统进化上,山核桃CcCOP1 E3连接酶与毛果杨Populus trichocarpa等的CcCOP1 E3连接酶同源基因相似度较高。进化树聚类分析发现,在已公布的同源基因中,CcCOP1 E3连接酶与蓖麻等的亲缘关系最近,与乌拉尔图小麦和穗短柄草等的亲缘关系相对较远,说明该基因与双子叶植物亲缘关系更为接近。
根据实时荧光定量反转录聚合酶链式反应(real-time RT PCR)分析显示,在山核桃的叶片、茎、花芽、果枝、果实中均有表达,尤其在花芽中的表达量为最高,推测该基因可能参与植物花的发育,这还有待进一步功能分析研究。具体地,CcCOP1 E3连接酶在山核桃雌花发育过程中3月12日达到高峰。黄有军等[20]从超微结构、显微结构和外部形态变化多个层次上对雌花发育过程的研究,总结出山核桃雌花花芽分化规律为:雌花发育过程从3月下旬开始到5月中旬基本完成,此时正是雌花芽开始分化的时期,进而促进开花。我们推测再花分化之前,基因的表达量上升,促进花分生组织的分化,进而促进开花。在雄花芽中的表达量到5月21日到达高峰,这与山核桃雄花生长习性相关,雄花芽分化自5月中下旬至7月中旬完成花序原基分化,7月中旬至次年3月中旬为休眠期,次年3月中下旬继续分化,花序伸长,完成雄配子体的发育,花粉粒成熟散粉后雄花序脱落[21]。该基因的表达量在雄花芽分化之前呈上升趋势,到5月中旬表达量最高,之后表达量显著降低,进而促进雄花芽的分化。纵观山核桃整个花发育过程,总体上,CcCOP1 E3连接酶在山核桃雌花和雄花中的表达均呈现“上升—高峰—下降”的趋势分析,在雌花和雄花的分化过程中,担当重要角色。
结合本课题组对山核桃开花相关基因CcCOP1 E3连接酶的研究,将更有利于对于调控山核桃花芽分化的分子机制的研究,促进雌花的分化及控制雄花的分化对于提高山核桃的产量有着重要的价值。
对于植物COP1进行了深入研究,其结构、功能及其发挥作用时的分子机制得到了更多的阐释, 但是植物COP1蛋白仅是一个大的光信号传递复合物中的一个组分, 而高等植物的光信号传递更加是一个复杂的网络, 光形态建成的调节机制仍然尚未完全明了。另外,其他植物中也存在COP1蛋白, 但其结构和功能以及作用机制是否与山核桃完全一致呢?这些有待于进一步深入研究。













 DownLoad:
DownLoad: