-
叶绿体为植物细胞质体之一,是植物进行光合作用的特有细胞器,内部具有独立而完整的基因组 [ 1] 。植物叶绿体基因组一般由1个大单拷贝区(LSC)、1个小单拷贝区(SSC)、2个反向重复区(IRs)组成的环状四分体结构 [ 2] ,序列长度约120~160 kb [ 3] ,含有大量可以自我调节的蛋白编码基因、rRNA编码基因以及tRNA编码基因 [ 4] ,遵循单亲遗传规律,结构简单且总体保守,进化速率也相对适中。随着基因测序技术的快速发展,基于植物叶绿体基因组的研究已被广泛应用于系统进化 [ 5] 、物种鉴别 [ 6] 、转基因 [ 7] 以及核质协作 [ 8] 等领域。
黄连木属 Pistacia是漆树科Anacardiaceae的关键演化分支,全球约10个物种 [ 9] ,中国主要有中国黄连木 P. chinensis、清香木 P. weinmaniifolia、阿月浑子 P. vera [ 10] 。中国黄连木分布于南北干旱丘陵区,成熟种子富含油脂(含油率>40%) [ 11] ,是一种潜在的生物能源树种 [ 12] ;清香木生于云南、四川等喀斯特地貌区,叶片富含抗菌活性的挥发性萜烯类化合物 [ 13] ,是传统药用植物;阿月浑子原产于中亚,在中国新疆地区已形成独特的栽培种群,是世界第五大坚果作物 [ 14] 。然而,生长于中国的黄连木属植物受不同地区气候变化和人为干扰的影响,导致部分野生种群萎缩,亟需通过叶绿体基因组研究指导种质资源保护与利用。因此,本研究拟选取中国黄连木、清香木、阿月浑子这3种黄连木属植物,解析叶绿体基因组的结构分化,为黄连木属植物的系统分类和进化历史提供科学依据。
-
从美国国家生物技术信息中心(NCBI)数据库(http://www.ncbi.nlm.nih.gov/),搜索并下载已公布的中国黄连木(MK738124)、清香木(MF630953)、阿月浑子(MN551174)叶绿体基因组序列信息展开分析。
-
利用Geneious Prime软件统计黄连木属叶绿体基因组的各项参数,包括基因组大小、各区序列长度、GC含量、基因总数、蛋白编码序列(CDS)数量、核糖体RNA (rRNA)数量、转运RNA (tRNA)数量,并在Chloroplot在线程序 [ 15] (http://irscope.shinyapps.io/chloroplot/)绘制叶绿体基因组图谱。
-
利用MISA在线工具 [ 16] (http://webblast.ipk-gatersleben.de/misa/)计算黄连木属叶绿体基因组中的简单重复序列数量(simple sequence repeat,SSR),最小重复单元设置:单核苷酸重复为10,二核苷酸重复为5,三核苷酸重复为4,四核苷酸重复为3,五核苷酸重复为3,六核苷酸重复为3,重复序列之间的最小间隔为100 bp。利用REPuter在线工具 [ 17] 检测不同的散在重复序列类型,包括回文重复(P)、互补重复(C)、正向重复(F)和反向重复(R),参数设置:最大重复距离为5 000 bp,最小重复长度为30 bp,海明距离为3。
-
以中国黄连木为参考,利用mVISTA在线工具 [ 18] (http://genome.lbl.gov/vista/index.shtm)选择Shuffle-LAGAN模式对黄连木属叶绿体基因组开展结构可视化差异分析;利用CPJSdraw软件 [ 19] 完成四分体边界差异分析。
-
利用DnaSP V6软件 [ 20] 检测黄连木属叶绿体基因组的核苷酸多态性(Pi),参数设置:窗口长度为600 bp,序列步长为200 bp。
-
选择盐肤木属 Rhus植物旁遮普麸杨 R. punjabensis (MT230555)、盐肤木 R. chinensis (OP326720)为外类群,另收集其他国外黄连木属叶绿体基因组信息,包括大西洋黄连木 P. atlantica (PP101613)、埃及黄连木 P. khinjuk (PP101614),利用MAFFT V7在线工具 [ 21] (http://mafft.cbrc.jp/alignment/server/)完成叶绿体全基因组对比,再通过MEGA 7.0软件 [ 22] 以邻接法(neighbor-joining)构建系统进化树。
-
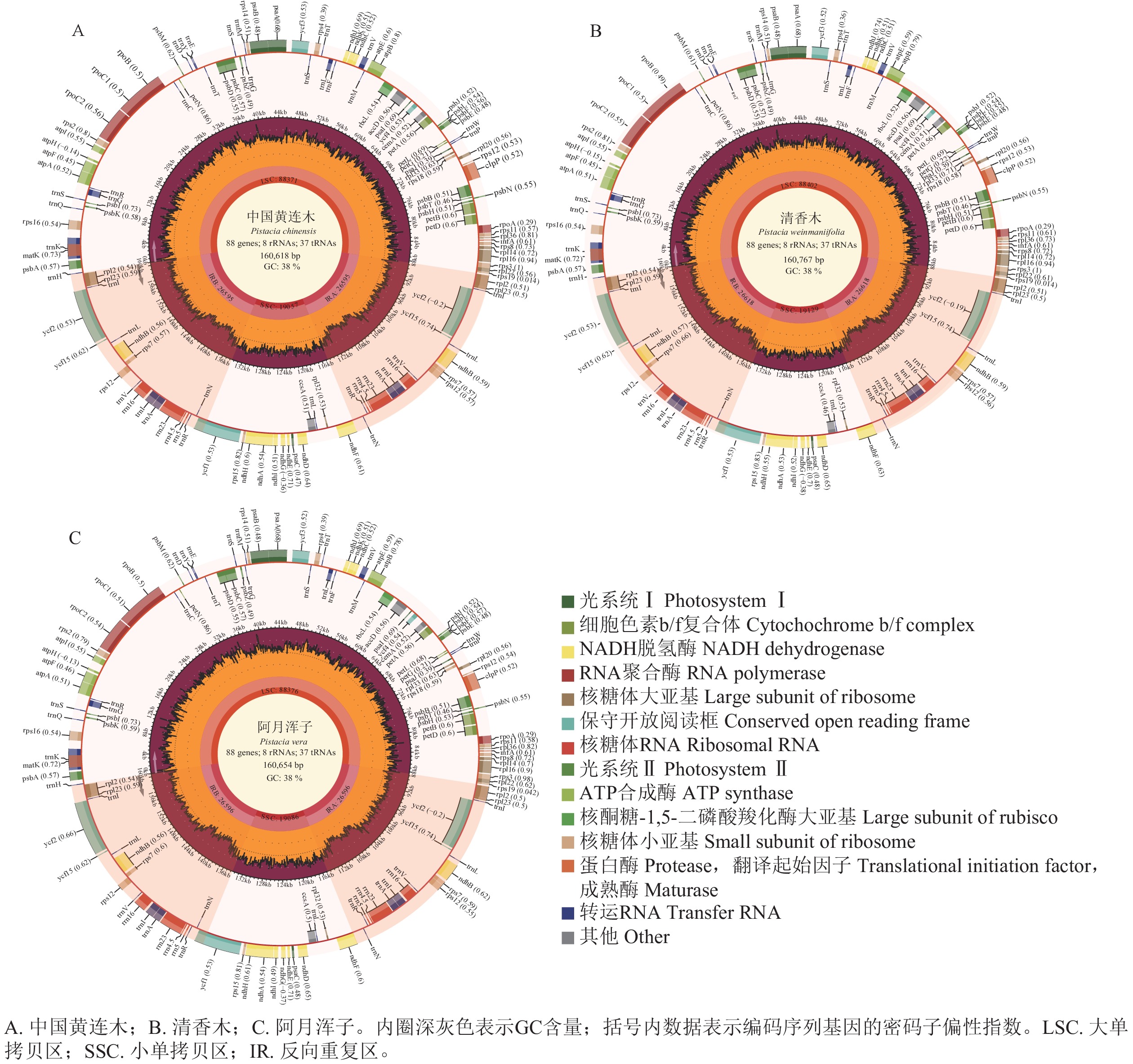
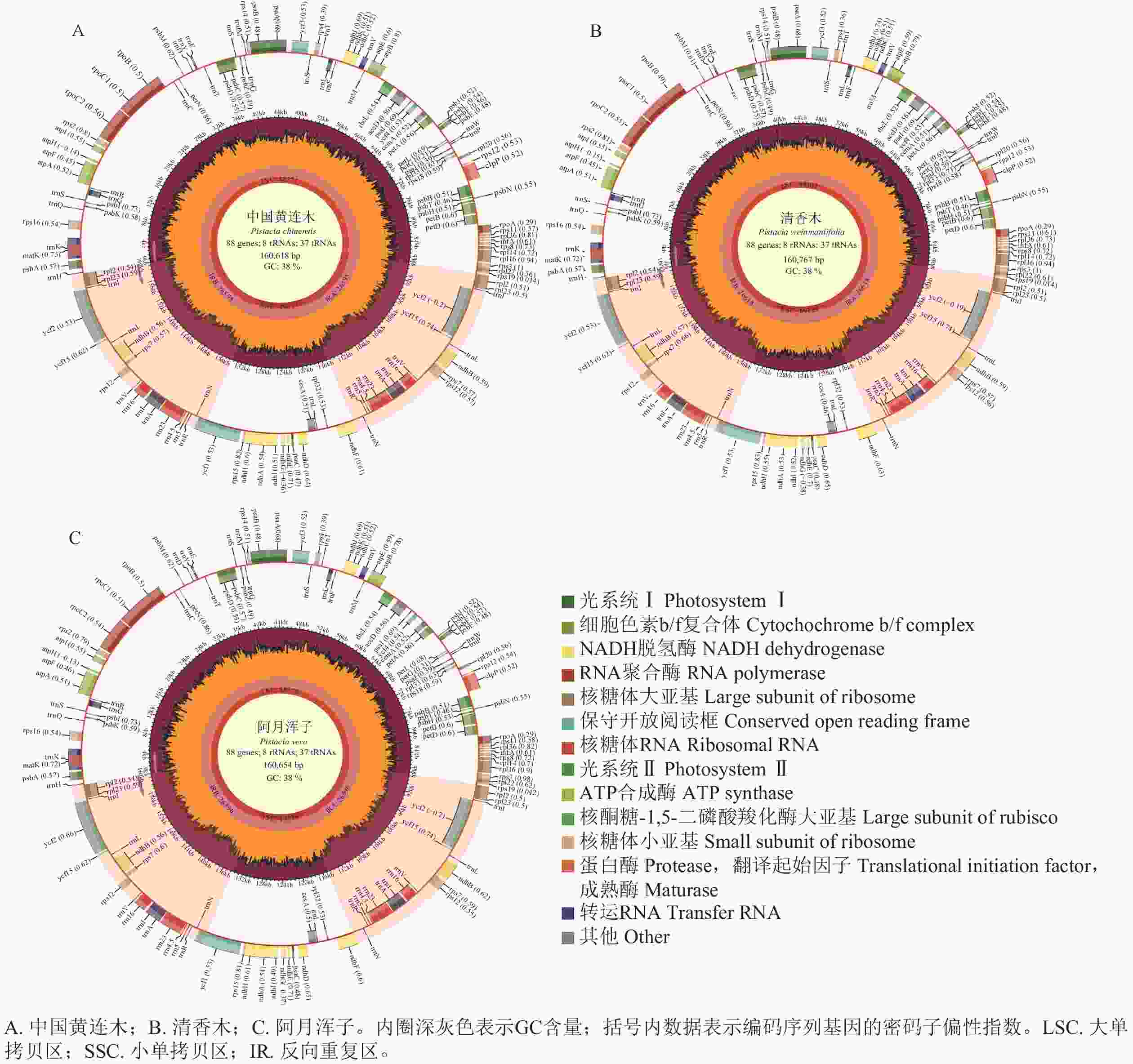
由 图1可见:3种黄连木属叶绿体基因组均由1个大单拷贝区域(LSC)、1个小单拷贝区域(SSC)和2个反向重复区域(IRs,包括IRa和IRb)组成的四分体环状结构。3种黄连木属叶绿体基因组的长度为160 618~160 767 bp (GC为37.87%~37.90%),其中LSC为88 371~88 402 bp (GC为36.00%~36.04%),IRs为26 595~26 618 bp (GC为42.93%~42.95%),SSC为19 057~19 129 bp (GC为32.42%~32.45%),且在蛋白编码序列数量(87个)、核糖体RNA数量(8个)和转运RNA数量(37个)保持一致( 表1)。
表 1 黄连木属植物叶绿体基因组的基本特征信息
Table 1. Basic characteristic information of the chloroplast genomes of Pistacia species
物种 全基因组 大单拷贝区 反向重复区 小单拷贝区 基因总
数/个蛋白编码序
列数量/个核糖体RNA
数量/个转运RNA
数量/个长度/bp GC含量/% 长度/bp GC含量/% 长度/bp GC含量/% 长度/bp GC含量/% 中国黄连木 160 618 37.90 88 371 36.04 26 595 42.95 19 057 32.45 132 87 8 37 清香木 160 767 37.87 88 402 36.00 26 618 42.93 19 129 32.42 132 87 8 37 阿月浑子 160 654 37.89 88 376 36.04 26 596 42.95 19 086 32.41 132 87 8 37 -
利用MISA在线工具在3种黄连木属植物叶绿体基因组中筛选出5种简单重复序列类型,共有262个潜在的简单重复序列位点,但未检测到六核苷酸。从 图2A可知:在这些潜在的简单重复序列位点中,单核苷酸中的A或T重复单元的数量最多,占比分别为34.35%、39.69%,其次是二核苷酸中的AT或TA重复单元,占比分别为3.44%、2.67%,其他重复单元的数量总体差异不大且占比很低。
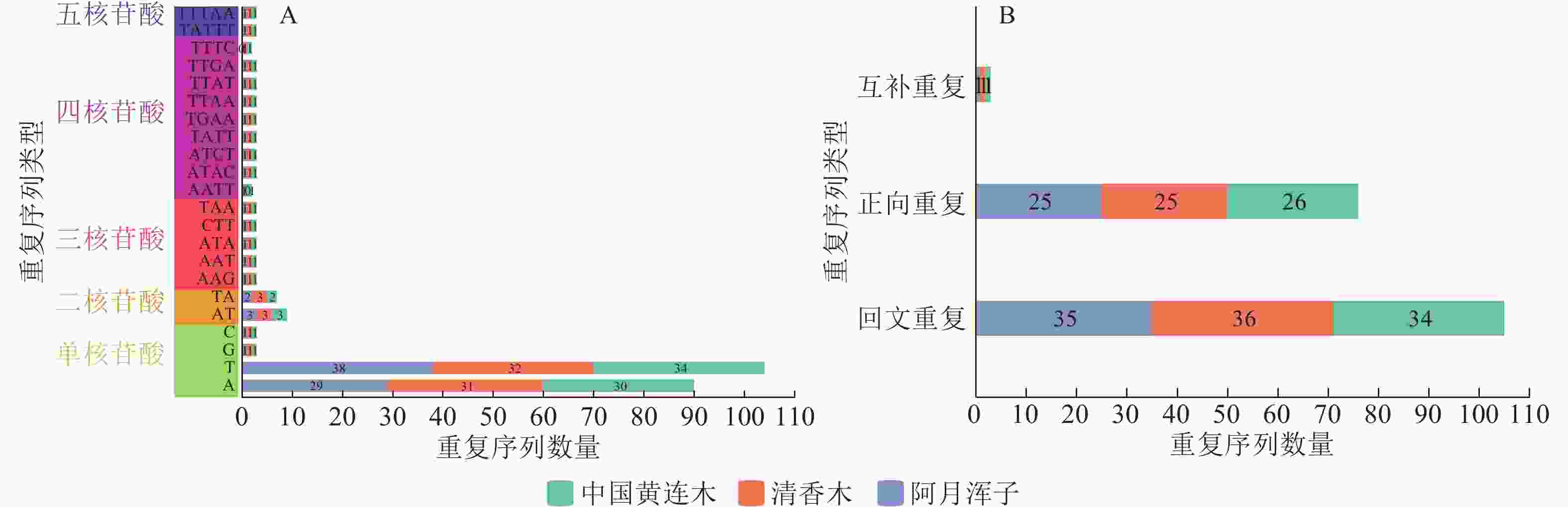
图 2 3种黄连木属植物叶绿体基因组的简单重复序列(A)和散在重复序列(B)统计
Figure 2. Simple repeat sequence (A) and scattered repeat sequence (B) statistics of chloroplast genomes of 3 Pistacia species
散在重复序列经REputer在线工具统计结果( 图2B)显示:3种黄连木属植物叶绿体基因组中共检测到184个散在重复序列位点,且同类型重复在各物种之间无明显差异,其中回文重复数量最多(105个),其次是正向重复(76个),互补重复最少(3个),各物种均未检测到反向重复序列。
-
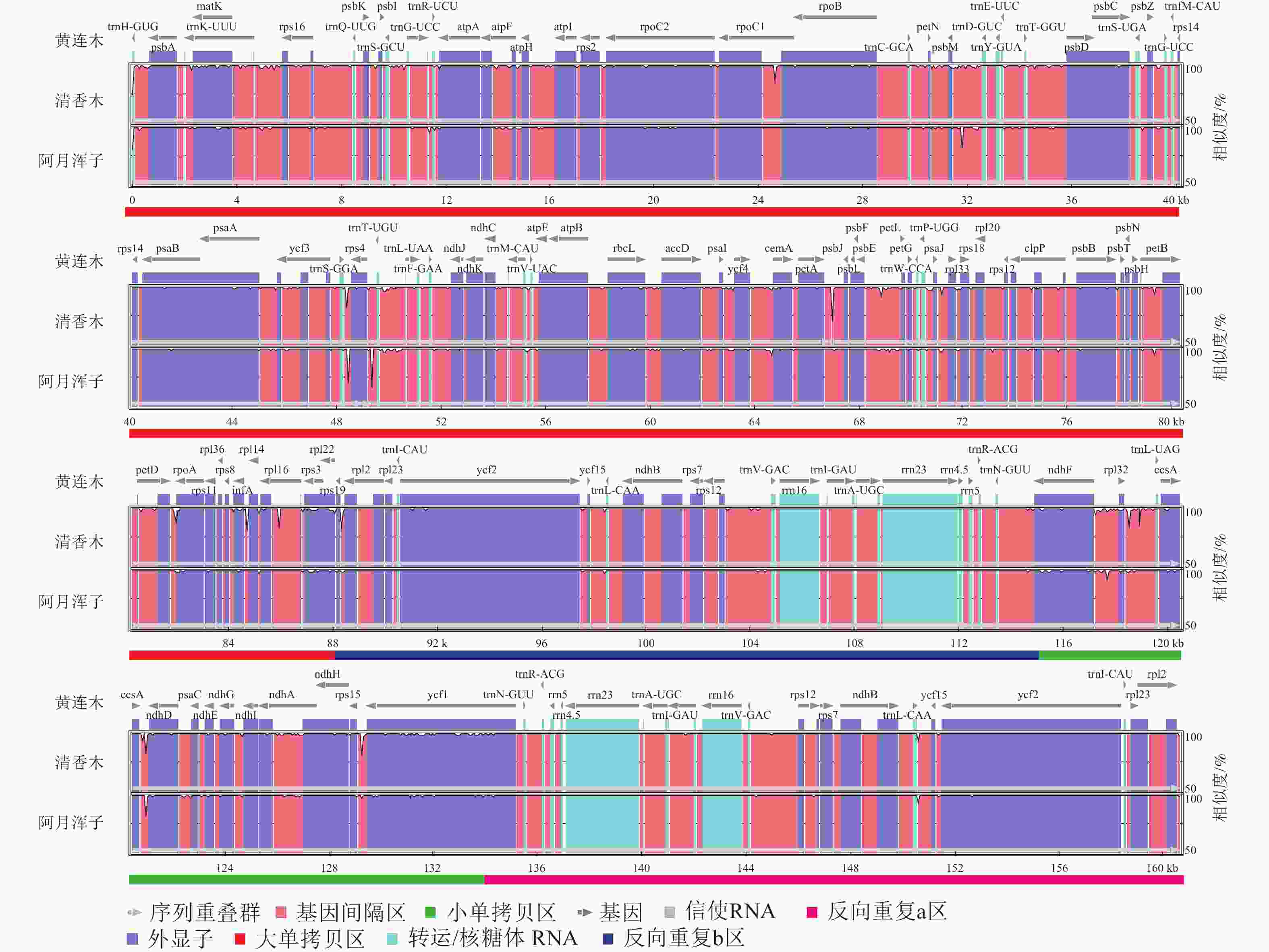
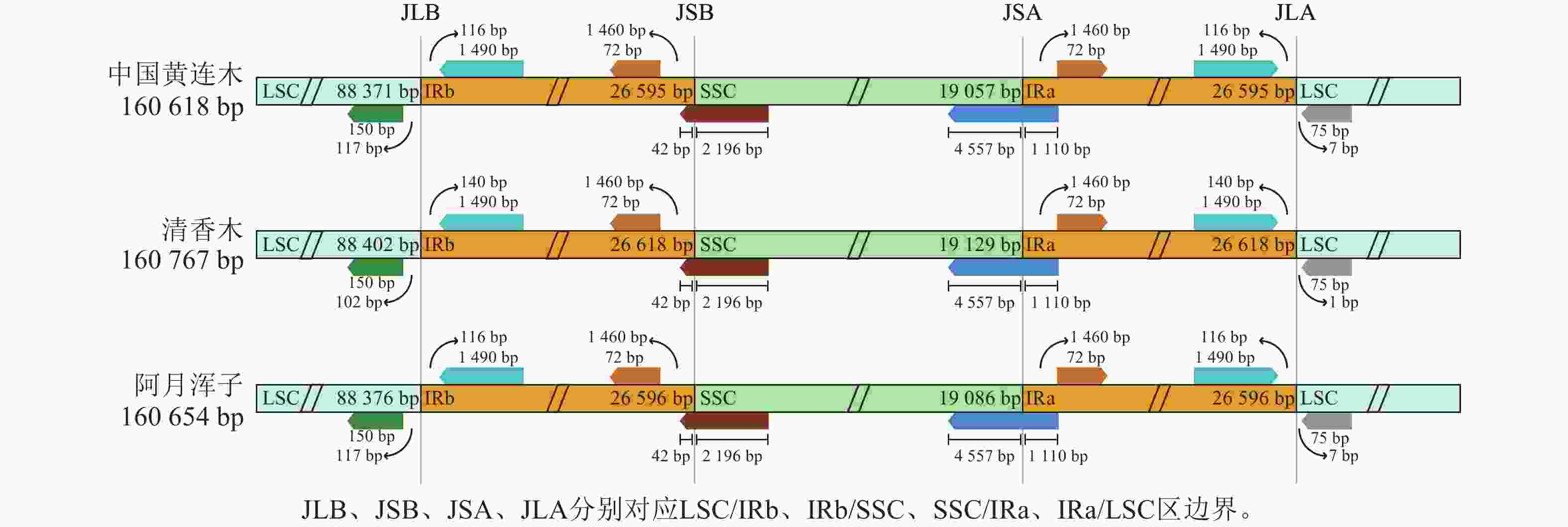
黄连木属植物叶绿体基因组的全序列差异对比结果( 图3)显示:3种黄连木属植物叶绿体基因组的转运/核糖体RNA (t/rRNA)、外显子(exon)的序列总体保持较高的保守性,而基因间隔区(CNS)序列发生不同程度的变异,集中在LSC区与SSC区,其中 trnS- GGA~ rps4、 ccsA~ ndhD等序列区域差异较为明显。四分体边界对比结果( 图4)显示: ndhF、 ycf1分别位于JSB、JSA边界线上, rpl2基因位于IR区且其3'端距离JSB/JSA边界线为116~140 bp,表明3种黄连木属植物在IRs区边界(JLB、JSB、JSA、JLA)附近的基因在收缩或扩张上无明显差异。
-
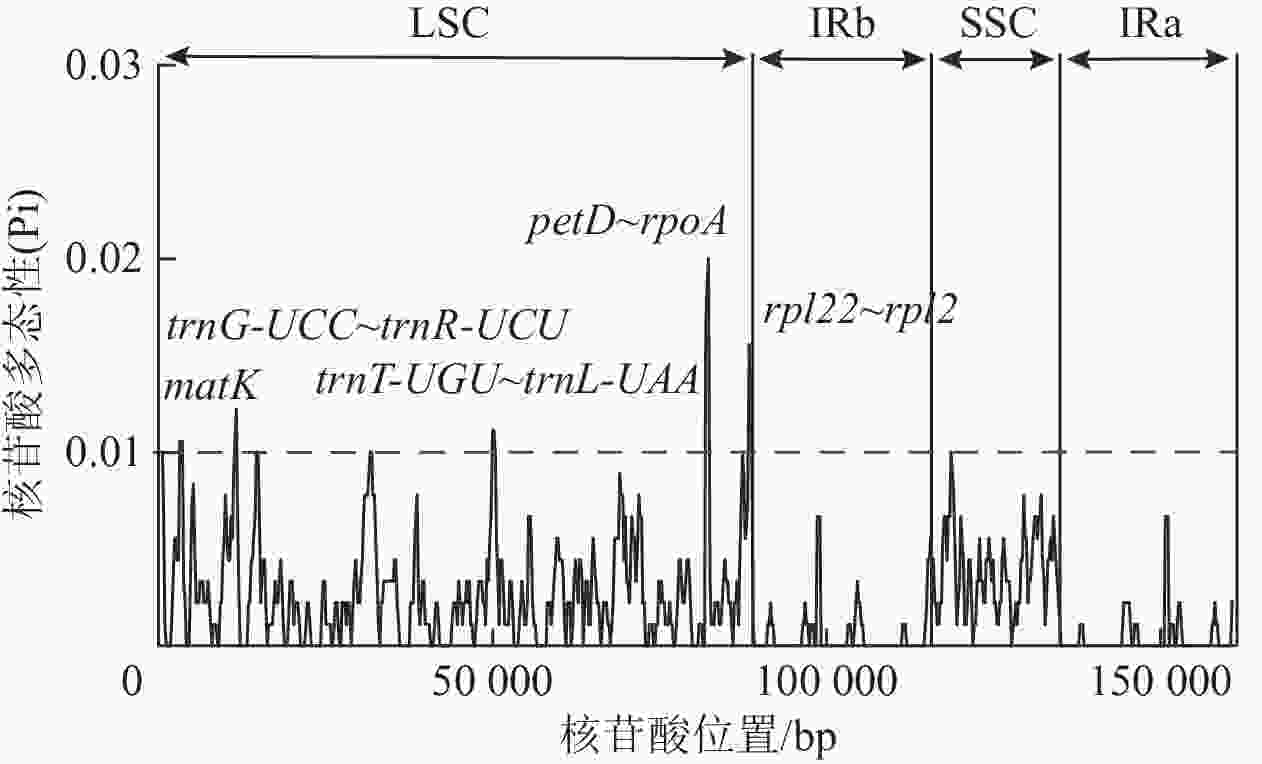
核苷酸多态性(Pi)分析结果( 图5)表明:3种黄连木属植物叶绿体基因组共包含了535个多态性位点,Pi为0~0.02,平均为0.002 23,并检测到 matK (2 830~3 829 bp)、 trnG- UCC~ trnR- UCU (11 112~11 911 bp)、 trnT- UGU~ trnL- UAA (49 664~50 487 bp)、 petD~ rpoA (81 644~82 650 bp)、 rpl22~ rpl2 (88 121~88 932 bp)等5个高变序列(Pi>0.01),除了 rpl22~ rpl2序列跨越LSC与IRb区,其余都位于LSC区内。
-
以2种盐肤木属植物为外类群,另加入其他黄连木属植物。植物系统发育树结果( 图6)显示:黄连木属与盐肤木属外类群分割开,清香木与中国黄连木的亲缘关系比较相近,而阿月浑子与大西洋黄连木以100%的支持率可单独划分一支。
-
与万寿菊属 Tagetes [ 23] 、大百合属 Cardiocrinum [ 24] 等陆生植物叶绿体基因组一样,3种黄连木属植物叶绿体基因组也均为闭合环状四分体结构,且在基因长度、基因顺序和GC含量等方面具有高度的保守性 [ 25− 26] 。GC含量在3种黄连木属叶绿体基因组的不同区域呈不均等分布,IRs区的GC含量(42.93%~42.95%)明显高于LSC区(36.00%~36.04%)和SSC区(32.41%~42.45%),可能是IRs区存在4个高GC含量的RNA基因( rrn4.5、 rrn5、 rrn16、 rrn23) [ 27− 28] ,不过IRs区的高GC含量也有助于叶绿体基因组的稳定性 [ 29] 。此外,本研究还发现叶绿体各种类型基因数量保持一致,表明了3种黄连木属植物在基因组成方面有较高的相似性,也是黄连木属植物难分类的重要因素之一。
叶绿体基因组中的重复序列具有较高的多态性,是植物群体遗传和进化研究的重要分子标记 [ 30− 31] 。本研究在检测3种黄连木属植物的所有散在重复序列类型中,发现回文重复与正向重复比例较大,与楠属 Phoebe [ 32] 、石蒜属 Lycoris [ 33] 等植物的结果基本一致。在黄连木属植物叶绿体基因组的简单重复序列类型中,单核苷酸的A或T重复单元所占比例最高,在忍冬属 Lonicera [ 34] 、紫麻属 Oreocnide [ 35] 等植物也有类似报道。A/T重复频率高的1个潜在原因是叶绿体基因组中的mRNA末端发生聚腺苷酸化 [ 36] 。另外,在质体复制过程中,A/T碱基的链比G/C碱基更容易分离,导致滑链错配 [ 37] 。这些分析所得到的重复序列位点代表了黄连木属植物丰富的变异,有助于检测居群、种内和栽培品种水平的遗传多态性,以及比较更远的亲缘关系。
研究叶绿体四分体不同区域的基因分布、边界信息、变异序列,对了解叶绿体基因组结构的差异、物种进化等具有重要意义。本研究表明:3种黄连木属植物叶绿体基因组具有很高的序列相似性,且IRs区序列变异程度很低,差异表现集中于LSC、SSC区的非编码区,这与紫薇属 Lagerstroemia [ 38] 、栎属 Quercus [ 39] 等的植物表现一致。虽然IRs区序列在大多数植物,特别在同一属内表现高度保守,但IRs区的边界普遍存在扩张或收缩现象,是导致不同植物群体的整个叶绿体基因组长度差异的主要因素 [ 40] 。通过比较3种黄连木属植物,发现四分体IRs边界没有明显的扩张或收缩差异,表明了3种黄连木属植物叶绿体基因组大小具有高度保守性。此外,本研究3种黄连木属植物叶绿体基因组的核苷酸多态性分析检测到5个变异序列( matK、 trnG- UCC~ trnR- UCU、 trnT- UGU~ trnL- UAA、 petD~ rpoA、 rpl22~ rpl2),这些高度变异序列可作为鉴定黄连木属的候选分子标记,特别是编码成熟蛋白酶的相关基因 matK,具有相对较快的突变率 [ 41] ,正成为研究多数被子植物系统学和进化的潜在候选基因 [ 42− 44] 。
明确黄连木属植物及其近缘属在系统进化中的生态位置,对于理解共同进化背景下黄连木属植物不同种的适应性进化及生物多样性具有重要意义。本研究系统发育树结果表明:黄连木属与盐肤木属在遗传进化上明确了两者的属级分类,与早期基于核糖体基因内的转录间隔区(ITS)序列构建的系统发育结果一致 [ 45] ;从黄连木属分支距离来看,中国黄连木与清香木在遗传进化中表现亲近,而阿月浑子与大西洋黄连木关系更为密切,这可能与地理距离及环境差异相关联。遗传分化是环境压力所施加的选择或遗传漂变的影响以及地理隔离限制部分群体基因交流的结果 [ 46] 。原产东亚的中国黄连木与清香木叶绿体基因组的亲近性暗示两者仍保留较高的母系基因交流历史。相比之下,产于中亚的阿月浑子与分布地中海的大西洋黄连木的密切关系反映了其共同祖先可能在气候干旱化过程中分化出适应不同干旱生态位的类群。这一结果为研究不同地域的黄连木属植物的“抗旱性”进化策略提供新的思路,也为黄连木属植物种质资源保护提供参考依据。
-
本研究发现:3种黄连木属植物叶绿体基因组都是序列长度在160 618~160 767 bp的环状四分体,序列结构类似且相对保守;简单重复序列以单核苷酸中的A或T重复单元为主,散在重复序列以回文重复与正向重复为主。检测到5个高变序列( matK、 trnG- UCC~ trnR- UCU、 trnT- UGU~ trnL- UAA、 petD~ rpoA、 rpl22~ rpl2)可作为黄连木属植物的潜在分子标记。基于全叶绿体基因比较,中国黄连木与清香木在遗传距离上相近,而阿月浑子与前两者相对疏远。
Structural characteristics and genetic relationship of chloroplast genomes of 3 Pistacia species
-
摘要:
目的 开展3种黄连木属 Pistacia植物叶绿体基因组的对比分析,旨在解析其结构特征及遗传进化关系。 方法 以美国国家生物技术信息中心(NCBI)的3种黄连木属植物叶绿体基因组为材料,利用生物信息学手段对其叶绿体基因组结构、重复序列、核苷酸多态性及遗传关系开展分析。 结果 3种黄连木属植物叶绿体基因组均呈四分体环状结构,且蛋白编码序列(CDS)、核糖体RNA (rRNA)及转运RNA (tRNA)的数目完全一致;简单重复序列统计显示:以单核苷酸中的A (34.35%)或T (39.69%)重复单元为主,但未检测到六核苷酸。散在重复序列统计显示:以回文重复(105个)、正向重复(76个)为主,但未检测到反向重复;3种黄连木属植物叶绿体基因组在大单拷贝区(LSC)、小单拷贝区(SSC)区域的基因间隔区(CNS)序列有不同程度的变异,但在四分体边界附近的基因在收缩或扩张上均无明显差异;核苷酸多态性分析在LSC区及LSC与反向重复b区(IRb)的边界检测到5个高度变异序列( matK、 trnG- UCC~ trnR- UCU、 trnT- UGU~ trnL- UAA、 petD~ rpoA、 rpl22~ rpl2);遗传进化关系显示:黄连木属植物与盐肤木属 Rhus植物相互分开,中国黄连木 P. chinensis与清香木 P. weinmaniifolia关系亲近,而阿月浑子 P. vera与大西洋黄连木 P. atlantica可单独划分一支。 结论 3种黄连木属植物叶绿体基因组结构类似且相对保守,在各类基因数量上保持一致;检测出的5个变异序列可作为黄连木属植物的候选分子标记;中国黄连木与清香木在遗传关系上更为亲近,而阿月浑子与前两者相对疏远。图6表1参46 Abstract:Objective In this study, the chloroplast genomes of 3 Pistacia species were compared to analyze their structural characteristics and genetic evolution. Method 3 publicly available chloroplast genomes of Pistacia species from National Center for Biotechnology Information (NCBI) were selected as research materials, and their chloroplast genome structure, repeat sequence, nucleotide polymorphism and genetic relationship were analyzed by relevant bioinformatics methods. Result The chloroplast genomes of the 3 species were tetrad ring structure, and the numbers of protein-coding sequence (CDS), ribosomal RNA (rRNA), and transfer RNA (tRNA) were exactly the same among them; The simple repeat sequences were mainly A (34.35%) or T (39.69%) repeat units in mono-nucleotide, but no hexa-nucleotide. And the number of scattered repeat sequences were mainly palindromic repeats (105) and forward repeats (76), but no reverse repeats; There were different degrees of variation in the conserved non-coding sequences (CNS) of the large single copy (LSC) and small single copy (SSC) of the chloroplast genomes of three species, but there was no significant difference in the contraction or expansion of genes near the tetrad boundary; 5 highly variable sequences ( matK, trnG- UCC~ trnR- UCU, trnT- UGU~ trnL- UAA, petD~ rpoA, rpl22~ rpl2) were identified in the LSC region and the boundary between LSC and inverted repeat b (IRb); Phylogenetic reconstruction based on complete chloroplast genomes demonstrated clear segregation between Pistacia and Rhus, and P. chinensis was closely related to P. weinmaniifolia, while P. vera and P. atlantica could be formed a distinct clade. Conclusion The chloroplast genome structure of the 3 Pistacia species was similar and relatively conservative, and the number of various genes was consistent; The 5 mutant sequences detected could be used as candidate molecular markers for Pistacia species; The genetic relationship between P. chinensis and P. weinmannifolia was similar, while P. vera was relatively distant from former 2 species. [Ch, 6 fig. 1 tab. 46 ref.] -
Key words:
- Pistacia /
- chloroplast genome /
- sequence repeat /
- genetic relationship
-
表 1 黄连木属植物叶绿体基因组的基本特征信息
Table 1. Basic characteristic information of the chloroplast genomes of Pistacia species
物种 全基因组 大单拷贝区 反向重复区 小单拷贝区 基因总
数/个蛋白编码序
列数量/个核糖体RNA
数量/个转运RNA
数量/个长度/bp GC含量/% 长度/bp GC含量/% 长度/bp GC含量/% 长度/bp GC含量/% 中国黄连木 160 618 37.90 88 371 36.04 26 595 42.95 19 057 32.45 132 87 8 37 清香木 160 767 37.87 88 402 36.00 26 618 42.93 19 129 32.42 132 87 8 37 阿月浑子 160 654 37.89 88 376 36.04 26 596 42.95 19 086 32.41 132 87 8 37 -
[1] ALZAHRANI D A. Complete chloroplast genome of Abutilon fruticosum: genome structure, comparative and phylogenetic analysis [J]. Plants, 2021, 10(2): 270. DOI: 10.3390/plants10020270. [2] 柳婷婷, 许丽爱, 胡紫蔚, 等. 菜心叶绿体基因组特征及系统发育分析[J/OL]. 浙江农林大学学报, 2025-09-29. https://kns.cnki.net/kcms/detail/33.1370.S.20250928.1915.006.html. LIU Tingting, XU Liai, HU Ziwei, et al. Characteristics of the chloroplast genome and phylogenetic analysis of flowering Chinese cabbage[J/OL]. Journal of Zhejiang A&F University, 2025-09-29 . https://kns.cnki.net/kcms/detail/33.1370.S.20250928.1915.006.html. LIU Tingting, XU Liai, HU Ziwei, et al. Characteristics of the chloroplast genome and phylogenetic analysis of flowering Chinese cabbage[J/OL]. Journal of Zhejiang A&F University, 2025-09-29 . https://kns.cnki.net/kcms/detail/33.1370.S.20250928.1915.006.html. [3] PALMER J. Comparative organization of chloroplast genomes [J]. Annual Review of Genetics, 1985, 19: 325−354. DOI: 10.1146/annurev.genet.19.1.325. [4] TAN Wei, GAO Han, JIANG Weiling, et al. The complete chloroplast genome of Gleditsia sinensis and Gleditsia japonica: genome organization, comparative analysis, and development of taxon specific DNA mini-barcodes [J]. Scientific Reports, 2020, 10: 16309. DOI: 10.1038/s41598-020-73392-7. [5] 朱梦飞, 胡迎峰, 师雪芹. 濒危植物新绒苔叶绿体基因组特征及系统发育位置分析[J]. 浙江农林大学学报, 2025, 42(1): 55−63. ZHU Mengfei, HU Yingfeng, SHI Xueqin. Characterization and phylogenetic location analysis of chloroplast of the endangered plant Neotrichocolea bissetii [J]. Journal of Zhejiang A&F University, 2025, 42(1): 55−63. DOI: 10.11833/j.issn.2095-0756.20240356. ZHU Mengfei, HU Yingfeng, SHI Xueqin. Characterization and phylogenetic location analysis of chloroplast of the endangered plant Neotrichocolea bissetii [J]. Journal of Zhejiang A&F University, 2025, 42(1): 55−63. DOI: 10.11833/j.issn.2095-0756.20240356 .[6] 江转转, 陈红, 鲍红艳, 等. 狼尾草属叶绿体基因组特征与分子标记开发[J]. 浙江农林大学学报, 2025, 42(2): 365−372. JIANG Zhuanzhuan, CHEN Hong, BAO Hongyan, et al. Chloroplast genome characteristics and molecular marker development of Pennisetum [J]. Journal of Zhejiang A&F University, 2025, 42(2): 365−372. DOI: 10.11833/j.issn.2095-0756.20240371. JIANG Zhuanzhuan, CHEN Hong, BAO Hongyan, et al. Chloroplast genome characteristics and molecular marker development of Pennisetum [J]. Journal of Zhejiang A&F University, 2025, 42(2): 365−372. DOI: 10.11833/j.issn.2095-0756.20240371 .[7] MOLINA A, HERVÁS-STUBBS S, DANIELL H, et al. High-yield expression of a viral peptide animal vaccine in transgenic tobacco chloroplasts [J]. Plant Biotechnology Journal, 2004, 2(2): 141−153. DOI: 10.1046/j.1467-7652.2004.00057.x. [8] JIN Honglei, DUAN Sujuan, ZHANG Pengxiang, et al. Dual roles for CND1 in maintenance of nuclear and chloroplast genome stability in plants [J]. Cell Reports, 2023, 42(3): 112268. DOI: 10.1016/j.celrep.2023.112268. [9] 沈植国, 宋宏伟, 韩健, 等. 世界黄连木属种质资源种类与分布综述[J]. 世界林业研究, 2012, 25(5): 29−34. SHEN Zhiguo, SONG Hongwei, HAN Jian, et al. Species and distribution for world germplasm resources of Pistacia L. [J]. World Forestry Research, 2012, 25(5): 29−34. DOI: 10.13348/j.cnki.sjlyyj.2012.05.010. SHEN Zhiguo, SONG Hongwei, HAN Jian, et al. Species and distribution for world germplasm resources of Pistacia L. [J]. World Forestry Research, 2012, 25(5): 29−34. DOI: 10.13348/j.cnki.sjlyyj.2012.05.010 .[10] 郑万钧. 中国树木志: 第4卷[M]. 北京: 中国林业出版社, 2004: 4227−4230. ZHENG Wanjun. Chinese Tree Chronicles: Volume 4[M]. Beijing: China Forestry Publishing House, 2004: 4227−4230. ZHENG Wanjun. Chinese Tree Chronicles: Volume 4[M]. Beijing: China Forestry Publishing House, 2004: 4227−4230. [11] LIU Xianghua, XING Chao, RUAN Ying, et al. Determination of fatty acid methyl esters in biodiesel produced from Pistacia chinensis oil by GC [J]. Applied Mechanics and Materials, 2013, 291/294: 253−256. DOI: 10.4028/www.scientific.net/amm.291-294.253. [12] TANG Mingli, ZHANG Pingping, ZHANG Liyun, et al. A potential bioenergy tree: Pistacia chinensis bunge [J]. Energy Procedia, 2012, 16: 737−746. DOI: 10.1016/j.egypro.2012.01.119. [13] 乔永锋, 彭永芳, 方云山, 等. 云南清香木绿叶和嫩红叶挥发性成分对比研究[J]. 安徽农业科学, 2013, 41(4): 1583−1584, 1587. QIAO Yongfeng, PENG Yongfang, FANG Yunshan, et al. Study on the volatile component of green and red leave of the Pistacia weinmannifolia [J]. Journal of Anhui Agricultural Sciences, 2013, 41(4): 1583−1584, 1587. DOI: 10.13989/j.cnki.0517-6611.2013.04.067. QIAO Yongfeng, PENG Yongfang, FANG Yunshan, et al. Study on the volatile component of green and red leave of the Pistacia weinmannifolia [J]. Journal of Anhui Agricultural Sciences, 2013, 41(4): 1583−1584, 1587. DOI: 10.13989/j.cnki.0517-6611.2013.04.067 .[14] ZENG Lin, TU Xiaolong, DAI He, et al. Whole genomes and transcriptomes reveal adaptation and domestication of pistachio [J]. Genome Biology, 2019, 20(1): 79. DOI: 10.1186/s13059-019-1686-3. [15] ZHENG Shuyu, POCZAI P, HYVÖNEN J, et al. Chloroplot: an online program for the versatile plotting of organelle genomes [J]. Frontiers in Genetics, 2020, 11: 576124. DOI: 10.3389/fgene.2020.576124. [16] BEIER S, THIEL T, MÜNCH T, et al. MISA-web: a web server for microsatellite prediction [J]. Bioinformatics, 2017, 33(16): 2583−2585. DOI: 10.1093/bioinformatics/btx198. [17] KURTZ S, CHOUDHURI J V, OHLEBUSCH E, et al. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucleic Acids Research, 2001, 29(22): 4633−4642. DOI: 10.1093/nar/29.22.4633. [18] FRAZER K A, PACHTER L, POLIAKOV A, et al. VISTA: computational tools for comparative genomics [J]. Nucleic Acids Research, 2004, 32: W273−W279. DOI: 10.1093/nar/gkh458. [19] LI Huie, GUO Qiqiang, XU Lei, et al. CPJSdraw: analysis and visualization of junction sites of chloroplast genomes [J]. PeerJ, 2023, 11: e15326. DOI: 10.7717/peerj.15326. [20] ROZAS J, FERRER-MATA A, SÁNCHEZ-DELBARRIO J C, et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets [J]. Molecular Biology and Evolution, 2017, 34(12): 3299−3302. DOI: 10.1093/molbev/msx248. [21] KATOH K, MISAWA K, KUMA K I, et al. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform [J]. Nucleic Acids Research, 2002, 30(14): 3059−3066. DOI: 10.1093/nar/gkf436. [22] KUMAR S, NEI M, DUDLEY J, et al. MEGA: a biologist-centric software for evolutionary analysis of DNA and protein sequences [J]. Briefings in Bioinformatics, 2008, 9(4): 299−306. DOI: 10.1093/bib/bbn017. [23] 刘潮, 叶秀传, 杨春会, 等. 万寿菊属物种叶绿体基因组特征及系统发育分析[J]. 曲靖师范学院学报, 2024, 43(3): 24−30. LIU Chao, YE Xiuchuan, YANG Chunhui, et al. Chloroplast genomic characterization and phylogenetic analysis of Tagetes species [J]. Journal of Qujing Normal University, 2024, 43(3): 24−30. DOI: 10.3969/j.issn.1009-8879.2024.03.005. LIU Chao, YE Xiuchuan, YANG Chunhui, et al. Chloroplast genomic characterization and phylogenetic analysis of Tagetes species [J]. Journal of Qujing Normal University, 2024, 43(3): 24−30. DOI: 10.3969/j.issn.1009-8879.2024.03.005 .[24] LU Ruisen, LI Pan, QIU Yingxiong. The complete chloroplast genomes of three Cardiocrinum (Liliaceae) species: comparative genomic and phylogenetic analyses [J]. Frontiers in Plant Science, 2016, 7: 2054. DOI: 10.3389/fpls.2016.02054. [25] STRAUSS S H, PALMER J D, HOWE G T, et al. Chloroplast genomes of two conifers lack a large inverted repeat and are extensively rearranged [J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(11): 3898−3902. DOI: 10.1073/pnas.85.11.3898. [26] 修志莹, 赵艳玲, 程永琴, 等. 牡荆属叶绿体基因组比较分析及系统发育分析[J]. 广西植物, 2024, 44(9): 1755−1771. XIU Zhiying, ZHAO Yanling, CHENG Yongqin, et al. Comparative analysis of chloroplast genomes and phylogenetic analysis of Vitex [J]. Guihaia, 2024, 44(9): 1755−1771. DOI: 10.11931/guihaia.gxzw202307027. XIU Zhiying, ZHAO Yanling, CHENG Yongqin, et al. Comparative analysis of chloroplast genomes and phylogenetic analysis of Vitex [J]. Guihaia, 2024, 44(9): 1755−1771. DOI: 10.11931/guihaia.gxzw202307027 .[27] YANG Tiange, WU Zhihua, TIE Jun, et al. A comprehensive analysis of chloroplast genome provides new insights into the evolution of the genus Chrysosplenium [J]. International Journal of Molecular Sciences, 2023, 24(19): 14735. DOI: 10.3390/ijms241914735. [28] SUN Jiahui, WANG Yiheng, LIU Yanlei, et al. Evolutionary and phylogenetic aspects of the chloroplast genome of Chaenomeles species [J]. Scientific Reports, 2020, 10: 11466. DOI: 10.1038/s41598-020-67943-1. [29] DING Shixiong, LI Jiachen, HU Ke, et al. Morphological characteristics and comparative chloroplast genome analyses between red and white flower phenotypes of Pyracantha fortuneana (Maxim. ) Li (Rosaceae), with implications for taxonomy and phylogeny [J]. Genes, 2022, 13(12): 2404. DOI: 10.3390/genes13122404. [30] BODIN S S, KIM J S, KIM J H. Complete chloroplast genome of Chionographis japonica (Willd. ) Maxim. (Melanthiaceae): comparative genomics and evaluation of universal primers for Liliales [J]. Plant Molecular Biology Reporter, 2013, 31(6): 1407−1421. DOI: 10.1007/s11105-013-0616-x. [31] ZHAO Yongbing, YIN Jinlong, GUO Haiyan, et al. The complete chloroplast genome provides insight into the evolution and polymorphism of Panax ginseng [J]. Frontiers in Plant Science, 2014, 5: 696. DOI: 10.3389/fpls.2014.00696. [32] 戴前莉, 朱恒星, 魏卓, 等. 2种楠属植物叶绿体基因组特征及系统发育[J]. 东北林业大学学报, 2024, 52(6): 58−63, 84. DAI Qianli, ZHU Hengxing, WEI Zhuo, et al. Characteristics and phylogenetic of the chloroplast genomes of two Phoebe plants [J]. Journal of Northeast Forestry University, 2024, 52(6): 58−63, 84. DOI: 10.13759/j.cnki.dlxb.2024.06.004. DAI Qianli, ZHU Hengxing, WEI Zhuo, et al. Characteristics and phylogenetic of the chloroplast genomes of two Phoebe plants [J]. Journal of Northeast Forestry University, 2024, 52(6): 58−63, 84. DOI: 10.13759/j.cnki.dlxb.2024.06.004 .[33] 付文佛, 肖涛, 张永洪, 等. 石蒜属植物叶绿体基因组结构及系统学研究[J]. 亚热带植物科学, 2023, 52(4): 271−286. FU Wenfo, XIAO Tao, ZHANG Yonghong, et al. Chloroplast genome characteristics and phylogeny of the genus Lycoris(Amaryllidaceae) [J]. Subtropical Plant Science, 2023, 52(4): 271−286. DOI: 10.3969/j.issn.1009-7791.2023.04.001. FU Wenfo, XIAO Tao, ZHANG Yonghong, et al. Chloroplast genome characteristics and phylogeny of the genus Lycoris(Amaryllidaceae) [J]. Subtropical Plant Science, 2023, 52(4): 271−286. DOI: 10.3969/j.issn.1009-7791.2023.04.001 .[34] 熊瑶, 童凌斐, 曹岚, 等. 四种忍冬属植物叶绿体基因组结构特征及系统发育分析[J]. 药学学报, 2024, 59(11): 3164−3171. XIONG Yao, TONG Lingfei, CAO Lan, et al. Structural characteristics and phylogenetic analysis of chloroplast genomes of four species of Lonicera [J]. Acta Pharmaceutica Sinica, 2024, 59(11): 3164−3171. DOI: 10.16438/j.0513-4870.2024-0306. XIONG Yao, TONG Lingfei, CAO Lan, et al. Structural characteristics and phylogenetic analysis of chloroplast genomes of four species of Lonicera [J]. Acta Pharmaceutica Sinica, 2024, 59(11): 3164−3171. DOI: 10.16438/j.0513-4870.2024-0306 .[35] 秃玉翔, 赵文植, 沈伟祥, 等. 紫麻属叶绿体全基因组特征及系统发育分析[J]. 种子, 2023, 42(12): 24−30, 37. TU Yuxiang, ZHAO Wenzhi, SHEN Weixiang, et al. Characterization of complete chloroplast genome and phylogenetic analysis of Oreocnide [J]. Seed, 2023, 42(12): 24−30, 37. DOI: 10.16590/j.cnki.1001-4705.2023.12.024. TU Yuxiang, ZHAO Wenzhi, SHEN Weixiang, et al. Characterization of complete chloroplast genome and phylogenetic analysis of Oreocnide [J]. Seed, 2023, 42(12): 24−30, 37. DOI: 10.16590/j.cnki.1001-4705.2023.12.024 .[36] YAN Linjun, WANG Huanli, HUANG Xi, et al. Chloroplast genomes of genus Tilia: comparative genomics and molecular evolution [J]. Frontiers in Genetics, 2022, 13: 925726. DOI: 10.3389/fgene.2022.925726. [37] ZHAO Fei, LI Bo, DREW B T, et al. Leveraging plastomes for comparative analysis and phylogenomic inference within Scutellarioideae (Lamiaceae) [J]. PLoS One, 2020, 15(5): e0232602. DOI: 10.1371/journal.pone.0232602. [38] XU Chao, DONG Wenpan, LI Wenqing, et al. Comparative analysis of six Lagerstroemia complete chloroplast genomes [J]. Frontiers in Plant Science, 2017, 8: 15. DOI: 10.3389/fpls.2017.00015. [39] YANG Yanci, ZHOU Tao, DUAN Dong, et al. Comparative analysis of the complete chloroplast genomes of five Quercus species [J]. Frontiers in Plant Science, 2016, 7: 959. DOI: 10.3389/fpls.2016.00959. [40] YU Xiaoyue, ZUO Lihui, LU Dandan, et al. Comparative analysis of chloroplast genomes of five Robinia species: genome comparative and evolution analysis [J]. Gene, 2019, 689: 141−151. DOI: 10.1016/j.gene.2018.12.023. [41] PENJOR T, YAMAMOTO M, UEHARA M, et al. Phylogenetic relationships of Citrus and its relatives based on matK gene sequences [J]. PLoS One, 2013, 8(4): e62574. DOI: 10.1371/journal.pone.0062574. [42] GAO Ting, SUN Zhiying, YAO Hui, et al. Identification of Fabaceae plants using the DNA barcode matK [J]. Planta Medica, 2011, 77(1): 92−94. DOI: 10.1055/s-0030-1250050. [43] 郑梦迪, 张春, 马瑞龙, 等. 基于matK和ITS2及二级结构对药材香薷及其混伪品的鉴别研究[J]. 中国现代应用药学, 2022, 39(17): 2222−2228. ZHENG Mengdi, ZHANG Chun, MA Ruilong, et al. Identification of moslae herba and its adulterants based on matK, ITS2 and its secondary structure [J]. Chinese Journal of Modern Applied Pharmacy, 2022, 39(17): 2222−2228. DOI: 10.13748/j.cnki.issn1007-7693.2022.17.008. ZHENG Mengdi, ZHANG Chun, MA Ruilong, et al. Identification of moslae herba and its adulterants based on matK, ITS2 and its secondary structure [J]. Chinese Journal of Modern Applied Pharmacy, 2022, 39(17): 2222−2228. DOI: 10.13748/j.cnki.issn1007-7693.2022.17.008 .[44] TAUFIQ PROBOJATI R, HADIYANTI N, HAPSARI L. Identification and genetic diversity of pineapple local accessions from kediri and closely related species (Bromeliaceae) based on matK gene [J]. Biotropika: Journal of Tropical Biology, 2024, 12(3): 111−118. DOI: 10.21776/ub.biotropika.2024.012.03.01. [45] YI Tingshuang, WEN Jun, GOLAN-GOLDHIRSH A, et al. Phylogenetics and reticulate evolution in Pistacia (Anacardiaceae) [J]. American Journal of Botany, 2008, 95(2): 241−251. DOI: 10.3732/ajb.95.2.241. [46] LIU Li, WANG Zhen, SU Yingjuan, et al. Population transcriptomic sequencing reveals allopatric divergence and local adaptation in Pseudotaxus chienii (Taxaceae) [J]. BMC Genomics, 2021, 22(1): 388. DOI: 10.1186/s12864-021-07682-3. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20250221






 下载:
下载: