-
景宁木兰Magnolia sinostellata是木兰科Magnoliaceae木兰属Magnolia植物,属于典型的极小种群濒危树种。景宁木兰幼苗生长需要一定的荫庇环境[1],但过度遮阴会抑制景宁木兰幼苗的光合速率[2]。自然状态下,群落所形成的遮阴是影响群落内濒危植物生长发育的重要因素[3]。景宁木兰多生长于光照充足的针叶群落,在针阔混交群落和阔叶群落分布较少[4]。据此推断,群落所形成的遮阴会削弱景宁木兰的生长势,是导致景宁木兰濒危的重要因素。光是影响植物生长发育的重要环境因子。植物的整个生命周期都受到不断变化的光环境影响。光敏色素是植物重要的光感受器,能够感受环境中光信号的变化。光敏色素作用因子(phytochrome interacting factors, PIFs)能够感知红光和远红光信号,在光信号传导和植物生长发育中起到重要作用。光信号在通过信号转导后,能够诱导一系列生理生化反应。光敏色素作用因子是光信号转导中的重要调控因子,属于bHLH超家族的第15亚族,其家族成员都具有高度保守的bHLH结构域。bHLH结构域由约15个氨基酸的碱性结构域(basic region)和60个氨基酸的HLH(helix-loop-helix)结构域组成[5]。目前对PIF家族转录因子的研究主要集中在模式植物拟南芥Arabidopsis thaliana中。PIF转录因子广泛参与了拟南芥的光调控反应,并在光敏色素介导的光信号网络调控中处于主导地位[6]。黑暗条件下,AtPIF1能够抑制拟南芥种子的萌发,AtPIF3、AtPIF4、AtPIF5能够促进叶绿素分解并抑制叶绿体活性,从而最终导致叶片衰老[7]。遮阴条件下,AtPIF1能够通过调控AtHB1的表达来促进下胚轴的伸长[8];AtPIF4、AtPIF5和AtPIF7通过促进AtFT和AtTSF的表达从而促进拟南芥开花[9];AtPIF4、AtBZR1和AtARF6通过整合生长素信号与BR和光敏色素通路来协同促进拟南芥下胚轴的伸长[10]。由此可见,PIF家族转录因子是遮阴条件下光信号转导途径中的关键调控因子。据此推测,PIF家族转录因子在光照强度对木兰属濒危树种生长发育的影响及光受体信号转导途径中起到重要作用。因此,本研究对景宁木兰PIF家族转录因子进行分析鉴定,探究其在极端遮阴条件下的表达模式,从而为揭示光介导的景宁木兰生长发育的分子机制、群落引起的遮阴而导致景宁木兰濒危的内在机制奠定基础,最终为珍稀植物景宁木兰的保育提供理论依据。
HTML
-
景宁木兰来自浙江省杭州市青山湖花园中心苗圃培育的3年生扦插苗。2019年6月下旬,选择长势一致且无病虫害的扦插苗作为材料,置于温度为(25±2) ℃、光照强度为2 000~2 500 lx、相对湿度为40%~60%的人工气候室中进行培养,光照与黑暗时间各12 h,定期浇水保持土壤湿润。
-
3层遮阳网形成的光照强度是景宁木兰自然生境光照条件之一,会显著削弱景宁木兰的生长势[1]。2019年7月上旬开始遮阴处理,本研究于浙江省杭州市临安区浙江农林大学人工气候室内,用3层黑色遮阳网及竹竿搭建极端遮阴处理(25%光照,ST)装置,以模拟遮阴条件下的景宁木兰野外生存环境。以100%全光照为对照(ck)。所有盆栽苗叶片之间无重叠。每个处理4个生物学重复。分别自实验开始处理第0、1、3、5、10、20、30天时,拍照记录植株表型变化、采集景宁木兰植株的中层叶片装入锡纸袋中,并迅速置于液氮中速冻,随后转入−80 ℃冰箱中储存,用于植物RNA的提取及后续实时荧光定量聚合酶链式反应(qRT-PCR)实验。
-
基于实验室已有的转录组测序数据,在NR、NT、Swiss-Prot这3个数据库中进行注释,初步注释到46个PIF转录因子相关基因,将这些基因编码蛋白质逐个进行NCBI Blast和NCBI CDD预测,去除重复序列及冗余转录本后,最终得到9个PIF家族转录因子的蛋白质序列。利用MEGA 5.0对景宁木兰PIF转录因子序列与拟南芥PIF转录因子序列进行比对分析,根据TOLEDO等[5]命名拟南芥PIF家族的方法对9个景宁木兰PIF转录因子进行命名。拟南芥PIF转录因子序列从拟南芥转录因子数据库中获得。
-
采用Compute pI/Mw在线工具预测景宁木兰PIF转录因子蛋白质的等电点及分子量,采用在线网站Cell-PLoc 2.0预测亚细胞定位,利用NetPhos分析蛋白质磷酸化位点;利用MEME分析蛋白质保守结构域;利用DNAMAN比对分析保守结构域;利用在线软件heatmapper绘制热图;为研究PIF转录因子家族的进化关系,利用MEGA5.0将9个景宁木兰MsPIF蛋白质序列、15个拟南芥AtPIF蛋白质序列[11]、6个玉米Zea mays ZmPIF蛋白质序列[12]、10个杨树Populus trichocar PtPIF蛋白质序列[13]进行序列比对,并用邻接法构建系统进化树,进行Bootstrap测试,重复设置为1 000 次。
-
采用植物RNA提取试剂盒提取样品总RNA。取1 μg RNA进行第1链 cDNA的合成。所用定量PCR试剂为BCG qPCR Master Mix。根据景宁木兰转录组数据设计特异性引物(表1)。以景宁木兰的EF1-α基因作为内参基因[1],利用Light Cycler 480II(Roche)仪器进行目的基因qRT-PCR表达分析。反应程序结束后,65~95 ℃每隔0.2 s作溶解曲线,采用
${2^{ - \Delta \Delta {C_{\rm{t}}}}} $ 法计算目的基因的相对表达量。每个样品重复3次。基因名称 正向(反向)引物序列(5'→3') 退火温度/℃ 扩增效率/% 相关系数R2 MsbHLH68 F-CCTCAGGCTCTGTCATTGGG 59.5 90.86 0.998 R-AGCGGTTGGGCTTCTTCACG 59.5 MsPIF4 F-GCCGAAGAATCTGAGTGCCA 57.4 96.98 0.999 R-CTATCCCTCCGTCTCCTTTC 57.4 MsbHLH1 F-CTTTGTGCTATCTTCGGGAACG 57.7 104.48 0.997 R-GGGACCCACCAATCAACGAC 59.5 MsPIF8 F-AAGCGAGGATGAGAACAAGG 55.4 93.01 0.983 R-TGCGTTCGGATTGGTTATGG 55.4 MsbHLH66 F-AGCAGTAACGGCACGCAGAC 59.5 101.57 0.995 R-GAAATGGGCATGAGGCAGAG 57.4 MsPIF3 F-CAGACTCAGCCGTCAACTCA 57.4 92.93 0.991 R-GCAGGCCCACTTCCACCAAT 59.5 MsPIF1 F-CCACATATTCCTCCATTTGAT 51.7 100.90 0.994 R-GAAGGCTGAGGTAGTGTTGATA 55.8 MsbHLH48 F-CCGTCGAGTCTCCAGTGGTC 61.6 96.92 0.993 R-CGTTTGAAGGGAAGGTAGGG 57.4 MsbHLH23 F-GCAGACAAAGGACCCAAGGA 57.4 93.01 0.993 R-CACGAGGAGTGAGGAACAAGAA 57.7 Table 1. Primers used for qRT-PCR analysis
-
采用SPSS 19.0进行方差分析并用SigmaPlot 14绘图,对平均值采用独立样本T检验进行比较,显著水平为0.05。
1.1. 植物材料
1.2. 实验设计
1.3. 方法
1.3.1. 景宁木兰PIF家族转录因子的筛选
1.3.2. 景宁木兰PIF家族转录因子的理化性质分析
1.3.3. 极端遮阴条件下景宁木兰PIF家族转录因子的表达模式
1.3.4. 数据分析
-
利用ExPASy对9个基因编码的氨基酸进行理化性质进行分析(表2),发现9个基因编码的蛋白质大小不等,最小的为MsPIF1,编码188个氨基酸,最大的为MsPIF3,编码735个氨基酸。9个蛋白质的理论等电点为5.18(MsbHLH68)~8.22(MsPIF8),表明不同的PIF家族蛋白质在不同的微环境中发挥不同的生物学功能。9个PIF家族蛋白质均属于不稳定、亲水性蛋白质。
基因名称 蛋白质分子量/Da 氨基酸数/个 等电点 不稳定系数 脂肪系数 平均亲水性 亚细胞定位 磷酸化位点 Ser Thr Tyr MsbHLH68 53 604.79 506 5.18 58.19 77.51 −0.447 细胞核(N) 47 10 1 MsPIF4 57 883.06 527 5.96 56.05 64.57 −0.611 细胞核(N) 40 12 3 MsbHLH1 74 557.30 671 5.52 53.08 79.66 −0.452 细胞核(N) 34 14 6 MsPIF8 50 048.29 456 8.22 42.09 58.29 −0.686 细胞核(N) 36 20 4 MsbHLH66 45 976.46 430 6.79 60.45 69.72 −0.558 细胞核(N) 45 14 1 MsPIF3 78 957.02 735 6.31 67.37 63.80 −0.513 细胞核(N) 74 13 3 MsPIF1 20 314.56 188 5.93 60.78 60.32 −0.341 细胞核(N) 4 2 1 MsbHLH48 40 255.00 366 6.43 59.85 70.90 −0.639 细胞核(N) 40 6 2 MsbHLH23 39 480.07 362 5.82 72.07 70.14 −0.577 细胞核(N) 43 9 2 说明:Ser为丝氨酸,Thr为苏氨酸,Tyr为酪氨酸 Table 2. MsPIF gene family of M. sinostellata
-
通过在线软件Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)对9个PIF基因家族成员的氨基酸序列进行亚细胞定位预测(表2),9个基因编码的蛋白质均定位于细胞核内,符合其转录因子的功能,可能通过调控细胞核基因的转录来发挥功能。
-
蛋白质磷酸化在很多植物信号转导过程中起着重要作用[14]。蛋白质磷酸化的可逆性使得植物细胞能够快速响应外部环境变化[15]。在正常光条件下,PIF家族转录因子与光敏色素相互作用,从而导致PIF蛋白质的磷酸化和降解[16]。而在低光照条件下,PIF蛋白质的磷酸化和降解受到抑制[17]。因此,分析PIF蛋白质的磷酸化位点对研究其在信号转导中的功能有重要意义。利用NetPhos对9个PIF家族蛋白质的磷酸化位点进行分析(表2),发现9个蛋白质均具有丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)磷酸化位点,且Ser的磷酸化位点最多,Tyr的磷酸化位点最少。其中磷酸化位点最多的是MsPIF3,为94个,MsPIF1的磷酸化位点最少,仅7个。
-
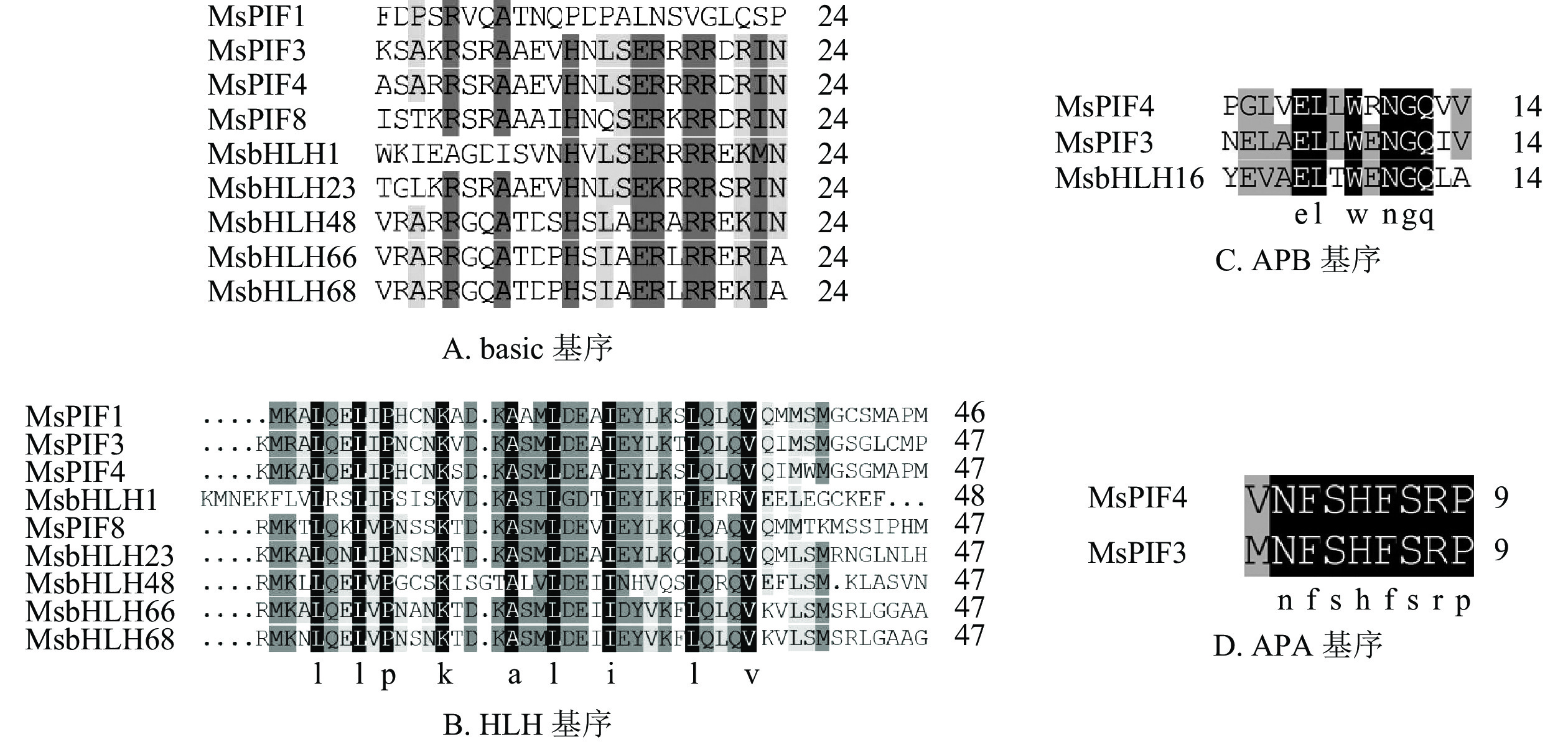
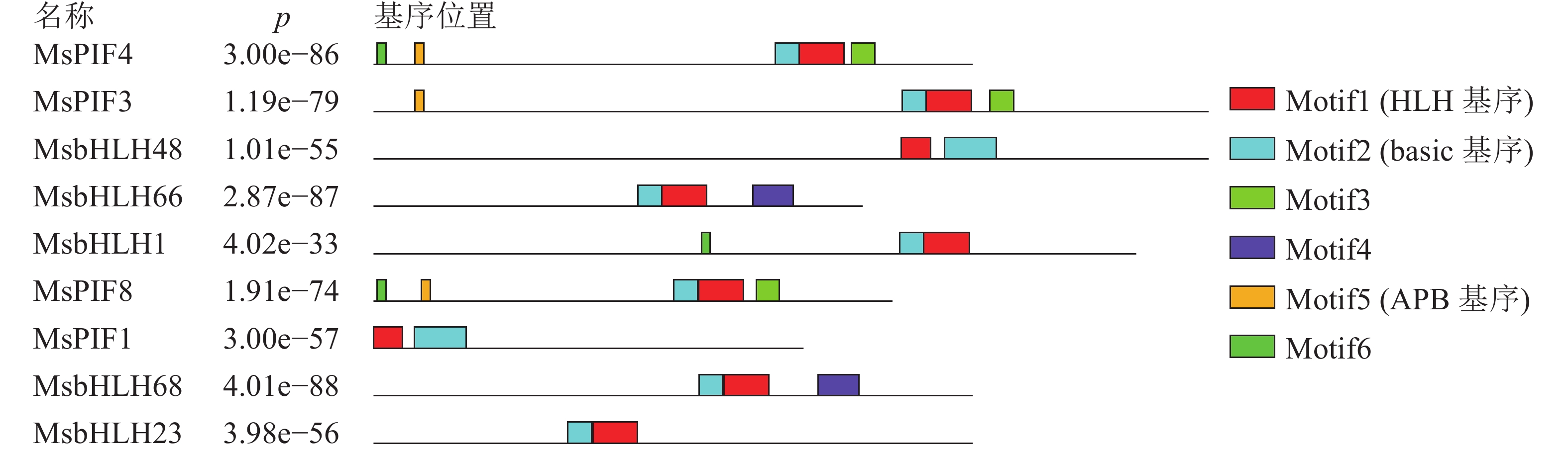
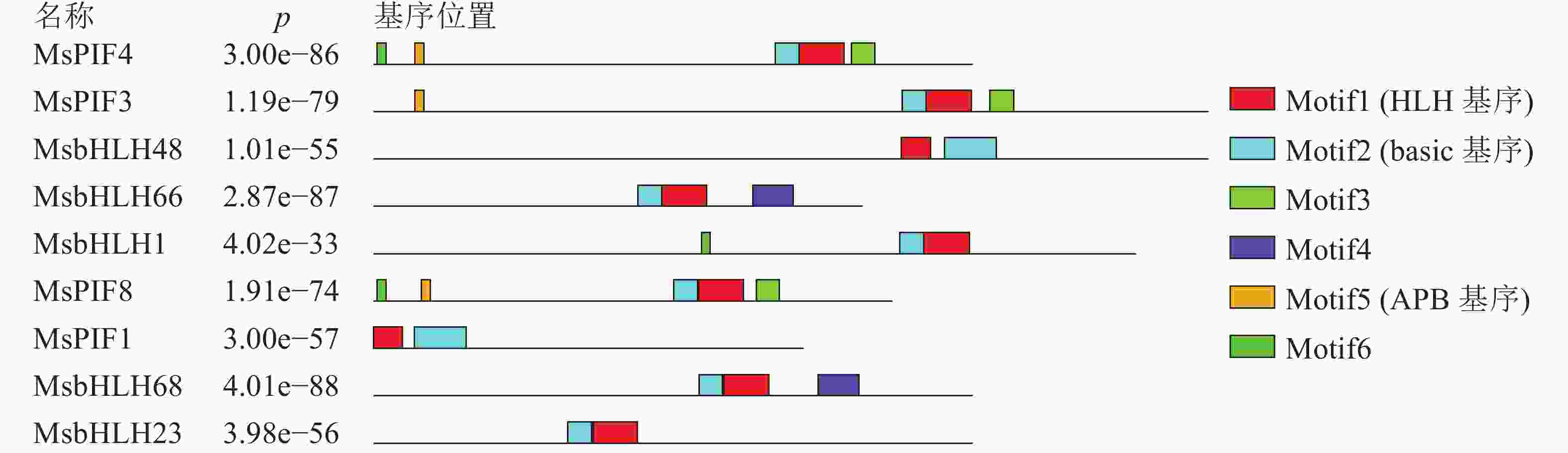
进一步利用MEME在线软件分析MsPIF蛋白质[16],共鉴定到6个基序(表3)。结果显示(图1):9个蛋白质含有不同数目的基序,MsPIF4和MsPIF8含有的基序最多,为5个,而MsbHLH48中含有的基序最少,仅2个。bHLH家族蛋白质均含有bHLH基序,该基序由位于N端的basic基序(图2A)和位于C端的HLH基序(图2B)组成[5]。9个PIF家族蛋白质均含有basic基序及HLH基序。不同蛋白质的同源异型基序之间存在一定差异。MsPIF4、MsPIF3和MsPIF8中含有APB基序(active phyB-binding motif)(图2C),表明其可以与PhyB(phytochrome B)相结合,从而对下游基因的表达进行调控[16]。此外,MsPIF4和MsPIF3中还含有APA基序(active phyA-binding motif)(图2D),表明其既可以与PhyA(phytochrome A)相结合也可以与PhyB相结合。
基序 宽度/个 最佳匹配 Motif1 40 MKALQELIPNSNKTDKASMLDEAIEYLKSLQLQVQMLSM Motif2 21 RGRAAEVHNLSERRRRERINE Motif3 21 MMFPGVQQYMPPMGMGMGMGM Motif4 36 FEQZIVKLMEEDMGSAMQYLQGKGLCLMPIALAAAI Motif5 8 ELLWENGQ Motif6 8 CVPBWBTD Table 3. Major MEME motif sequences in M. sinostellata PIF protein
-
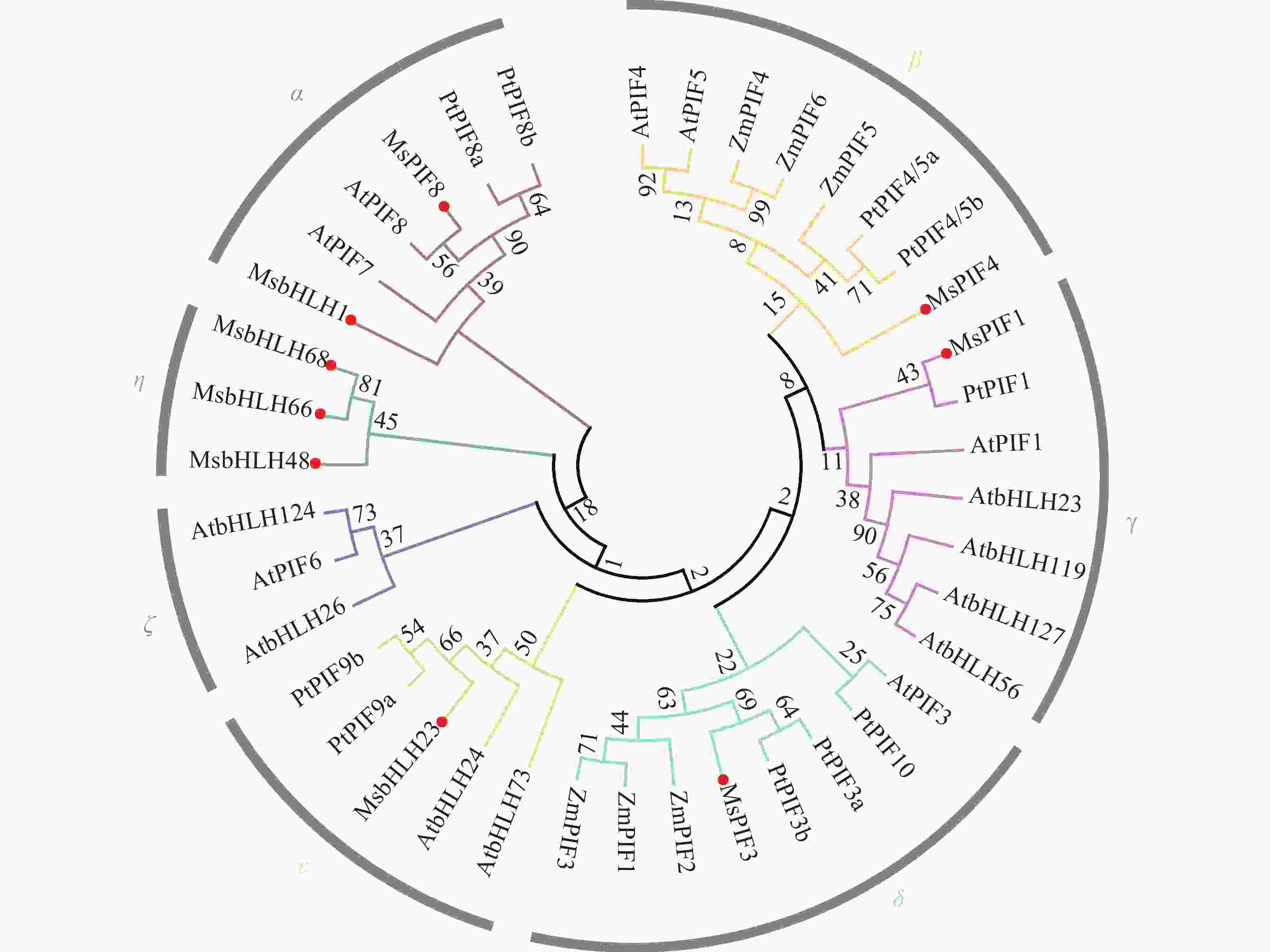
运用MEGA5对所得景宁木兰的蛋白质序列与拟南芥、玉米、杨树中的序列对比分析(图3),采用邻接法及Bootstrap分析(1 000次重复)构建系统进化树[18]。结果表明:植物PIF家族蛋白质表现出多样性,可以分为7个进化分枝。其中,9个MsPIF蛋白质分布在α、β、γ、δ、η、ε等6个进化枝。15个AtPIF蛋白质分布在α、β、γ、δ、ζ、ε等6个进化枝,其中ζ为拟南芥所特有。ZmPIF蛋白质仅分布于β和δ进化枝,10个PtPIF蛋白质分布在α、β、γ、δ、ε等5个进化枝。从进化树可以看出,MsPIF8、MsPIF4、MsPIF1、MsPIF3及MsbHLH23均与相应的拟南芥蛋白质与杨树蛋白质聚类在一起,表明其亲缘关系较为接近。MsbHLH68、MsbHLH66、MsbHLH48汇聚于η枝,为景宁木兰所特有。
-
光照是植物生长发育过程中不可或缺的因素,极端遮阴处理过程中,景宁木兰叶片表型变化十分明显(图4)。处理10 d,景宁木兰叶片出现褐色斑点;处理20 d,褐色斑点面积增大,植株开始萎蔫;处理30 d,大量叶片干枯脱落,植株萎蔫严重。
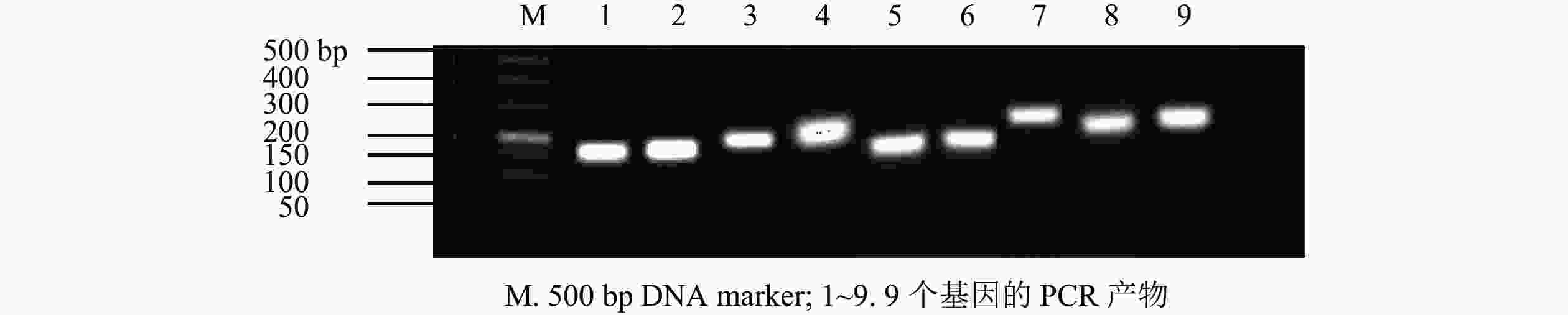
PCR扩增琼脂糖凝胶电泳检测发现9个基因均扩出预期条带(图5)。qRT-PCR检测结果(图6)表明:遮阴处理1 d时,MsbHLH23和MsbHLH66的表达水平显著上调,MsbHLH1、MsbHLH48、MsPIF8、MsPIF3的表达水平均显著下调,其余基因表达水平无显著变化。遮阴处理3 d时,MsPIF8的表达显著低于对照,其余基因的表达均显著上调。遮阴处理5 d时,MsPIF8的表达仍显著降低,MsbHLH68、MsbHLH23、MsbHLH66、MsbHLH1、MsbHLH48的表达显著上调,其余基因的表达无显著差异。遮阴处理10 d时,MsPIF8、MsPIF7、MsPIF3的表达均显著下调,MsbHLH68、MsbHLH23、MsbHLH66、MsPIF4的表达显著上调。遮阴处理20 d时,MsPIF8、MsbHLH23、MsPIF1的表达相对于对照显著下调,MsbHLH68和MsbHLH66的表达显著上调,其余基因无显著差异。遮阴处理30 d时,MsPIF8、MsbHLH23的表达相对于对照显著下调,MsbHLH68、MsbHLH1、MsbHLH66、MsbHLH48、MsPIF3的表达显著上调。其中,MsbHLH23的表达变化比其他基因更为明显,遮阴处理5和10 d时的表达量分别上调为对照的52.77与20.03倍。
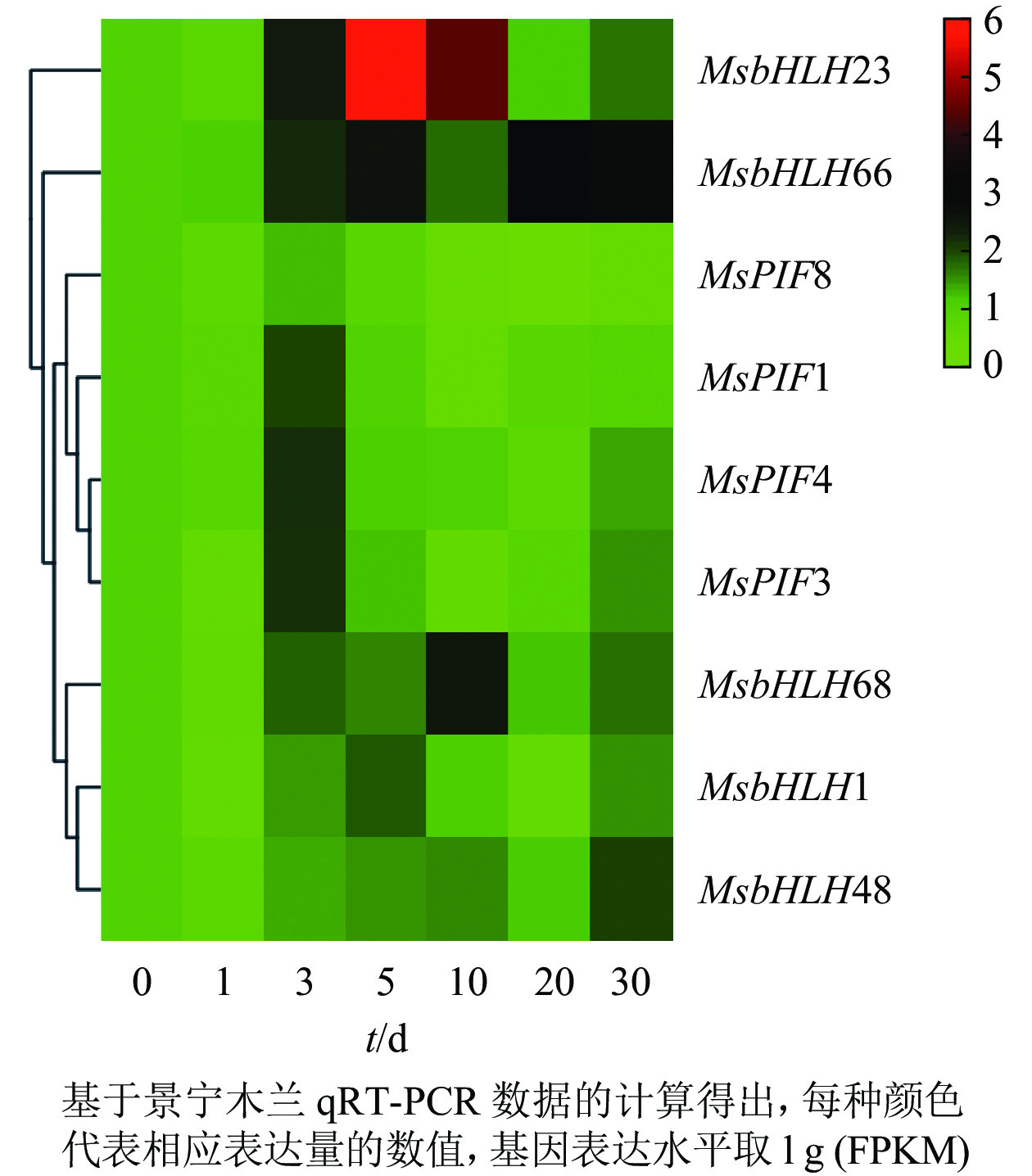
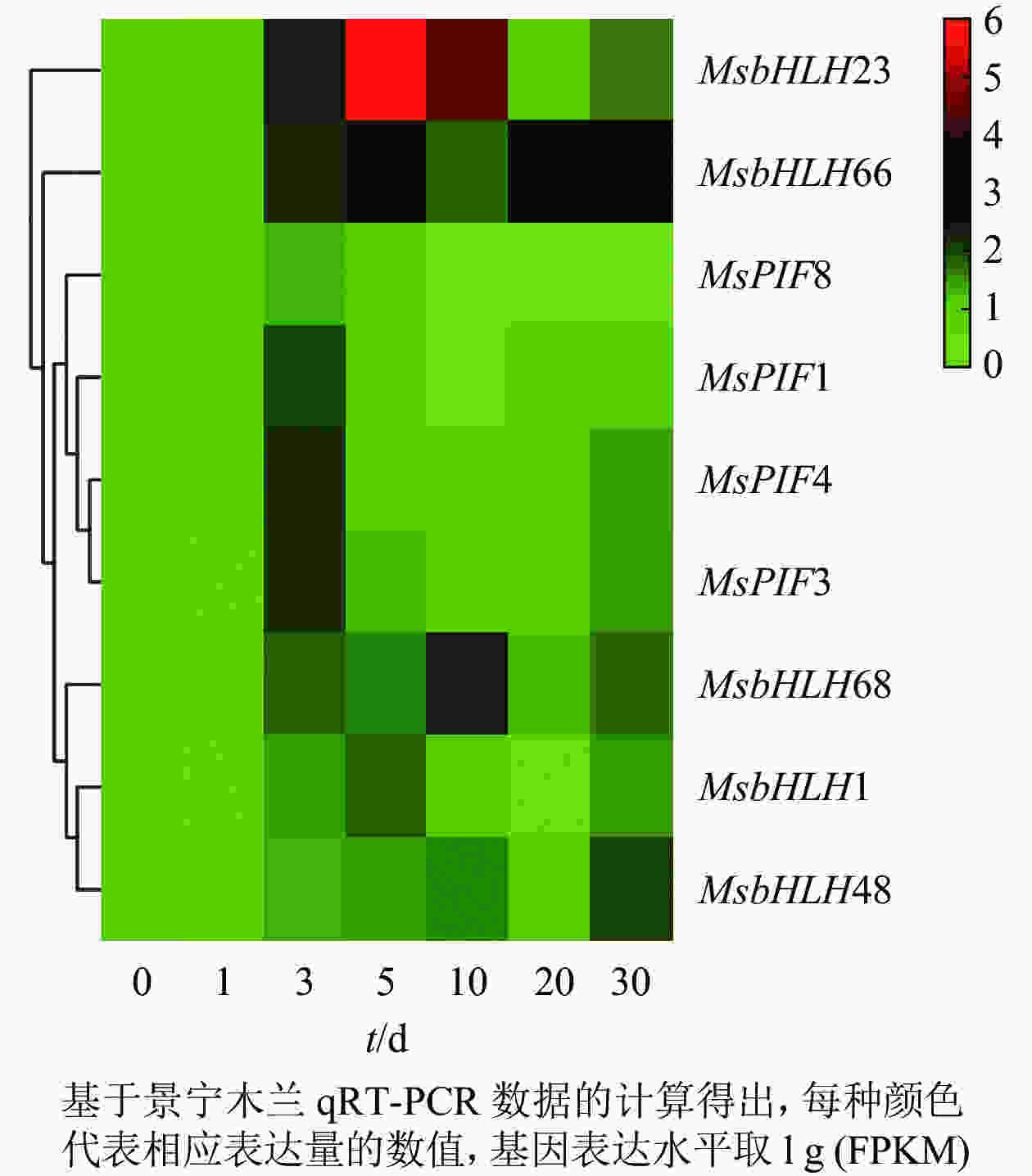
基因的表达模式与基因功能关系密切,基于获得的景宁木兰处理组的qRT-PCR数据,采用heatmap在线软件对这些基因的差异表达进行分层聚类分析(图7)。结果发现:MsPIF1、MsPIF3、MsPIF4、MsPIF8的表达模式十分相似,其表达均在处理3 d时明显升高,随后逐渐下降。MsbHLH48的表达在处理0~30 d逐渐上调,其中处理30 d时的表达上调最为明显。MsbHLH1、MsbHLH68、MsbHLH66、MsbHLH23的表达模式均为先逐渐上调后逐渐下调,其中MsbHLH1和MsbHLH23的表达在处理5 d显著上调,MsbHLH68和MsbHLH66的表达分别在处理10和20 d明显上调。
2.1. 序列鉴定及理化性状分析
2.2. 蛋白质亚细胞定位预测
2.3. 蛋白质磷酸化位点分析
2.4. 蛋白质的基序分析
2.5. 系统进化分析
2.6. 长期极端遮阴条件下MsPIF家族基因的表达
-
PIF家族转录因子广泛存在于植物中。目前PIF家族转录因子在拟南芥中研究的最为透彻,共有15个[5]。在其他物种中,杨树至少存在10个[13],玉米有6个[12],水稻Oryza sativa有6个[19]。本研究从景宁木兰中仅筛选到9个,这一方面是由于景宁木兰本身具有特异性,另一方面可能是因为转录组数据库还不够丰富造成的。NCBI blast及NCBI CDD预测结果表明:9个PIF蛋白质均含bHLH结构域,属于PIF转录因子家族。同源性较高的蛋白质通常功能也相似[18]。因此,本研究用所得9个景宁木兰PIF家族转录因子编码蛋白质与6个ZmPIFs、10个PtPIFs 和15个功能明确的AtPIFs编码蛋白质作系统进化树,主要根据与15个拟南芥PIF家族转录因子编码蛋白质进化关系的远近推测景宁木兰PIF转录因子的功能。AtPIF3与PtPIF3a、PtPIF3b同源性较高,且与AtPIF3和ZmPIF3位于同一进化枝,据此推测MsPIF3与AtPIF3功能相似。MsPIF4与PtPIF4、ZmPIF4、AtPIF4位于同一进化枝。AtPIF3蛋白质在植株刚刚暴露到光下时会急剧减少,在黑暗条件下会重新积累,在拟南芥幼苗阶段,AtPIF3在光诱导的叶绿体发育中起到积极的作用[20]。AtPIF3、AtPIF4和AtPIF5在年龄引起的和黑暗引起的植物衰老中起到重要作用,其中AtPIF4通过调控叶绿素降解和维持叶绿体活性的相关基因从而促进植株衰老[7]。AtPIF4能够调控下游基因从而促进拟南芥开花[21]。
此外,AtPIF4能与AtBZR1共同调节下游光响应的相关基因。AtPIF7与MsbHLH1同源性较高,推测其功能可能类似。遮阴条件下,AtPIF7是调控拟南芥茎伸长的重要因子[17]。AtPIF7是调节植物去黄化的弱负反馈因子,与PhyB结合的过程中通常不伴随着可以监测到的磷酸化和分解[22]。MsPIF8与AtPIF8同源,推测其功能相似。与AtPIF3不同,AtPIF8蛋白质在远红光下的积累比在黑暗条件中或红光条件下更多,AtPIF8能够抑制由PhyA介导的种子萌发、下胚轴伸长等生命活动[23]。MsbHLH23与AtbHLH24功能相似,AtbHLH24在抑制种子萌发和维持种子休眠状态中具有重要作用[24]。AtbHLH24能够激活细胞分裂素信号,同时激活生长素生物合成和雄蕊内部结构域的转运基因,并且在花瓣和果实发育中起到重要作用[25]。MsbHLH68,MsbHLH66和MsbHLH48共同汇集于同一进化枝,其功能还需要进一步挖掘。但是,即使是同一分支内蛋白质功能也可能存在差异,并且不同物种之间具有较大的差异[18]。因而关于MsPIF家族蛋白质的具体功能还需要通过后续研究。
-
群落层片造成的遮阴会降低下层植被的生长率与成活率[26]。本研究极端遮阴处理组的景宁木兰在处理过程中逐步萎蔫死亡。光敏色素作用因子(PIFs)在植物的生长发育中起到重要调节作用。PIFs作为光信号转导途径中的负调控因子,对植物生长发育的多个过程都有调控作用[27-32]。PIF家族转录因子是光敏色素介导的光信号转导途径中的负反馈因子[28]。遮阴条件下,PIF蛋白质大量积累,从而调控植物发生一系列生理生化反应。考虑到植物的个体差异和基因表达差异,本研究的每个取样时间点都作了相应的空白对照。本研究中,长期极端遮阴条件下,景宁木兰的PIF类家族转录因子的表达水平多数显著上调,只有MsPIF8的表达水平显著下调。在拟南芥中,与其他PIF家族成员相比,AtPIF8能够与PhyB结合但效果微弱[23]。MsPIF8的表达模式与前人的研究结论存在差异,可能与物种特异性相关,有关MsPIF8的功能还需要进一步研究。MsPIF1、MsPIF3和MsPIF4的表达模式与前人的研究结论[29]吻合。黑暗条件下,AtPIF1的表达上调,从而抑制叶绿素的合成与种子萌发;AtPIF3、AtPIF4的表达显著上调,从而促进叶绿素的降解与叶片衰老[29]。植物群落造成的遮阴使得AtPIF4的表达上调,从而使成花诱导相关基因的表达上调,最终诱导拟南芥提早开花[21]。PIF功能十分复杂,景宁木兰PIF家族转录因子在遮阴调控网络中发挥的作用还需要进一步研究。













 DownLoad:
DownLoad: