-
苦荞Fagopyrum tataricum是一种既可食用又可药用的粮食作物,喜旱怕水,在中国西南部高海拔地区广泛种植[1−2],其籽粒含有抗性淀粉(RS)和γ-氨基丁酸(GABA)等功能性成分,对预防和治疗慢性疾病具有特殊功效和巨大药用价值,有较高的营养和医疗保健价值[3],成为功能性食品开发和研究的热点之一[4]。RS主要指不能被健康人体小肠所吸收的部分淀粉及其降解产物的总和[5]。食用富含RS的食物有助于增加饱腹感,减少热量摄入[6],且引起的血糖波动较小,有助于控制血糖浓度。对于苦荞中RS的研究主要集中在RS的制备,如利用高压糊化、多次湿热处理等制备苦荞RS[7]。淀粉合成酶(SS)主要有颗粒结合淀粉合成酶(GBSS)和可溶性淀粉合成酶(SSS) 2种,是淀粉生物合成过程中的关键酶之一[8],WANG等[9]从苦荞基因组中鉴定了15个SS基因家族成员,对其分子特征进行了分析,WANG等[10]分析了苦荞淀粉颗粒结合酶基因(FtGBSSI)完整的基因组序列。GABA是一种广泛分布于动物、植物和微生物体内的非蛋白氨基酸[11],可作为应激反应信号参与植物的抗逆性调节,也可作为神经递质,参与动物神经元的兴奋和抑制调节,帮助改善睡眠,缓解抑郁和焦虑[11−13]。外源摄入GABA还具有降血脂、预防肥胖症等功能[14−15]。对于苦荞中GABA的研究主要集中在富集、功能性食品应用等方面,张钰等[16]利用柠檬酸和亚精胺联合处理提高了苦荞芽中GABA的含量,CHEN等[17]使用冷常压等离子体促进苦荞籽粒中GABA的积累。
数量性状位点(QTL)定位是揭示复杂数量性状和鉴定候选基因的有效植物育种方法[18−19]。在不同的遗传背景下挖掘QTL对植物育种具有重要价值。RS和GABA作为植物重要的功能性成分,挖掘与其相关的QTL,了解其遗传基础对提高作物产量和优化作物品质具有重要意义。近年对RS和GABA的QTL研究多集中在水稻Oryza sativa等[20]农作物中,目前还没有对苦荞籽粒中RS和GABA的QTL报道。
本研究以父本‘晋荞麦2号’‘Jinqiaomai No. 2’、母本‘小米荞’‘Xiaomiqiao’及其构建的重组自交系(XJ-RILs)群体为供试材料,基于黎瑞源等[21]构建的遗传图谱,对XJ-RILs群体的RS和GABA 2个功能成分进行QTL定位研究,以期鉴定控制苦荞RS和GABA的关键QTL,通过生物信息学分析找到相关的候选基因,探索这2个功能成分在苦荞中的遗传基础,为苦荞品种遗传改良及精确定位RS和GABA含量相关的基因提供基础材料。
-
父本‘晋荞麦2号’、母本‘小米荞’及其构建的XJ-RILs群体于2022年种植于贵州师范大学荞麦产业技术研究中心安顺基地,常规大田管理。成熟期收获籽粒,在30 ℃烘箱中烘干后剥壳,磨粉,存放于4 ℃冰箱备用。取‘晋荞麦2号’和‘贵米苦11号’‘Guimiku No. 11’根、茎、叶、花、籽粒等不同部位组织,用液氮速冻后存放于−80 ℃冰箱备用。
-
参考张焕新[22]的方法,略有改动,试剂购自伯远生物。准确称取0.100 0 g 样品,添加3 mL HCl-KCl (pH 2~3)缓冲液,加入1 mL胃蛋白酶,60 ℃水浴振荡1.0 h。取出冷却后用HCl和NaOH调pH至5.4,加入2 mL α-淀粉酶,90 ℃水浴振荡1.5 h。样品冷却至室温整pH为4.1~4.3,加入1 mL葡萄糖淀粉酶,60 ℃水浴振荡2.0 h。加入10 mL无水乙醇,10 000 r·min−1离心2 min;弃去上清液,沉淀用80%乙醇洗3遍;加入2 mL KOH溶解沉淀,调整pH至4.1~4.3,加入1 mL葡萄糖淀粉酶,60 ℃水浴振荡1.0 h;沸水浴5 min,10 000 r·min−1离心2 min,收集上清液,加3 mL ddH2O洗涤沉淀2次,上清液合并,定容至15 mL(待测样)。使用二硝基水杨酸(DNS)法在540 nm处测定吸光度[D(540)],根据公式计算RS质量分数:RS质量分数=(葡萄糖质量×0.9/样品淀粉质量)×待测样体积×100%。
-
参考王传蔚[23]的方法,略有改动,试剂购自伯远生物。准确称取0.100 0 g样品,加入4 mL甲醇,室温振荡10 min;5 000 r·min−1离心15 min,弃上清液;重复上述甲醇处理步骤1次,待沉淀完全干燥后,加入4 mL ddH2O,40 ℃水浴振荡10 min,5 000 r·min−1离心15 min,取上清液备用;重复上述步骤1次,合并上清液,定容至10 mL。取2 mL溶液加入0.8 mL 0.5 mol·L−1 AlCl3,室温振荡5 min,12 000 r·min−1离心5 min,弃沉淀;加入1.2 mL的10.5 mol·L−1 KOH,室温振荡5 min,12 000 r·min−1离心5 min,取上清液备用;重复上述步骤,直至将10 mL提取液全部处理完毕,合并所有得到的上清液;加入4 mL的四硼酸钠缓冲液(pH 8.0)和1.4 mL的质量分数为6%苯酚溶液;缓慢滴加2.5 mL质量分数为5%NaCIO溶液,水浴20 min;定容至50 mL后在625 nm处测定吸光度[D(625)]。根据公式计算样品GABA质量分数:GABA质量分数=待测液中总GABA质量浓度×待测液的体积×样品的总稀释倍数/(样品的质量×106)×1 000‰。
-
使用WinQTLCart 2.5的复合区间作图法(CIM),设置对数优势比(LOD)为2.5,对XJ-RILs群体进行QTL分析,采用“q+性状英文名缩写+所在染色体编号+序列号”对QTL位点进行命名[24]。
-
基于XJ-RILs群体的SNP遗传图谱定位到控制RS和GABA相应的物理区间[25],将各QTL物理区间对应苦荞‘品苦1号’‘Pinku No. 1’参考基因组(https://www.mbkbase.org/Pinku1/)以获得QTL区间内基因及注释信息[26]。
-
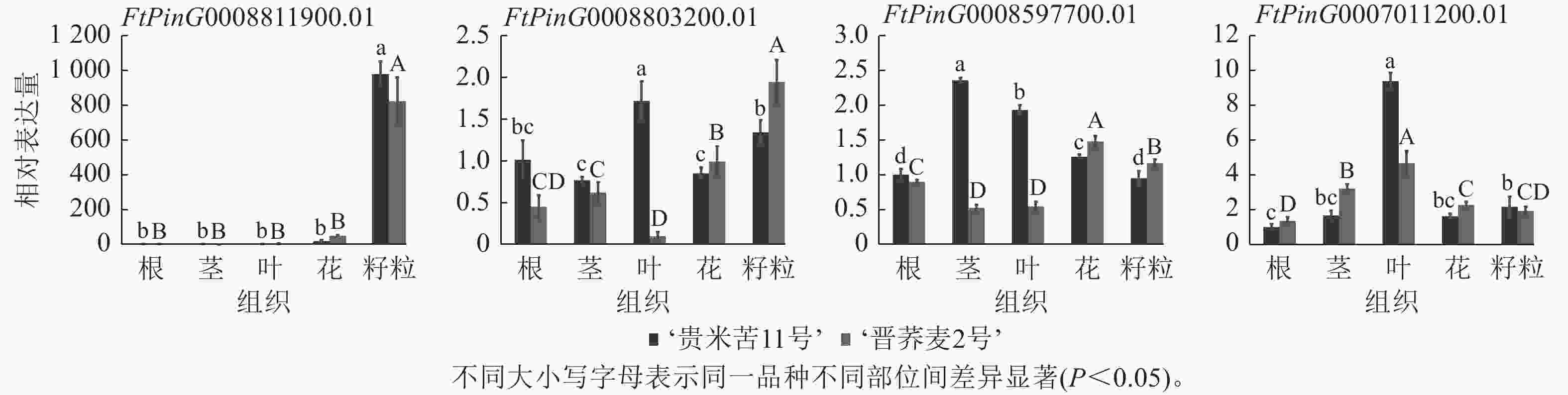
使用RNA Easy Fast植物组织RNA快速提取试剂盒(天根生化公司)提取‘贵米苦11号’和‘晋荞麦2号’不同组织的RNA,使用PrimeScriptTMRT Master Mix(TaKaRa生物公司)反转录成cDNA,选择了FtPinG0008597700.01、FtPinG0007011200.01、FtPinG0008803200.01和FtPinG0008811900.01等4个候选基因用Primer3 Plus设计RT-qPCR引物,由生工生物工程有限公司合成(引物序列见表1),内参基因为FtActUniv。以‘贵米苦11号’和‘晋荞麦2号’不同组织的cDNA为模板,按照Eastep qPCR Mix(2x)试剂盒(普洛麦格生物公司)说明书进行RT-qPCR。采用2−∆∆Ct法计算候选基因的表达量。
表 1 RT-qPCR引物序列
Table 1. Primers used for RT-qPCR analysis
引物 正向引物(5′→3′) 反向引物(5′→3′) q-FtPinG0008597700.01 GCGAACTATGGAGGGCTTGT TCGTGCTCAGCCTGAAAAGT q-FtPinG0007011200.01 CCATCATCAACTGGCTTCGG GCAACAGAATTCACAAGGGACG q-FtPinG0008803200.01 TTCAGAATCCCAAGTCCGGC ATGAGGGGACAGAGACGTCA q-FtPinG0008811900.01 GTTTGTTCAGGTGGCAGTCG CCTGCAAGTAGAAGGGCACA ActUniv GAGTTATGAGCTTCCTGATG CCGCCACTCAACACAATGTT -
利用Excel 2019计算均值和变异系数,采用IBM SPSS Statistics 26计算峰度、偏度、绘制频率分布图和进行各类群间研究性状的方差分析;采用RStudio应用程序作聚类分析,采用iTOL工具绘制聚类图[27]。
-
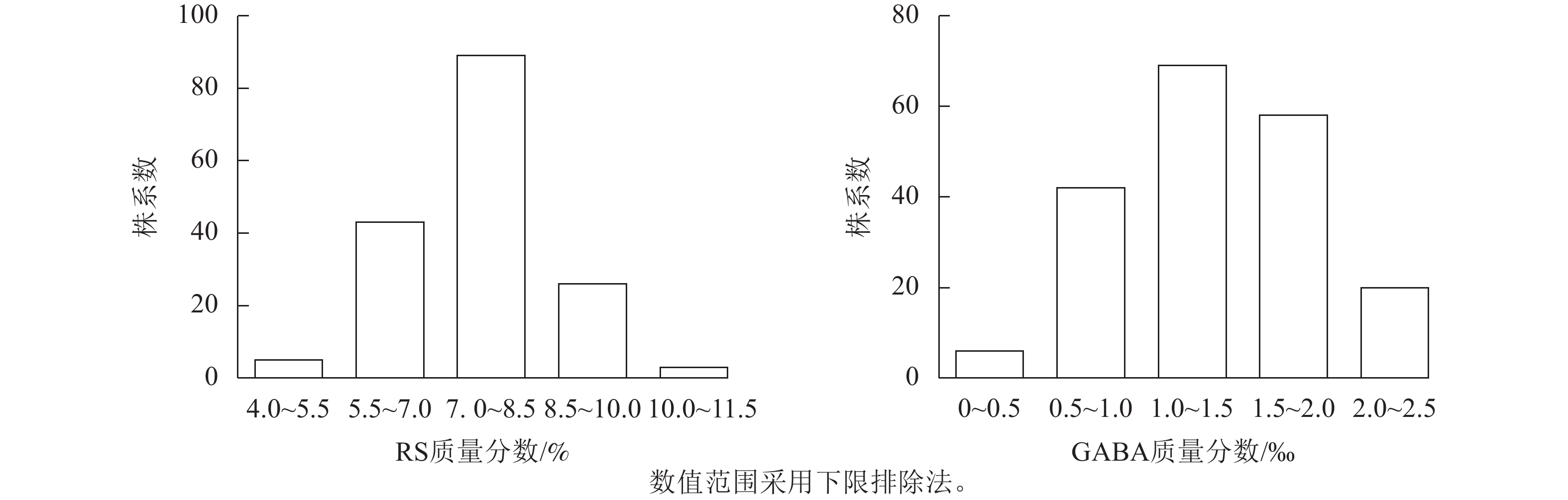
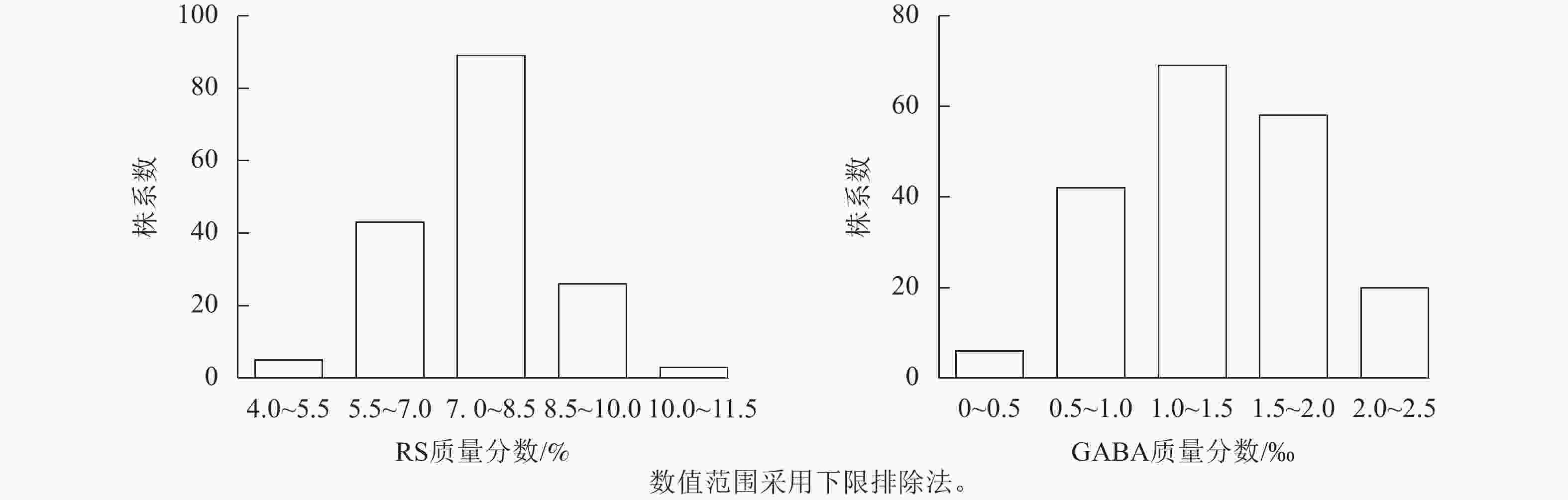
苦荞XJ-RILs群体亲本‘晋荞麦2号’和‘小米荞’RS质量分数分别为8.84%和9.26%,GABA质量分数分别为0.50‰和0.56‰(表2),双亲间差异不显著;XJ-RILs群体RS和GABA质量分数分别为4.51%~10.52%和0.37‰~2.50‰,平均值分别为7.53%和1.35‰,变异系数分别为14.43%和38.21%(表2),均表现为偏正态分布,具超亲优势(图1),偏度分别为0.123和0.018,峰度分别为0.179和−0.875 (表2),表明这2种性状均是由多基因控制的数量性状。
表 2 苦荞XJ-RILs群体以及亲本RS和GABA的描述统计分析
Table 2. Descriptive statistical analysis of RS and GABA in the XJ-RILs population of Tartary buckwheat and the parents
样本 RS GABA 平均值±标准差/% 范围/% 偏度 峰度 变异系数/% 平均值±标准差/‰ 范围/‰ 偏度 峰度 变异系数/% ‘晋荞2号’ 8.84±0.41 0.50±0.15 ‘小米荞’ 9.26±0.35 0.56±0.04 XJ-RILs 群体 7.53±1.09 4.51~10.52 0.123 0.179 14.43 1.35±0.52 0.37~2.50 0.018 −0.875 38.21 -
根据RS和GABA的质量分数对苦荞XJ-RILs群体及其亲本共计153个株系进行聚类分析。结果表明:153个株系被分为5个类群(图2),C1类群包含20个株系,RS平均质量分数为8.78%,GABA平均质量分数为1.99‰,均高于其他4个类群(表3);C2类群包含40个株系,RS平均质量分数为6.89%,GABA平均质量分数为1.83‰;C3类群包含19个株系,较其他4个类群最少,RS质量分数也最低,为6.00%,GABA平均质量分数为1.00‰;C4类群包含34个株系,RS平均质量分数为8.27%,GABA质量分数最低,为0.69‰;C5类群包含40个株系,RS平均质量分数为7.71%,GABA平均质量分数为1.26‰。
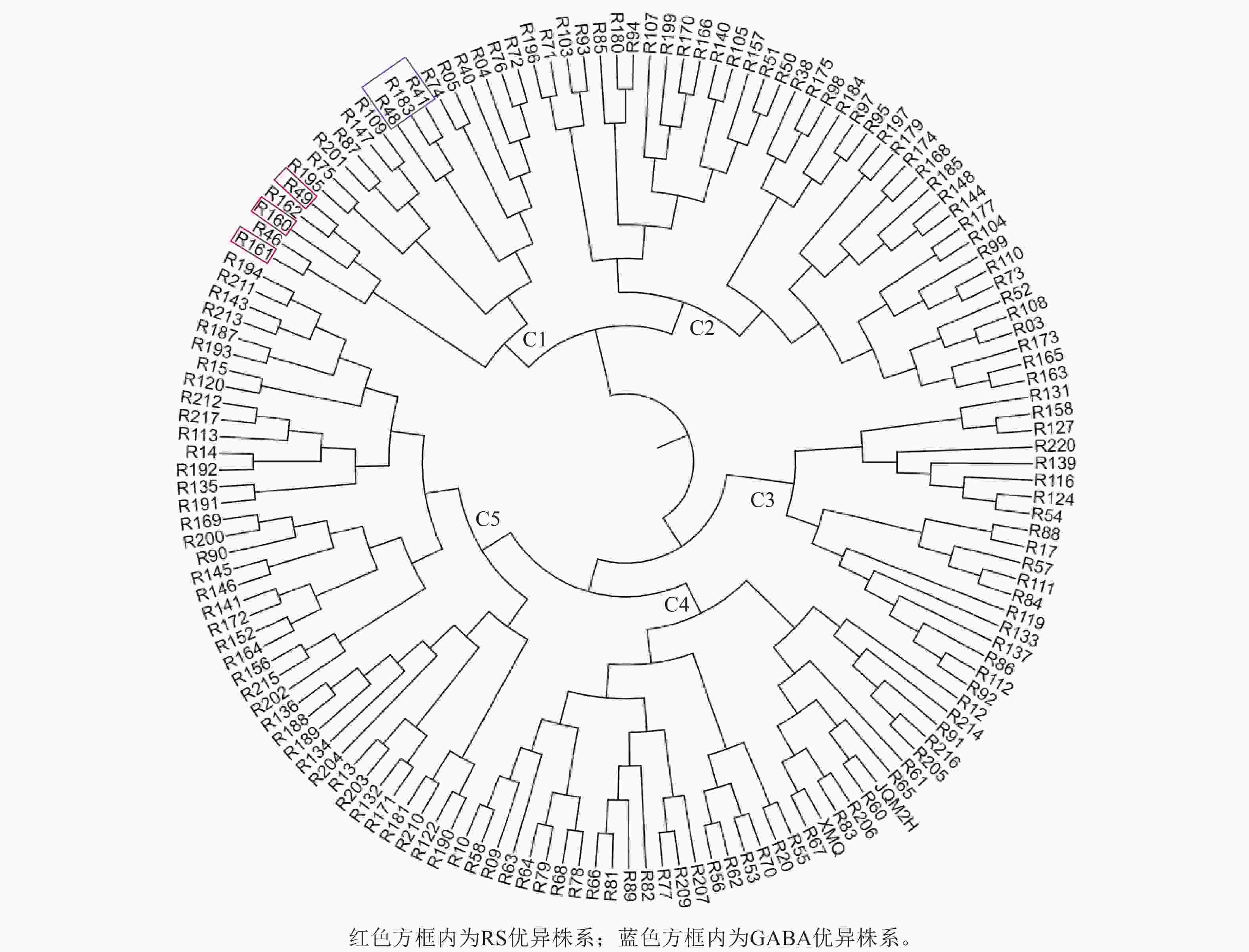
图 2 XJ-RILs群体RS和GABA性状聚类图
Figure 2. Cluster analysis of the XJ-RILs population based on their RS and GABA
表 3 XJ-RILs 群体各类群RS和GABA性状的方差分析
Table 3. ANOVA analysis of RS and GABA among different groups in XJ-RILs population
类群 株系数/个 RS质量分数/% GABA质量分数/‰ C1 20 8.78±0.72 a 1.99±0.27 a C2 40 6.89±0.62 d 1.83±0.20 b C3 19 6.00±052 e 1.00±0.22 d C4 34 8.27±0.97 b 0.69±0.24 e C5 40 7.71±0.46 c 1.26±0.14 c 说明:不同小写字母表示差异显著(P<0.05)。 从XJ-RILs群体中筛选出R49、R160和R161 3个RS优异株系可作为RS高含量的推荐材料,其RS质量分数分别为9.93%、10.52%和9.91% (表4);从XJ-RILs群体中筛选出GABA表达优异的3个株系可作为GABA高含量的推荐材料,即R41、R48和R183,其GABA质量分数分别为2.39‰、2.50‰和2.47‰ (表4)。
表 4 XJ-RILs 群体优异株系的RS和GABA质量分数
Table 4. Contents of RS and GABA in excellent lines of XJ-RILs
株系 RS质量分数/% GABA质量分数/‰ R41 8.20 2.39 R48 8.75 2.50 R49 9.93 2.00 R160 10.52 1.91 R161 9.91 1.73 R183 8.23 2.47 -
对XJ-RILs群体苦荞籽粒RS和GABA进行QTL定位,RS共检测到2个QTL位点,即qRS3-1和qRS4-1;GABA检测到1个QTL位点,即qGABA7-1。qRS3-1位于第3染色体Block5310~Block5518区间,表型贡献率(R2)为7.59%,LOD为3.2;qRS4-1位于第4染色体Block9147~Block9171区间,R2为6.34%,LOD为2.82;qGABA7-1位于第7染色体Block14135~Block14149区间,R2为5.05%,LOD为1.01;其中qRS3-1和qRS4-1来自‘小米荞’,加性效应分别为−0.31和−0.28,qGABA7-1来自于‘晋荞麦2号’,加性效应为0.11 (表5)。
表 5 苦荞XJ-RILs群体RS和GABA的QTL定位
Table 5. QTL mapping of RS and GABA content in the XJ-RILs population of Tartary buckwheat
性状 QTL 染色体 位置/cM LOD R2/% 加性效应 置信区间/cM 标记区间 物理区间/Mb RS qRS3-1 Chr 3 0.39 3.20 7.59 −0.31 38.7~44.0 Block5310~Block5518 12.17~14.26 qRS4-1 Chr 4 1.66 2.82 6.34 −0.28 165.1~168.9 Block9147~Block9171 50.92~51.33 GABA qGABA7-1 Chr 7 1.01 2.52 5.05 0.11 98.9~101.8 Block14135~Block14149 32.352~33.13 说明:LOD. 对数优势比;R2. 表型贡献率。 -
对QTL区段内的候选基因进行检测,qRS3-1、qRS4-1和qGABA7-1分别检测到96、20和70个基因。基于转录组数据,3个QTL位点中有172个基因在苦荞根、茎、叶、花和籽粒等至少一种组织中表达,其表达模式如图3所示。根据RS相关候选基因的表达情况,将在苦荞组织中表达的108个基因聚为6个类群(图3A),即R1~R6。其中,R1类群含17个基因,该类群内基因在花中表达量最高,茎和叶次之,根中相对较低,籽粒中最低;R2类群含23个基因,除FtPinG0004083100.01基因在籽粒中表达量较高外,其余基因在根中表达量最高,茎和叶中次之,籽粒中表达量较低,花中最低;R3类群含20个基因,该类群内基因在叶中表达量最高,在根、茎和花中表达量次之,在籽粒中表达量最低;R4类群含12个基因,除FtPinG0004080900.01和FtPinG0001690000.01基因在籽粒中表达量较低外,其余基因在叶和籽粒中表达量最高,茎和花中表达量次之,根中表达量最低;R5类群含9个基因,该类群内基因在籽粒中表达量最高,在其余各组织中表达量相对较低;R6类群含27个基因,除11个基因在籽粒中表达较高外,其余基因在根和叶中表达量较高,茎和籽粒中次之,花中最低。根据各类群基因在籽粒中的表达情况,R4 (10个)、R5 (9个)和R6 (11个)中的30个基因可能参与籽粒中RS合成。
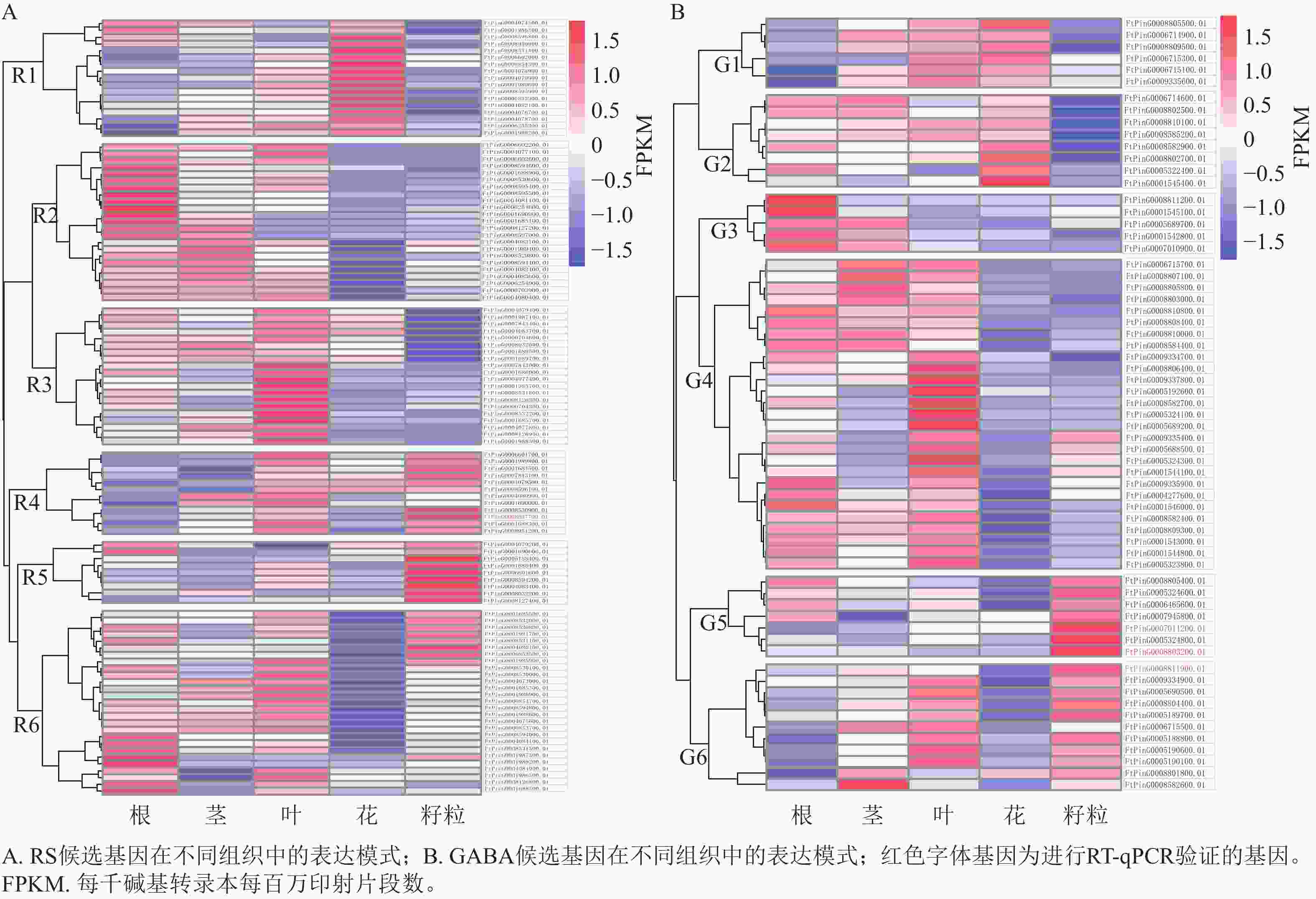
图 3 RS和GABA候选基因在不同组织中的表达模式
Figure 3. The expression patterns of candidate genes controlling RS and GABA in different tissues
根据GABA相关候选基因的表达情况,将在苦荞组织中表达的64个基因划分为6个类群(图3B),即G1~G6。其中,G1类群含6个基因,该类群内基因在花和叶中表达量最高,茎中次之,根和籽粒中最低;G2类群含8个基因,该类群内基因在花中表达量最高,根、茎和叶中次之,在籽粒中表达量最低;G3类群含5个基因,该类群内基因在根中表达量最高,在茎中表达量次之,在叶、花和籽粒中表达量较低;G4类群含27个基因,除FtPinG0009335400.01、FtPinG0005688500.01、FtPinG0005324300.01和FtPinG0001544100.01 这4个基因在籽粒中表达量较高外,其余基因在根和叶中表达量最高,茎中次之,在花和籽粒中较低;G5类群含7个基因,该类群内基因在籽粒中表达量最高,根中次之,茎、叶和花中均较低;G6类群含11个基因,除FtPinG0006715500.01在籽粒中表达量较低外,其余基因在籽粒中表达量最高,叶中次之,其次在茎中表达量也较高,在根和花中表达量最低。根据各类群基因在籽粒中的表达情况,G4 (4个)、G5 (7个)、G6 (10个)中的21个基因可能参与籽粒中GABA的合成。
-
为了确认基于苦荞RNA-seq所作QTL区段内基因组织特异性的准确性,选择了FtPinG0008597700.01、FtPinG0007011200.01、FtPinG0008803200.01和FtPinG0008811900.01等4个在籽粒中高表达的候选基因进行RT-qPCR验证(图4)。基于‘品苦1号’转录组数据对候选基因所作表达模式分析与实际表达情况存在差异,FtPinG0008811900.01在2个品种的不同组织中表达模式与转录组一致,均在籽粒中表达量最高;FtPinG0008803200.01在‘晋荞麦2号’的不同组织中表达量基本与转录组数据一致,但在‘贵米苦11号’的不同组织中表达情况与转录组存在较大差异;FtPinG0008597700.01和FtPinG0007011200.01基因在2个品种中的实际表达量与转录组表达情况均存在差异,但与转录组数据一致的是4个基因在籽粒中均高表达。可见,基因的表达情况会受到遗传背景和环境因素的影响,但基于转录组在籽粒中高表达的基因实际也在籽粒中不同程度表达,参与籽粒中RS和GABA的合成调控。
-
基于XJ-RILs群体的SNP遗传图谱[25],对筛选的候选基因进行变异位点分析。表6结果表明:qRS3-1、qRS4-1和qGABA7-1中有22个基因存在变异,均只有1个变异位点。qRS3-1中有7个基因存在SNP变异,5个存在Indel变异,其中有4个基因功能未得到注释,其余8个基因分别得到注释:Protein NLP (FtPinG0008950900.01)、SWEET sugar transporter (FtPinG0008951200.01)、Galactose-binding-like (FtPinG0008595500.01)、Homeobox (FtPinG0004083400.01)、Phosphoribosyltransferase C-terminal (FtPinG0001685300.01)、Protein kinase-like (FtPinG0008594800.01)、Protein DMP (FtPinG0008594400.01)和P-type ATPase (FtPinG0001685100.01)。
表 6 候选基因的SNP/Indel变异位点
Table 6. Variant SNP/Indel sites of candidate genes
序号 基因 QTL 染色体 变异类型 变异位点信息
(‘小米荞’/‘晋荞麦2号’)基因注释 1 FtPinG0001690600.01 qRS3-1 Chr 3 SNP A/G 2 FtPinG0008950900.01 qRS3-1 Chr 3 SNP T/A NLP蛋白 3 FtPinG0008951200.01 qRS3-1 Chr 3 SNP T/A SWEET糖转运蛋白 4 FtPinG0008595500.01 qRS3-1 Chr 3 SNP A/T 半乳糖结合蛋白 5 FtPinG0004079200.01 qRS3-1 Chr 3 SNP G/A 6 FtPinG0004083400.01 qRS3-1 Chr 3 SNP A/G 同源框基因 7 FtPinG0001685300.01 qRS3-1 Chr 3 SNP G/A 磷酸核糖基转移酶 8 FtPinG0008594800.01 qRS3-1 Chr 3 Indel G/GT 蛋白激酶 9 FtPinG0008594400.01 qRS3-1 Chr 3 Indel CTTTTTA/C DMP蛋白 10 FtPinG0008127400.01 qRS3-1 Chr 3 Indel C/CA 11 FtPinG0008127200.01 qRS3-1 Chr 3 Indel A/AC 12 FtPinG0001685100.01 qRS3-1 Chr 3 Indel A/AATCAT P型ATP酶 13 FtPinG0001989400.01 qRS4-1 Chr 4 SNP G/A 14 FtPinG0001991700.01 qRS4-1 Chr 4 SNP T/C 核糖体蛋白L7Ae 15 FtPinG0006853900.01 qRS4-1 Chr 4 SNP G/A 硫氧还原蛋白类超家族 16 FtPinG0006854700.01 qRS4-1 Chr 4 Indel T/TG 细胞色素b5 17 FtPinG0009335600.01 qGABA7-1 Chr 7 Indel AT/A 亲环素类肽基脯氨酸顺反异构酶 18 FtPinG0008807100.01 qGABA7-1 Chr 7 Indel TC/T 泛醇-细胞色素c还原酶 19 FtPinG0008810800.01 qGABA7-1 Chr 7 Indel AATAT/A B-box型锌指结构 20 FtPinG0008811900.01 qGABA7-1 Chr 7 Indel G/GT 质体蓝素 21 FtPinG0006715500.01 qGABA7-1 Chr 7 Indel T/TATAATAATAATAATAATA 异戊二烯合成酶 22 FtPinG0005188800.01 qGABA7-1 Chr 7 Indel CTTGTTAAAGAAGTTTTG/C qRS4-1中有4个基因存在变异,3个基因存在SNP变异,1个基因存在Indel变异。这些基因中只有1个基因未得到注释,其余3个基因分别得到注释:Ribosomal protein L7Ae (FtPinG0001991700.01)、Thioredoxin-like superfamily (FtPinG0006853900.01)、Cytochrome b5-like heme (FtPinG0006854700.01)。
qGABA7-1中6个基因均为Indel变异,其中只有1个基因未得到注释,其余分别得到注释:Cyclophilin-type peptidyl-prolyl cis-trans isomerase (FtPinG0009335600.01)、Ubiquinol-Cytochromec Reductase (FtPinG0008807100.01)、B-box-type zinc finger (FtPinG0008810800.01)、Phytocyanin (FtPinG0008811900.01)和Isoprenoid synthase (FtPinG0006715500.01)。
-
本研究XJ-RILs群体中RS质量分数为4.51%~10.52%,低于周一鸣等[28]测得的RS范围(7.00%~35.00%),但也有研究表明苦荞中RS质量分数大于4.00%[29],与本研究所测结果相符合。郑发英等[30]研究表明不同提取方法得到的RS质量分数差异显著,因此推测可能是受到所用材料和RS提取方法的影响。不同品种的荞麦中,GABA的质量分数存在差异,本研究所测GABA质量分数为0.37‰~2.50‰,低于张余等[31]所测不同地方品种荞麦中GABA的质量分数(0.98‰~1.68‰);也低于朱丽伟等[32]所测多年生荞麦中GABA的质量分数(1.439‰~3.968‰);而邹亮等[33]所测的‘米荞1号’的GABA质量分数为0.048‰,低于本研究所测‘小米荞’(0.50‰),推测可能是GABA合成相关基因在不同品种荞麦中存在差异表达导致,RT-qPCR验证实验也证明了同一基因在不同品种的苦荞中存在差异表达,因而在不同品种的苦荞中GABA的质量分数存在较大差异。
聚类分析作为评价种质资源的重要手段,可以为资源的挖掘利用提供参考[34]。本研究通过聚类分析筛选到了苦荞XJ-RILs群体中RS优异的3个株系(R49、R160和R161),以及GABA优异的3个株系(R41、R48和R183),筛选到的株系可用于苦荞品种的遗传改良,也可作为高RS和GABA的选育材料。
本研究基于QTL定位预测到控制苦荞籽粒中RS和GABA的候选基因,在所定位筛选到的候选基因中,FtPinG008951200.01注释到SWEET家族基因,SWEET家族是一类糖转运蛋白,可以协助糖类顺浓度梯度运输[35]。本研究中该基因存在SNP变异,即A变异为T,在籽粒中高表达,推测该基因可以在苦荞籽粒发育的过程中协助蔗糖运输,为籽粒发育提供碳素营养。张计育等[36]的研究也表明SWEET家族内基因与籽粒中淀粉积累有关,LI等[37]研究了21个SWEET基因,其中有7个在籽粒发育的糖运输和从糖到淀粉的转化中起着重要作用。FtPinG0008807100.01注释到UQCR (ubiquinol-cytochromec reductase)家族内基因,UQCR是一种多亚基跨膜复合物,是线粒体电子传递链的一部分,参与ATP生成[38],而GABA的合成和代谢也与细胞内能量代谢相关,推测其可能参与GABA合成过程中能量代谢的调控。FtPinG0008811900.01注释到PC (phytocyanin),PC是一类铜电子转移蛋白,铜电子可作为GABA合成过程中的催化剂[39-40],RT-qPCR结果也显示FtPinG0008811900.01基因在籽粒中高表达,表明该基因调控了苦荞籽粒中GABA的合成。
-
本研究筛选到了RS高含量的株系(R49、R160和R161)以及GABA高含量的株系(R41、R48和R183)。在3号和4号染色体上定位到了2个RS的QTL位点,在7号染色体上定位到了1个GABA的QTL位点。对QTL区段内的候选基因进行检测,qRS3-1、qRS4-1和qGABA7-1分别检测到96、20和70个基因,从这些基因中初步筛选出30个调控RS的候选基因,21个调控GABA的候选基因,22个存在SNP/Indel变异的基因。本研究可为高RS和高GABA苦荞分子育种奠定基础,并为高RS和高GABA苦荞育种提供候选材料。
QTL mapping and candidate gene analysis of RS and GABA based on the recombinant inbred lines (RILs) of Fagopyrum tataricum
-
摘要:
目的 抗性淀粉(RS)和γ-氨基丁酸(GABA)是苦荞Fagopyrum tataricum功能性成分,对苦荞籽粒功能成分进行数量性状位点(QTL)定位对苦荞分子遗传改良具有重要意义。 方法 以‘小米荞’‘Xiaomiqiao’和‘晋荞麦2号’‘Jinqiaomai No. 2’为亲本构建的重组自交系(XJ-RILs)群体作为供试材料,测定了XJ-RILs群体及其亲本的RS和GABA质量分数;基于遗传图谱定位控制RS和GABA的QTL,并对QTL区间内的候选基因进行鉴定和分析。 结果 XJ-RILs群体RS和GABA质量分数分别为4.51%~10.52%和0.37‰~2.50‰,表现出明显的超亲优势和偏正态分布。根据RS和GABA质量分数将XJ-RILs分为5类(C1~C5),其中C1类株系的RS和GABA质量分数均较高,从中筛选出高RS和高GABA优异株系各3株。对XJ-RILs群体的RS和GABA进行QTL定位,共检测到2个控制RS的QTL和1个控制GABA的QTL,这3个位点表型贡献率分别为7.59%、6.34%和5.05%。对QTL内的基因进行鉴定,共有108个RS候选基因,将其表达模式分为6类(R1~R6),其中R4、R5和R6中的30个基因在籽粒中的表达量较高。共鉴定到64个GABA候选基因,其表达模式可以分6类(G1~G6),其中G4、G5和G6中的21个基因在籽粒中的表达量较高。在RS和GABA候选基因中,分别有16和6个基因存在SNP/Indel变异。 结论 筛选出高RS和高GABA优异株系各3株,定位到2个控制RS和1个控制GABA的QTL位点,其区间内分别筛选到108和64个基因,初步锁定30个RS相关及21个GABA相关候选基因。图4表6参40 -
关键词:
- 苦荞 /
- 数量性状位点(QTL) /
- 抗性淀粉(RS) /
- γ-氨基丁酸(GABA) /
- 候选基因
Abstract:Objective Tartary buckwheat (Fagopyrum tataricum), as a high-quality minor grain crop, contains a variety of functional components, such as resistant starch (RS) and γ-aminobutyric acid (GABA). Quantitative trait locus (QTL) mapping of functional components in Tartary buckwheat grains is of great significance for molecular genetic improvement of Tartary buckwheat. Method The recombinant inbred lines population constructed by using ‘Xiaomijiao’ and ‘Jinqiaomai No. 2’ as parents (XJ-RILs) was used as the test material. The contents of RS and GABA of the XJ-RILs population and its parents were determined. Based on the genetic map, QTLs controlling RS and GABA were located, and candidate genes within the QTL intervals were identified and analyzed. Result The XJ-RILs population exhibited significant transgressive segregation and a skewed normal distribution for RS and GABA contents, ranging from 4.51% to 10.52% and 0.37‰ to 2.50‰, respectively. Based on their contents, the population was classified into five categories (C1 to C5), among which the C1 category had high levels of both RS and GABA, and three superior lines with high RS and three with high GABA were selected. QTL mapping for RS and GABA in the XJ-RILs population detected a total of two QTLs controlling RS and one controlling GABA, with phenotypic contribution rates of 7.59%, 6.34%, and 5.05%, respectively. A total of 108 candidate genes for RS were identified within the QTL regions, and their expression patterns were classified into six categories (R1 to R6). Among them, 30 genes in R4, R5, and R6 showed higher expression levels in seeds. Similarly, 64 candidate genes for GABA were identified and their expression patterns were classified into six categories (G1 to G6), with 21 genes in G4, G5, and G6 exhibiting higher expression in seeds. Among the candidate genes for RS and GABA, 16 and 6 genes, respectively, harbored SNP/Indel variations. Conclusion Three elite lines with high RS and three with high GABA were selected. Two QTL loci controlling RS and one QTL locus controlling GABA were identified. Within these loci intervals, 108 and 64 genes were screened respectively, with 30 RS-related and 21 GABA-related candidate genes preliminarily identified . [Ch, 4 fig. 6 tab. 40 ref.] -
Key words:
- Tartary buckwheat /
- quantitative trait locus /
- resistant starch /
- γ-aminobutyric acid /
- candidate gene
-
表 1 RT-qPCR引物序列
Table 1. Primers used for RT-qPCR analysis
引物 正向引物(5′→3′) 反向引物(5′→3′) q-FtPinG0008597700.01 GCGAACTATGGAGGGCTTGT TCGTGCTCAGCCTGAAAAGT q-FtPinG0007011200.01 CCATCATCAACTGGCTTCGG GCAACAGAATTCACAAGGGACG q-FtPinG0008803200.01 TTCAGAATCCCAAGTCCGGC ATGAGGGGACAGAGACGTCA q-FtPinG0008811900.01 GTTTGTTCAGGTGGCAGTCG CCTGCAAGTAGAAGGGCACA ActUniv GAGTTATGAGCTTCCTGATG CCGCCACTCAACACAATGTT 表 2 苦荞XJ-RILs群体以及亲本RS和GABA的描述统计分析
Table 2. Descriptive statistical analysis of RS and GABA in the XJ-RILs population of Tartary buckwheat and the parents
样本 RS GABA 平均值±标准差/% 范围/% 偏度 峰度 变异系数/% 平均值±标准差/‰ 范围/‰ 偏度 峰度 变异系数/% ‘晋荞2号’ 8.84±0.41 0.50±0.15 ‘小米荞’ 9.26±0.35 0.56±0.04 XJ-RILs 群体 7.53±1.09 4.51~10.52 0.123 0.179 14.43 1.35±0.52 0.37~2.50 0.018 −0.875 38.21 表 3 XJ-RILs 群体各类群RS和GABA性状的方差分析
Table 3. ANOVA analysis of RS and GABA among different groups in XJ-RILs population
类群 株系数/个 RS质量分数/% GABA质量分数/‰ C1 20 8.78±0.72 a 1.99±0.27 a C2 40 6.89±0.62 d 1.83±0.20 b C3 19 6.00±052 e 1.00±0.22 d C4 34 8.27±0.97 b 0.69±0.24 e C5 40 7.71±0.46 c 1.26±0.14 c 说明:不同小写字母表示差异显著(P<0.05)。 表 4 XJ-RILs 群体优异株系的RS和GABA质量分数
Table 4. Contents of RS and GABA in excellent lines of XJ-RILs
株系 RS质量分数/% GABA质量分数/‰ R41 8.20 2.39 R48 8.75 2.50 R49 9.93 2.00 R160 10.52 1.91 R161 9.91 1.73 R183 8.23 2.47 表 5 苦荞XJ-RILs群体RS和GABA的QTL定位
Table 5. QTL mapping of RS and GABA content in the XJ-RILs population of Tartary buckwheat
性状 QTL 染色体 位置/cM LOD R2/% 加性效应 置信区间/cM 标记区间 物理区间/Mb RS qRS3-1 Chr 3 0.39 3.20 7.59 −0.31 38.7~44.0 Block5310~Block5518 12.17~14.26 qRS4-1 Chr 4 1.66 2.82 6.34 −0.28 165.1~168.9 Block9147~Block9171 50.92~51.33 GABA qGABA7-1 Chr 7 1.01 2.52 5.05 0.11 98.9~101.8 Block14135~Block14149 32.352~33.13 说明:LOD. 对数优势比;R2. 表型贡献率。 表 6 候选基因的SNP/Indel变异位点
Table 6. Variant SNP/Indel sites of candidate genes
序号 基因 QTL 染色体 变异类型 变异位点信息
(‘小米荞’/‘晋荞麦2号’)基因注释 1 FtPinG0001690600.01 qRS3-1 Chr 3 SNP A/G 2 FtPinG0008950900.01 qRS3-1 Chr 3 SNP T/A NLP蛋白 3 FtPinG0008951200.01 qRS3-1 Chr 3 SNP T/A SWEET糖转运蛋白 4 FtPinG0008595500.01 qRS3-1 Chr 3 SNP A/T 半乳糖结合蛋白 5 FtPinG0004079200.01 qRS3-1 Chr 3 SNP G/A 6 FtPinG0004083400.01 qRS3-1 Chr 3 SNP A/G 同源框基因 7 FtPinG0001685300.01 qRS3-1 Chr 3 SNP G/A 磷酸核糖基转移酶 8 FtPinG0008594800.01 qRS3-1 Chr 3 Indel G/GT 蛋白激酶 9 FtPinG0008594400.01 qRS3-1 Chr 3 Indel CTTTTTA/C DMP蛋白 10 FtPinG0008127400.01 qRS3-1 Chr 3 Indel C/CA 11 FtPinG0008127200.01 qRS3-1 Chr 3 Indel A/AC 12 FtPinG0001685100.01 qRS3-1 Chr 3 Indel A/AATCAT P型ATP酶 13 FtPinG0001989400.01 qRS4-1 Chr 4 SNP G/A 14 FtPinG0001991700.01 qRS4-1 Chr 4 SNP T/C 核糖体蛋白L7Ae 15 FtPinG0006853900.01 qRS4-1 Chr 4 SNP G/A 硫氧还原蛋白类超家族 16 FtPinG0006854700.01 qRS4-1 Chr 4 Indel T/TG 细胞色素b5 17 FtPinG0009335600.01 qGABA7-1 Chr 7 Indel AT/A 亲环素类肽基脯氨酸顺反异构酶 18 FtPinG0008807100.01 qGABA7-1 Chr 7 Indel TC/T 泛醇-细胞色素c还原酶 19 FtPinG0008810800.01 qGABA7-1 Chr 7 Indel AATAT/A B-box型锌指结构 20 FtPinG0008811900.01 qGABA7-1 Chr 7 Indel G/GT 质体蓝素 21 FtPinG0006715500.01 qGABA7-1 Chr 7 Indel T/TATAATAATAATAATAATA 异戊二烯合成酶 22 FtPinG0005188800.01 qGABA7-1 Chr 7 Indel CTTGTTAAAGAAGTTTTG/C -
[1] ZHOUA Yiming, LU Haoyou, ZHAO Shen, et al. The beneficial effects of Tartary buckwheat (Fagopyrum tataricum Gaertn. ) on diet-induced obesity in mice are related to the modulation of gut microbiota composition[J]. Food Science and Human Wellness, 2023, 12(4): 1323−1330. [2] 吕勇, 许学微, 陈庆富, 等. 苦荞种子活力测定方法与其田间成苗率的相关性[J]. 贵州师范大学学报(自然科学版), 2024, 42(2): 112−117. LÜ Yong, XU Xuewei, CHEN Qingfu, et al. Relativity analysis between the percentage of seedlings in the field and the methods of seed testing of Tartary buckwheat[J]. Journal of Guizhou Normal University (Natural Sciences), 2024, 42(2): 112−117. [3] 陈旭玲, 李显团, 王横, 等. 60Co-γ辐射诱变苦荞品系中淀粉与可溶性糖突变种质的鉴定[J]. 贵州师范大学学报(自然科学版), 2025, 43(2): 85−92. CHEN Xuling, LI Xiantuan, WANG Heng, et al. Identification of mutant germplasm of starch and soluble sugar in Tartary buckwheat strain induced by 60Co-γ radiation.[J]. Journal of Guizhou Normal University (Natural Sciences), 2025, 43(2): 85−92. [4] 吴姗姗, 夏雨, 吴俏槿, 等. 5种苦荞的营养成分分析及比较[J]. 食品工业, 2021, 42(9): 334−337. WU Shanshan, XIA Yu, WU Qiaojin, et al. Analysis of nutritional components of Tartary buckwheat in different varieties and different producing areas[J]. The Food Industry, 2021, 42(9): 334−337. [5] CHAMP M, LANGKILDE A M, BROUNS F, et al. Advances in dietary fibre characterisation. 2. Consumption, chemistry, physiology and measurement of resistant starch; implications for health and food labelling[J]. Nutrition Research Reviews, 2003, 16(2): 143−161. [6] REGINA A, BERBEZY P, KOSAR-HASHEMI B, et al. A genetic strategy generating wheat with very high amylose content[J]. Plant Biotechnology Journal, 2015, 13(9): 1276−1286. [7] 张楚佳, 窦博鑫, 高嫚, 等. 物理法制备RS3型抗性淀粉的研究进展[J]. 食品工业科技, 2023, 44(13): 425−433. ZHANG Chujia, DOU Boxin, GAO Man, et al. Progress in the preparation of RS3 resistant starch by physical methods[J]. Science and Technology of Food Industry, 2023, 44(13): 425−433. [8] 闵芮涵, 孙敏译, 吴昀, 等. 离体百合鳞茎发育及淀粉合成酶基因LohGBSSI的克隆与表达[J]. 浙江农林大学学报, 2020, 37(2): 201−208. MIN Ruihan, SUN Minyi, WU Yun, et al. In vitro bulblet development and analysis of starch synthase gene (LohGBSSI) from Lilium ‘Sorbonne’[J]. Journal of Zhejiang A&F University, 2020, 37(2): 201−208. [9] WANG Lei, LIU Linling, WU Huala, et al. Evolutionary and expression analysis of starch synthase genes from Tartary buckwheat revealed the potential function of FtGBSSII-4 and FtGBSSII-5 in seed amylose biosynthesis[J]. Crop Science, 2023, 63(5): 2925−2940. [10] WANG Xun, FENG Bo, XU Zhibin, et al. Identification and characterization of granule bound starch synthase I (GBSSI) gene of Tartary buckwheat (Fagopyrum tataricum Gaertn. )[J]. Gene, 2014, 534(2): 229−235. [11] HEPSOMALI P, GROEGER J A, NISHIHIRA J, et al. Effects of oral gamma-aminobutyric acid (GABA) administration on stress and sleep in humans: a systematic review[J/OL]. Frontiers in Neuroscience, 2020, 14: 923[2024-09-25]. DOI: 10.3389/fnins.2020.00923. [12] ZHAO Mingxia, TUO Houzhen, WANG Shuhui, et al. The effects of dietary nutrition on sleep and sleep disorders[J/OL]. Mediators of Inflammation, 2020, 2020: 3142874[2024-09-25]. DOI: 10.1155/2020/3142874. [13] RONG Mei, JIA Jiujie, LIN Minqiu, et al. The effect of modified Qiyuan paste on mice with low immunity and sleep deprivation by regulating GABA nerve and immune system[J/OL]. Chinese Medicine, 2024, 19(1): 84[2024-09-25]. DOI: 10.1186/s13020-024-00939-5. [14] 金国明, 陈笑笑, 严国伟, 等. 发芽糙米γ-氨基丁酸富集、调控及其功能食品研究进展[J]. 浙江大学学报(农业与生命科学版), 2024, 50(3): 353−367. JIN Guoming, CHEN Xiaoxiao, YAN Guowei, et al. Recent advances in enrichment, regulation and the functional foods of γ-aminobutyric acid in germinated brown rice[J]. Journal of Zhejiang University (Agriculture and Life Sciences), 2024, 50(3): 353−367. [15] 路静, 白术群, 郑学玲. 萌芽谷物中γ-氨基丁酸代谢及应激积累机制的研究进展[J]. 食品与发酵工业, 2024, 50(17): 380−386. LU Jing, BAI Shuqun, ZHENG Xueling. Research progress on the metabolism of γ-aminobutyric acid and the mechanism of stress accumulation in germinated grains[J]. Food and Fermentation Industries, 2024, 50(17): 380−386. [16] 张钰, 李静媛, 雷琦, 等. 亚精胺和柠檬酸联合处理促进荞麦芽中γ-氨基丁酸的富集[J]. 现代食品科技, 2024, 40(3): 200−208. ZHANG Yu, LI Jingyuan, LEI Qi, et al. Improving the enrichment of γ-aminobutyric acid in buckwheat sprouts by combined treatments using spermidine and citric acid[J]. Modern Food Science & Technology, 2024, 40(3): 200−208. [17] CHEN Guiyun, WANG Yushan, ZHANG Mingyue, et al. Cold atmospheric plasma treatment improves the γ-aminobutyric acid content of buckwheat seeds providing a new anti-hypertensive functional ingredient[J/OL]. Food Chemistry, 2022, 388: 1330643[2024-09-25]. DOI: 10.1016/j.foodchem.2022.133064. [18] RAJ S R G, NADARAJAH K. QTL and candidate genes: techniques and advancement in abiotic stress resistance breeding of major cereals[J/OL]. International Journal of Molecular Sciences, 2023, 24(1): 63[2024-09-25]. DOI: 10.3390/ijms24010006. [19] 吴家胜, 汪旭升. 数量性状位点(QTLs)内候选基因的生物信息学分析方法[J]. 浙江林学院学报, 2008, 25(1): 104−108. WU Jiasheng, WANG Xusheng. Methods of bioinformatic analysis for candidate genes underlying quantitative trait loci(QTLs)[J]. Journal of Zhejiang Forestry College, 2008, 25(1): 104−108. [20] 孙晓雪. 水稻功能性营养品质性状QTL定位及环境互作分析[D]. 哈尔滨: 东北农业大学, 2017. SUN Xiaoxue. Analysis of QTL Mapping and Environmental Interaction of Functional Nutritional Quality Traits in Rice[D]. Harbin: Northeast Agricultural University, 2017. [21] 黎瑞源, 梁龙兵, 石桃雄, 等. 苦荞重组自交系群体F5代SSR遗传图谱的构建[J]. 贵州师范大学学报(自然科学版), 2017, 35(4): 31−45. LI Ruiyuan, LIANG Longbing, SHI Taoxiong, et al. Construction of a microsatellite-based genetic map of Tartary buckwheat using F5 recombinant inbred lines[J]. Journal of Guizhou Normal University (Natural Sciences), 2017, 35(4): 31−45. [22] 张焕新. 抗性淀粉酶法制备及其特性与应用的研究[D]. 无锡: 江南大学, 2012. ZHANG Huanxin. The Preparation and Application of Resistant Starch with a Combination of α-Amylase and Pullulanse[D]. Wuxi: Jiangnan University, 2012. [23] 王传蔚. 多年生荞麦植物菜用品质评价[D]. 贵阳: 贵州师范大学, 2016. WANG Chuanwei. The Vegetable Quality Evaluation of Perennial Buckwheat[D]. Guiyang: Guizhou Normal University, 2016. [24] 薛贤滨, 黎瑞源, 任蓉蓉, 等. 苦荞(Fagopyrum tataricum) RILs群体果壳率相关性状的变异分析和QTL定位[J]. 基因组学与应用生物学, 2024, 43(3): 428−441. XUE Xianbin, LI Ruiyuan, REN Rongrong, et al. Variation analysis and QTL mapping of grain shell rate-related traits of RILs population in Tartary buckwheat (Fagopyrum tataricum)[J]. Genomics and Applied Biology, 2024, 43(3): 428−441. [25] SHI Taoxiong, LI Ruiyuan, ZHENG Ran, et al. Mapping QTLs for 1000-grain weight and genes controlling hull type using SNP marker in Tartary buckwheat (Fagopyrum tataricum)[J/OL]. BMC Genomics, 2021, 22(1): 1423[2024-09-25]. DOI: 10.1186/s12864-021-07449-w. [26] ZHANG Lijun, LI Xiuxiu, MA Bin, et al. The Tartary buckwheat genome provides insights into rutin biosynthesis and abiotic stress tolerance[J]. Molecular Plant, 2017, 10(9): 1224−1237. [27] 石桃雄, 郑俊青, 郑冉, 等. 苦荞重组自交系群体淀粉组分含量和产量的变异分析[J]. 贵州师范大学学报(自然科学版), 2021, 39(5): 1−6, 28. SHI Taoxiong, ZHENG Junqing, ZHENG Ran, et al. Variation analysis of starch components content and yield in recombinant inbred lines of Tartary buckwheat (Fagopyrum tataricum)[J]. Journal of Guizhou Normal University (Natural Sciences), 2021, 39(5): 1−6, 28. [28] 周一鸣, 李保国, 崔琳琳, 等. 荞麦淀粉及其抗性淀粉的颗粒结构[J]. 食品科学, 2013, 34(23): 25−27. ZHOU Yiming, LI Baoguo, CUI Linlin, et al. Granular structure of buckwheat starch and resistant starch[J]. Food Science, 2013, 34(23): 25−27. [29] CHRISTA K, SORAL-ŚMIETANA M. Buckwheat grains and buckwheat products-nutritional and prophylactic value of their components - a review[J]. Czech Journal of Food Sciences, 2008, 26(3): 153−162. [30] 郑发英, 曾姗姗, 刘思曼, 等. 黑苦荞淀粉与普通荞麦淀粉理化和结构性质的比较分析[J]. 食品与发酵工业, 2024, 50(10): 160−168. ZHENG Faying, ZENG Shanshan, LIU Siman, et al. Comparison and analysis of physicochemical and structural properties of black Tartary buckwheat starch and common buckwheat starch[J]. Food and Fermentation Industries, 2024, 50(10): 160−168. [31] 张余, 吴兴慧, 黄凯丰. 不同产地金荞叶菜的产量与品质分析[J]. 中国果菜, 2019, 39(12): 52−55. ZHANG Yu, WU Xinghui, HUANG Kaifeng. Yield and quality of Fagopyrum cymosums from different producing areas[J]. China Fruit & Vegetable, 2019, 39(12): 52−55. [32] 朱丽伟, 周焱, 蔡芳, 等. NIRS法定量分析多年生苦荞叶片蛋白质与GABA含量[J]. 光谱学与光谱分析, 2020, 40(8): 2421−2426. ZHU Liwei, ZHOU Yan, CAI Fang, et al. Quantitative analysis of perennial buckwheat leaves protein and GABA using near infrared spectroscopy[J]. Spectroscopy and Spectral Analysis, 2020, 40(8): 2421−2426. [33] 邹亮, 彭镰心, 许丽佳, 等. UPLC-TOF/MS测定苦荞中的γ-氨基丁酸[J]. 华西药学杂志, 2012, 27(3): 326−328. ZOU Liang, PENG Lianxin, XU Lijia, et al. Determination of γ-aminobutyric acid in Tartary buckwheat by UPLC-TOF/MS[J]. West China Journal of Pharmaceutical Sciences, 2012, 27(3): 326−328. [34] 路子峰, 苏峻冬, 徐麟, 等. 110份鹰嘴豆种质品质性状遗传多样性分析与综合评价[J]. 西北农业学报, 2024, 33(6): 1041−1048. LU Zifeng, SU Jundong, XU Lin, et al. Genetic diversity analysis and comprehensive evaluation of quality traits in 110 Chickpea (Cicer arietinum L. ) germplasm[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 2024, 33(6): 1041−1048. [35] CHEN Liqing, LIN I W, QU Xiaoqing, et al. A cascade of sequentially expressed sucrose transporters in the seed coat and endosperm provides nutrition for the Arabidopsis embryo[J]. The Plant Cell, 2015, 27(3): 607−619. [36] 张计育, 王刚, 王涛, 等. SWEET蛋白在植物生长发育中的功能作用研究进展[J]. 植物资源与环境学报, 2023, 32(5): 1−15. ZHANG Jiyu, WANG Gang, WANG Tao, et al. Research progress on functional roles of SWEET proteins in plant growth and development[J]. Journal of Plant Resources and Environment, 2023, 32(5): 1−15. [37] LI Pei, WANG Lihan, LIU Hongbo, et al. Impaired SWEET-mediated sugar transportation impacts starch metabolism in developing rice seeds[J]. The Crop Journal, 2022, 10(1): 98−108. [38] CUI Tiezhong, CONTE A, FOX J L, et al. Modulation of the respiratory super complexes in yeast: enhanced formation of cytochrome oxidase increases the stability and abundance of respiratory supercomplexes[J]. Journal of Biological Chemistry, 2014, 289(9): 6133−6141. [39] MA Haoli, ZHAO Heming, LIU Zhi, et al. The phytocyanin gene family in rice (Oryza sativa L. ): genome-wide identification, classification and transcriptional analysis[J/OL]. PLoS One, 2011, 6(10): 25184[2024-09-25]. DOI: 10.1371/journal. pone. 0025184. [40] ZHU Nengbo, WANG Ting, GE Liang, et al. γ-amino butyric acid (GABA) synthesis enabled by copper-catalyzed carboamination of alkenes[J]. Organic Letters, 2017, 19(18): 4718−4721. -

-
链接本文:
https://zlxb.zafu.edu.cn/article/doi/10.11833/j.issn.2095-0756.20240570






 下载:
下载: